生物工程、机器学习、图神经网络、监督训练
简介
抗生素的发现是现代医学的基石之一。传统上,抗生素的研发是通过筛选抗菌性的微生物次级代谢物来进行,包括β-内酰胺、氨基糖苷、多粘菌素和糖肽等几大类抗生素都是这样被发现的。然而当前的研发速度已经有所减慢,同时全球性的抗生素耐药问题使得新的有效抗生素的诞生更为困难。从自然化合物中筛选出常常只是重复相同的发现,调整现有抗生素的结构产生的失败率过高,而从化学文库中大范围的筛选则面临成本的化合物多样性的双重限制。近年来,人们提出了将结构分析和筛选相结合的思路,利用机器学习算法从分子性质中预测具有潜在抗菌性能的化合物,为抗生素的发现开辟了新的道路。
2020年2月20日,来自MIT的James Collins和Regina Barzilay在Cell发表题为A Deep Learning Approach to Antibiotic Discovery的文章。他们基于已知化合物训练深度神经网络,然后在Drug Repurposing Hub上预测,发现了一种具有广谱抗菌效应的新型抗生素Halicin。
方法
整体流程
该研究的流程分为5步:
- 针对大肠杆菌选取了2335个已知分子作为训练集,这些化合物对于大肠杆菌的抑制效果均为已知。以80%生长抑制为标准,将这些化合物的抑菌效果二值化。
- 训练二分类模型,使用有方向的消息传递神经网络(directed-message passing neural network,DMPNN),输入为化合物的分子特征,输出则为该化合物可能抑菌的概率。
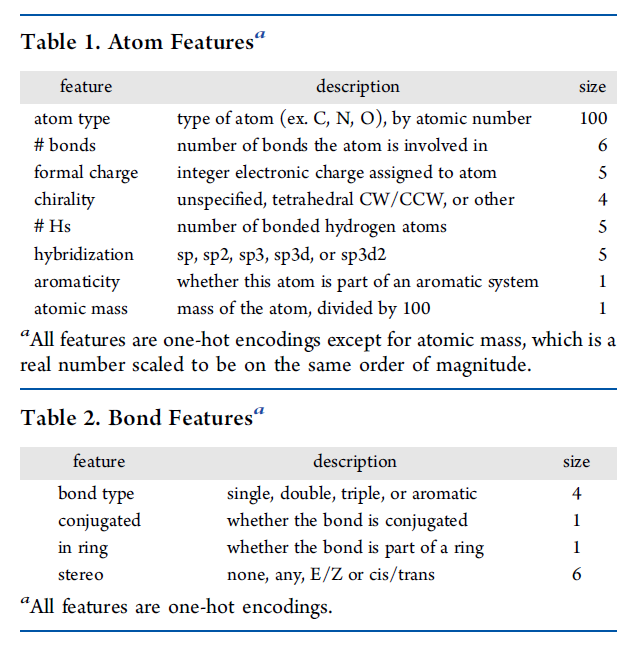
(输入的分子特征) - 集成了20个训练好的网络应用于Drug Repurposing Hub库,包含6111个分子。得到每个分子对于大肠杆菌抑制效果的预测评分(0-1之间的数)。
- 按预测评分排位,剔除一些与训练集结构相似的化合物,进一步通过实验筛选最后得到Halicin。
- 后续该算法还被应用于包含1.07亿分子的ZINC15数据库,又发现了几种与已知抗生素结构差异较大并且效果不错的化合物。
DMPNN算法
DMPNN的算法原理与2017年Neural Message Passing for Quantum Chemistry提出的MPNN算法类似,均属于GNN类别。
MPNN整体分为消息传递阶段与读出阶段:

以原子为结点,以该原子以及其周围结构t时刻的隐藏层/键特征更新其t+1时刻的消息;以该原子t时刻的隐藏层,以及周围结构t+1时刻的消息来更新该原子t+1时刻的隐藏层。
DMPNN:
DMPNN整体结构与MPNN类似,不过变成以键为结点,从而引入方向。

DMPNN好处:
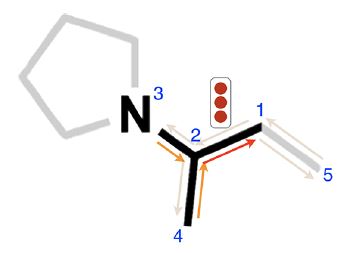
DMPNN的好处在于1-2的消息在下一轮迭代中只会传递给2-3或者2-4,但MPNN的话1传给2在下一轮迭代中又会传回1,带来噪声。
MPNN不足:
由于都是临近结构间传递信息,因此难以捕捉到整体结构对抗菌效果的影响。为此,该研究在读出阶段添加了200种已知的整体结构的信息。
参考文献
A Deep Learning Approach to Antibiotic Discovery
Analyzing Learned Molecular Representations for Property Prediction

