三维基因组
https://github.com/mdozmorov/HiC_tools
基因组并不是简单的线性序列,而是具有三维空间结构的,而且这种3D结构可以对DNA复制、基因转录调控、染色质浓缩和分离等基本生物学过程产生重要的影响。
三维基因组学以研究 真核生物核内基因组空间构象 及 其对不同基因转录调控的生物学效应 为主要研究内容,是后基因组学时代的一个重要研究领域和新兴学科方向。
技术发展
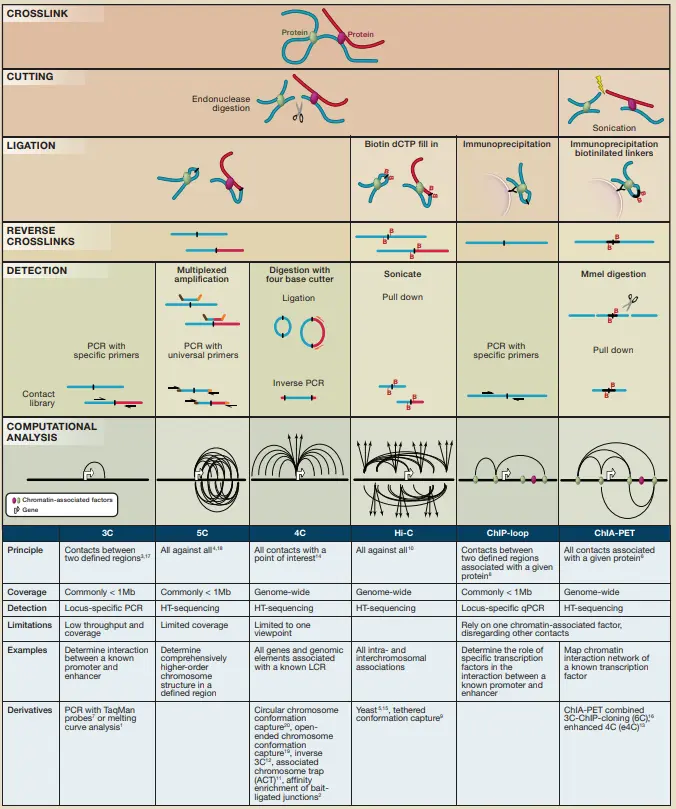
- 3C: 1 versus 1 (一对一)
chromosome conformation capture - 4C: 1 verus Many/All (一对多/全)
chromosome conformation capture-on-chip - 5C: Many versus Many (多对多)
chromosome conformation capture carbon copy - Capture-C: Many versus All (多对全)
chromosome conformation capture coupled with oligonucleotide capture technology - Hi-C: All versus All (全对全)
genome-wide chromosome conformation capture - ChIA-PET: 通过免疫沉淀法设计抗体来靶向特定蛋白进行三维结构捕获
- HiChIP
- ChiC: 设计引物或者探针来杂交富集靶向核酸的技术
- Pore-C、SPRITE、Tri-C、COLA ……
基于通过Hi-C技术获得的数十亿互作位点对而构建的全基因组互作图谱,能够非偏倚地鉴定出活性/惰性染色质间隔、拓扑关联域、环状结构域及增强子-启动子相互作用。但这些结构在基因调控中的作用还未能揭示。像其他细胞机制一样,染色质的功能性3D状态可能无法通过简单的成对相互作用进行完整描述。
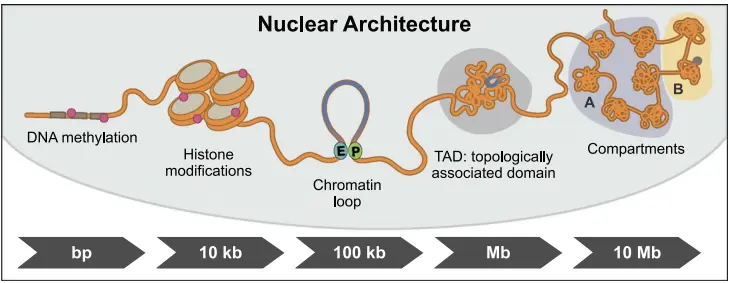
分辨率与三维结构
通过 Hi-C 技术可以确定全基因组范围内整个染色质在空间位置上的关系、较大尺度下染色质之间的互作关系,包括染色体之间的互作关系。
每个层次的分辨率可以鉴定出不同类别的功能元件,比如最小的分辨率,对应的是基因级别甚至 CTCF 等更小元件的互作观察,而达到 10kb 级别的分辨率时,一般可以鉴定出 TAD 之间比较精细的互作关系,再大一些,则是到一个相对宏观层次来判断 TAD 之间的关系;在往上到全基因组层次,可以清楚地观察搞染色体之间的互作关系。
不同分辨率所需的测序数据量也不同,一般分辨率越高的 3D 基因组互作图谱所需要的有效互作 Hi-C 数据量也就越大。
- 染色体疆域(chromosome territories)
- 活性/惰性区室(A/B compartment)
- 拓扑关联域(TAD,topologically associating domain)
- 染色质环(chromatin loop)
应用领域
基因组的三维结构在不同类型的细胞和细胞不同分化发育阶段是存在差异的。利用基因组三维结构可以在基因组组装、基因组三维构象重构、基因组三维构象调控元件等领域进行应用。
Hi-C
在众多的三维基因组技术中,从实用性、流通性、通量、有效率等方面考虑,Hi-C 技术被认为是研究全基因组范围内最主要手段。
在 Hi-C 实验的数据分析中,数据在经过序列比对、过滤及数据合并的预处理流程后,会生成一个对称的由一个个相同大小的基因组区间构成的染色质相互作用矩阵。矩阵中的每个区间都反映了相应的一对基因组区间之间的相互作用频率,该区间的大小被称为分辨率。
在 Hi-C 数据分析流程中,文库长度、GC 含量、序列比对效率等差异都会带来下游数据分析的系统误差,而 Hi-C 数据标准化软件则试图消除不必要的系统偏差,以便尽可能保留真实的互作频率。目前有许多进行 Hi-C 数据标准化的方法。这些方法根据模型假设,可以粗略分为显示和隐式方法。
显示方法假设系统偏差,如片段长度、GC 含量和序列比对效率是已知的,并且在统计模型中得到了解释。
而隐式方法则假设系统偏差的累积效应被捕获在每个 bin 的互作频率内,然后通过不同的算法将每个 bin 内的互作频率根据特征进行分解,典型的工具有 SCN、ICE、KR 及 chromoR 等。
HiCcompare 和 multiHiCcompare 是能够跨样本进行 Hi-C 数据标准化的软件:HiCcompare 可以进行两个样本同时标准化;而 multiHiCcompare 则可以对超过两个样本进行 Hi-C 数据标准化,同时 multiHiCcompare 考虑了 IF(interaction frequency,互作频率)对距离的衰减模式,这使 multiHiCcompare 在大多数情况下都能获得明显更好的性能(但需要更大的内存以同时加载多个样本的所有矩阵)。
三维记忆哦你组的质控和一般 NGS 数据或者三代测序数据的质控不一样:一般测序数据是为了去除低质量的 reads 或者碱基,而三维基因组的质控或者数据标准化是为了获得更精确、真实的位点之间的互作频率,因此会将文库长度、GC 含量、序列比对效率等差异考虑到数据标准化算法内,这是三维数据所特有的。换言之,这个标准化过程就像我们在做序列组装或者比对时,会预先将序列构建图或者比对到参考基因组一样,进行预处理,将质控后的序列转换为对下游分析有用的特征信息,例如,两种主流的限制性内切酶 Dpn II 和 Hind III,去除低覆盖区 bin 后的平均片段长度分别为 570bp 和 4500 bp,选用不同酶的标准化结果也差别很大。
- Hi-C实验流程:
- 使用细胞交联剂多聚甲醛处理细胞,固定DNA构象;
- 细胞裂解后,利用限制性内切酶处理教练的DNA,产生粘性末端;
- 末端补平修复,并同时引入生物素,标记寡核苷酸末端;
- 使用DNA连接酶连接临近的DNA片段;
- 蛋白酶消化解除与DNA的交联状态,纯化DNA并随机打断至300-500bp的片段;
- 亲和素磁珠捕获标记的DNA,进行二代建库测序。
Micro-C
使用微球菌 MAase 代替限制性内切酶,进一步显著提高了互作图谱的分辨率。Micro-C 可能会损失一些远距离的互作,于是使用双交联剂对细胞进行交联,从源头上交联到更多的互作。
在这些技术不断升级的过程中,研究人员注意到了两个影响 Hi-C 结果的重要因素,一是交联剂,二是片段话 DNA 的酶。
不同的交联剂由于分子臂的长度不同,其可以交联到的空间距离不同,最终就会影响互作的结果。比如最常用的交联剂甲醛的可交联半径为2-Å,空间距离稍远的 DNA 和蛋白就无法被交联上。而另外两种分子臂更长的交联剂则可以实现更远空间距离的交联,即 disuccinimidyl glutarate 和 ethylene glycol bis。因此,若使用双交联剂,则预期可以获得更多的空间相互作用。
另外一个影响因素则是片段话 DNA 的酶,不同的酶会将 DNA 切成不同大小范围的片段,进而影响到互作分辨率,如酶切后片段大小:六碱基单酶 > 四碱基单酶 > 四碱基双酶 > MNase。

检测 compartment 的能力:双交联 > 单交联;长片段 > 中片段 > 短片段
检测 loops 的能力:双交联 > 单交联;短片段 > 长片段
Hi-C 3.0: FA+DSG-Ddel+DpnII
虽然 MNAse 的方法检测到了最多的短距离互作,但是其检测长距离互作的能力却最差,原因是其将片段切得太碎,而 Hi-C 3.0 避免了长距离互作丢失的问题,兼顾到了捕获短距离和长距离互作的能力。
Micro-C 的主要目的是深入分析单基因之间的互作即 loops。
参考
公众号 | 生信菜鸟团 | 一文读懂三维基因组
公众号 | 30+张图片!带你走进三维基因组(附一篇Cell经典案例精读)
bilibili | 达澈小讲堂-10-讲解Hi-C的测序原理和应用
文献 | Nature “The 3D genome” 丛刊 https://www.nature.com/collections/rsxlmsyslk
文献 | Science | 2009 | Comprehensive Mapping of Long-Range Interactions Reveals Folding Principles of the Human Genome
文献 | Cell | 2014 | A 3D Map of the Human Genome at Kilobase Resolution Reveals Principles of Chromatin Looping
文献 | Science | 2018 | Chromatin plasticity: A versatile landscape that underlies cell fate and identity
Disease-Associated Short Tandem Repeats Colocalize with Chromatin Domain Boundaries
文献 | Trends in Plant Science | 2018 | Entering the Next Dimension: Plant Genomes in 3D
文献 | New Phy | 2021 | Plant 3D genomics: the exploration and application of chromatin organization
文献 | Plant and cell physiology | Plant 3D Chromatin Organization: Important Insights from Chromosome Conformation Capture Analyses of the Last 10 Years

