对于Illumina测序,我一直有一个大概的认知,就是:
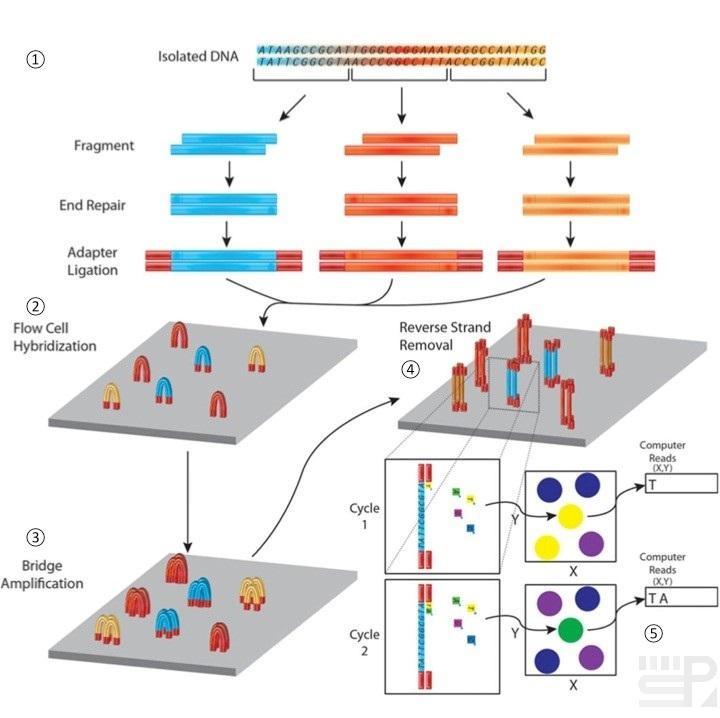
- 打碎DNA,加接头
- DNA片段结合到flow cell上,通过桥式扩增成簇
- 边合成边测序,识别不同簇在每一轮dNTP结合过程中发出的荧光
- 将荧光信号转化为序列信息
2006 年 Solexa 公司推出第二代测序系统 GA,其中 DNA 簇、桥式 PCR()和可逆阻断()等核心技术使 GA 系统具有高通量、低成本的显著优势。
Illumina 在创立之初仅是一家主要销售微阵列芯片的公司,2006 年底通过收购 Solex 公司及其测序技术,Illumina 开始大规模进军测序市场,并在数年内发布了多种测序仪,包括:
- 低通量的桌面测序仪:MiSeq、MiniSeq 及 ISeq 系列
- 高通量的台式测序仪:Sea 系列、HiSeq 系列及 NextSeq 系列
基于高通量、低成本、高准确度等优势,Illumina 渐渐成为测序市场的主流公司,基本覆盖行业内所有测序应用。
世界上采用最广泛的的第二代测序平台是 Illumina 公司的 HiSeq 系列及 NovaSeq 系列。HiSeq 2000 能够在单次运行中产生 600Gb 数据,每天最高产生 55Gb;HiSeq 1500/2500 系统是在 HiSeq 2000 系统的基础上开发的。
2014 年,Illumina 公司又发布了 HiSeq$ \times $Ten 系统,这是针对大规模人群全基因组测序的系统。
2017 年,Illumina 公司推出了 NovaSeq 系列测序系统,单次运行最多能产生 6Tb 的数据,是目前最高通量的测序仪之一。
Illumina 所有测序平台的核心原理均采用边合成边测序(SBS)的方法。
介绍
文库制备
利用超声波把待测的DNA片段打断成200~500bp长的小片段,并在这些小片段的两端连上特异性接头(adaptors),构建双链DNA文库。
破碎
问题:什么情况如何选择酶法打断和超声波打断?
1.根据样本的情况:比如起始量低(低于50ng以下) 这种情况不宜用机械打断,机械打断对样本损伤比较大,损失更多样本导致建库失败。更适合用酶切的方法;
2.实验的目的和需求:比如甲基化测序、Chip-Seq 测序更适合用机械打断。
| iSeq 100 | MiniSeq | MiSeq | NextSeq 550 | NextSeq 1000/1000-CN NextSeq 2000/2000-CN |
NovaSeq 6000 | NovaSeq X | |
|---|---|---|---|---|---|---|---|
 |
 |
 |
 |
 |
 |
 |
|
| 每个流动槽最大通量 | 1.2Gb | 7.5Gb | 15Gb | 120Gb | 540Gb | 3Tb | 8Tb |
| 运行时间(范围) | 约 9.5-19h | 约 2-24h | 约 5-56h | 约 11-29h | 约 8-44h | 约 13-44h | 约 17-48h |
| 每次运行的最大 reads 数(单端 reads) | 4M | 25M | 25M | 400M | 1.8B | 10B(单流动槽) 20B(双流动槽) |
26B(单流动槽) 52B(双流动槽) |
| 最大读长 | 2×150bp | 2×150bp | 2×300bp | 2×150bp | 2×300bp | 2×250bp | 2×150bp |
| 大型全基因组测序(人类、动物、植物) | - [ ] | - [ ] | - [ ] | - [ ] | - [ ] | - [x] | - [x] |
| 小型全基因组测序(微生物、病毒) | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] |
| 外显子组和大型 panel 测序(基于富集) | - [ ] | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] |
| 靶向基因测序(基于扩增子、基因 panel) | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] |
| 单细胞分析(scRNA-seq、scDNA-seq、寡核苷酸标记检测) | - [ ] | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] |
| 转录组测序(总 RNA-seq、mRNA-seq、基因表达图谱分析) | - [ ] | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] |
| miRNA 和小 RNA 分析 | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] |
| 染色质分析(ATAC-seq、ChIP-seq) | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] | - [x] |
| 甲基化测序 | - [ ] | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] |
| 16S宏基因组测序 | - [ ] | - [x] | - [x] | - [x] | - [x] | - [x] | - [x] |
| 宏基因组学分析(鸟枪法宏基因组学、宏转录组学) | - [ ] | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] |
| 游离核酸测序和液体活检分析 | - [ ] | - [ ] | - [ ] | - [x] | - [x] | - [x] | - [x] |
https://emea.illumina.com/systems/sequencing-platforms.html
PCR-free
单端测序or双端测序
而文库制备的目的是在待测序的DNA片段两端加上能够与测序仪匹配的接头序列。
构建好的文库通常由P5、Index2、Rd1 SP、DNA Insert、Rd2 SP、Index1、P7这7个部分构成(双端Index结构,如果是单端Index,无Index2)。其中,P5和P7分别与Flowcell上的接头引物序列互补和相同,进行簇生成。Index又称Barcode,是我们进行文库混合测序时用于数据拆分的一个标签,其中与P5端连接的Index称为Index 2,又称之为i5;与P7端连接的称为Index1,也称为i7。
RNA-seq PolyA富集
链特异性or非链特异性
https://hbctraining.github.io/Intro-to-rnaseq-hpc-salmon/lessons/Intro-to-RNAseq.html
流动槽(flow cell)杂交
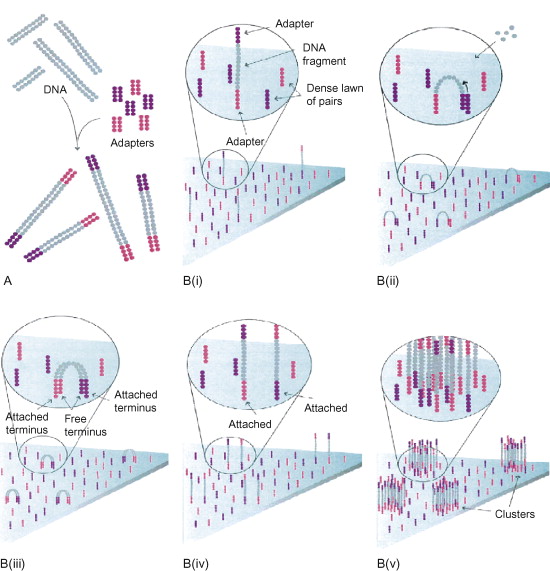
流动槽的表面结合着一层oligo接头,是用于吸附流动DNA片段的槽道。当DNA文库建好后,这些文库中的DNA在通过流动槽的时候会杂交到流动槽表面的oligo引物上。DNA片段杂交到流动槽表面后,oligo引物会在聚合酶的作用下延伸。

桥式PCR扩增与变性
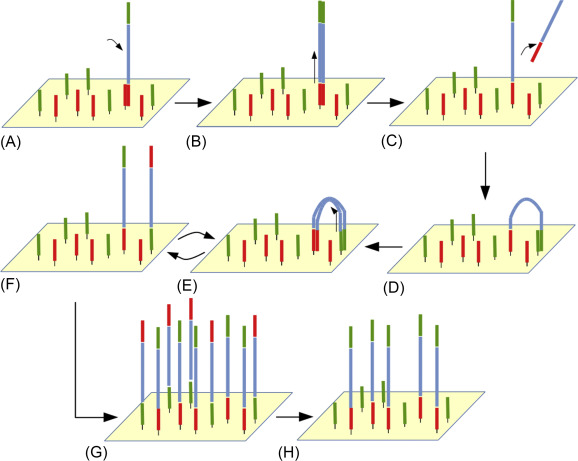
合成DNA双链之后,双链分子变性分开,其中的模板链被洗掉,新合成的单链以共价键的形式紧紧连接在流动槽表面。此外,新合成单链弯曲杂交在相邻oligo引物上形成一个桥式结构。杂交之后,引物在聚合酶的作用下延伸,形成双桥的桥式结构。双链的桥式结构变性打开,形成2个以共价键结合在流动槽表面的单链模板。单链再弯曲杂交在相邻的oligo引物上形成桥式结构,杂交之后再延伸。由此,桥式扩增一直循环重复,直至形成5000~10000个拷贝。拷贝足够多之后,双链DNA桥变性分开,DNA反链被剪接后洗掉,仅留下由正链组成的簇,并且游离的3’端被封闭,防止不必要的DNA延伸。最后,测序引物被杂交到接头序列上,便于之后的测序。
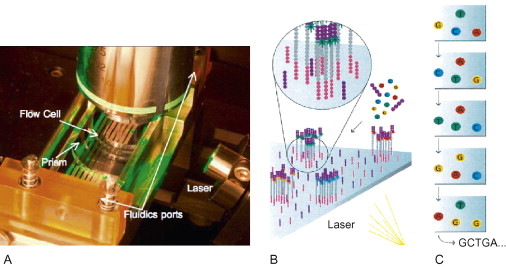
测序

测序采用的是边合成边测序的方法。向反应体系中同时添加DNA聚合酶、接头引物和带有碱基特异荧光标记的4种dNTP。这些dNTP的3’端羟基被化学方法所保护,因此每次只能添加一个dNTP。在dNTP被添加到合成链上后,所有未使用的游离dNTP和DNA聚合酶会被洗脱掉。接着,再加入激发荧光所需的缓冲液,用激光激发荧光信号,并有光学设备完成荧光信号的记录,最后利用计算机分析将光学信号转化为测序碱基。这样的荧光信号记录完成后,再加入化学试剂猝灭荧光信号并去除dNTP 3’端羟基保护基团,以便能进行下一轮的测序反应。Illumina测序技术每次只添加一个dNTP的特点,能够很好地解决同聚物长度的准确测量问题,它的主要测序错误来源是碱基的替换。
在延伸了一段时间后,聚合酶的活力就会大大降低,错误率也会越来越高。当错误率高到不可容忍的时候,就必须停止测序反应。
所以就有了陆续推出的单端SE50、SE100、SE150、SE250以及双端的PE50、PE100、PE150、PE300,也就意味着,测序仪可以控制读取长度的,仪器会顺着DNA一个碱基一个碱基的读取,在机器开始测序前,可以设置读取的循环数,例如100Cycle.那么NGS测序仪就会读取100个碱基,即SE100。
但是目前来讲,由于自身方法的局限性(包括酶的失活,阻断基团切割的效率等等都会导致测序质量随着测序长度不可逆地降低)仍然难以突破。这也就是二代达不到一代测序的长度,更不可能到三代的长度。

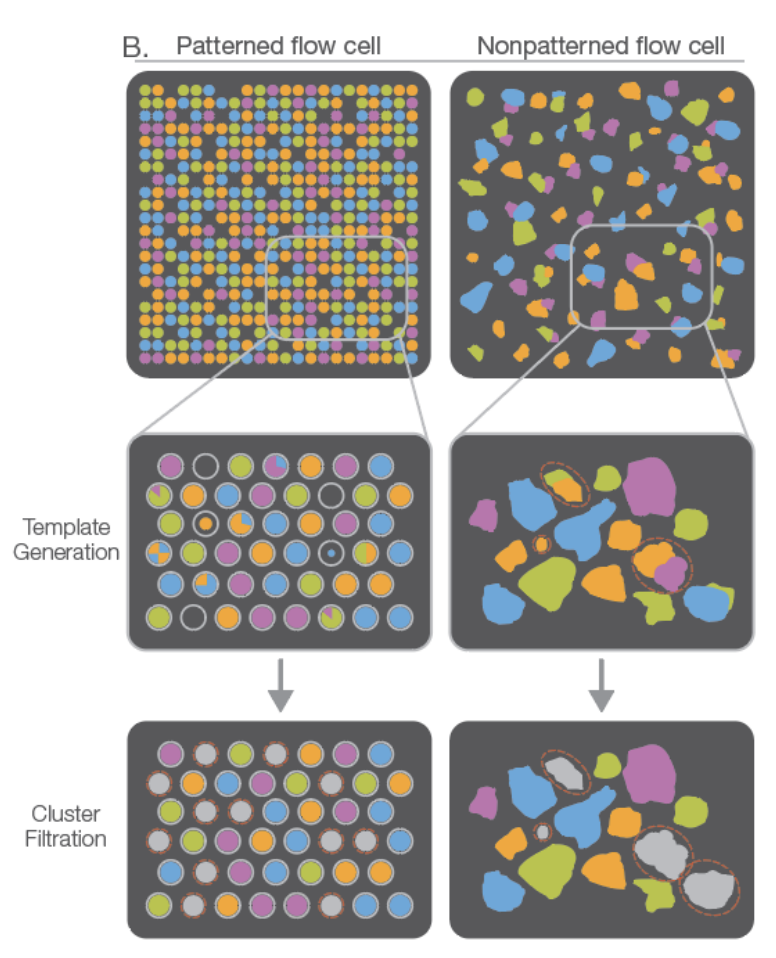
Patterned Flow Cell
参考
https://www.youtube.com/watch?v=fCd6B5HRaZ8
https://www.youtube.com/@IlluminaInc


