写在前面
命名、分类不断变化

转座子分类系统
目前,常用的转座子分类系统是 Wicker 等人提出的三类层级分类系统:
- 2 大类(Class)
- 9 目(Order)
- 29 个超家族(Superfamily)
转座子的最高层级分类主要根据转座过程中是否存在中间体 RNA,分为
目 (Order) 主要根据整合机制划分,如TIR 和Ctypton,其整合中间体分别是双链DNA (Double stranded DNA, dsDNA) 和环状双链DNA (Circular dsDNA, CirdsDNA)。除了上述亚类1和亚类2提到的4类目,
目继续分类成超家族 (Superfamily),主要考虑编码蛋白质的相似性和排列顺序、靶位点重复序列 (Target site duplication, TSD)。
超家族之后还可以分类,继续深层次的分类则主要依据转座子序列的特征及其特异性的差异划分。这种统一的分类方式很好地改进了先前Finnegan (Finnegan, 1989)提出的两类分类方法,对来自不同物种的转座子的比较和进化研究具有重要的意义。
分类系统概述
分类系统概述
1989年,Finnegan提出了第一个转座元件(TEs)的分类系统,按转座中介物的类型区分为两大类:
RNA(I类或逆转座子)和DNA(II类或DNA转座子)。
I类的转座机制通常被称为“复制粘贴”,而II类则称为“剪切粘贴”。随后,科学家发现了不依赖RNA中介物而复制粘贴的细菌和真核生物TEs,以及一种高度简化的非自主TEs,称为微型反向重复转座元件(MITEs),这些发现对两类系统提出了挑战。这些发现导致了新的分类方案的产生,或是引入了第三类,或是放弃了两类系统,转而采用基于酶学特征的分类。
我们提出的分层TE分类系统(图1)调和了这两种方法,保留了两大类的划分,同时应用了机械和酶学标准。该系统包括(按层级顺序):类、亚类、目、超家族、家族和亚家族。这些术语是为了与生物系统发生学中的术语保持一致,并承认某些术语(如超家族)已被广泛用于TE群体。最高层级(类)仍然按照是否有RNA转座中介物来区分TEs。
亚类此前用于区分I类TEs中的长末端重复(LTR)与非LTR(长散布核元件(LINE)和短散布核元件(SINE)),在此用于区分那些通过复制自己进行插入的元件与那些离开供体位点再整合到其他位置的元件。它同时反映了在TE供体位点上被剪切的DNA链的数量。一个新的分类级别——目,用于标记插入机制的主要差异,因此也反映了总体的组织和酶学特征,取代了I类TEs的亚类分类。
目内的超家族具有相同的复制策略,但通过统一且广泛的宏观特征(如蛋白质或非编码结构域的结构)进行区分。此外,超家族还在靶位点重复序列(TSD)的存在与大小上有所差异。TSD是一种短的直接重复序列,在TE插入时在其两侧生成。在超家族之间,几乎没有DNA序列的保守性,而蛋白质水平的相似性也很有限。
超家族进一步划分为家族,家族是通过DNA序列保守性定义的。同一超家族中的不同家族在蛋白质水平上通常具有较高的相似性;然而,DNA序列的保守性较小,且主要限于编码区内高度保守的部分。尽管TEs可以分为相对较少的目和超家族,一个大型基因组中仍可能包含数百或数千个不同的TE家族。亚家族的划分基于系统发育数据,在特定情况下,可能用于区分内部均一的自主和非自主群体。
最低的分类级别是插入,指的是一个特定的个体拷贝,代表一次特定的转座和插入事件,这对于基因组注释尤为重要。在插入之上的所有分类级别中,一个元件可以暂时被标为“未知”,作为其分类。例如,一个新的TE可能由于其具有典型的LTR结构被识别为LTR逆转座子,但由于缺乏编码序列,可能无法将其分类为某一超家族(例如Gypsy或Copia)。通过这种方式,复杂的分类任务留给专家,而不要求注释者进行。为了保持风格一致性,我们建议目以下的所有分类级别名称应以斜体书写。
I 类 RNA 转座子
(Class I,Retrotransposons)
Class I 类转座子主要通过RNA 介导的复制-粘贴 (Copy and paste) 过程迅速增殖。ClassⅠ类转座子也可分为 5 目,主要依据ClassⅠ类转座子的机制特征、组织结构和逆转录酶进行区分:
所有I类转座子(TEs)通过RNA中间体进行转座。这个类别不需要细分亚类——没有任何成员在供体位点切割或转移DNA链。相反,RNA中间体是由基因组中的一个拷贝转录而来,然后由TE编码的逆转录酶(RT)将其逆转录为DNA。每次完整的复制周期都会产生一个新的拷贝。因此,逆转座子往往是大型基因组中重复部分的主要贡献者。
逆转座子可以根据其机械特征、组织结构和逆转录酶系统发育分为五个目:
LTR 目

完整的 LTR(Long terminal repeat,长末端重复)具有显著的结构特征,在其两端是高度相似或相同的 LTR 序列,LTR 序列的长度从几十到上千碱基对 (bp) 不等 (图1-2)。LTR 序列具有典型的结构特征:
Copia
根据多个蛋白结构域的系统发育分析,超家族进一步被分为亚类。通常:
- Copia 逆转座子分为:Ivana (Sirevirus/Oryco)、Osser (hemivirus)、Bianca 和SIRE
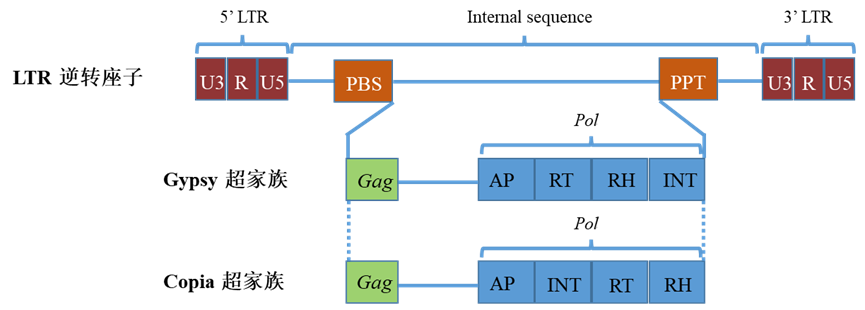
Gypsy
- Gypsy 逆转座子分为:
- 染色病毒 (Chromovirus) ,包含CRM、Galadriel、Reina、Tcn1 和 Tekay (Del/Del1)
- 非染色病毒 (Non-chromovirus),包含 Athila,Tat,Phygy 和 Selgy
LTR 逆转座子在动物中较为稀少,但在植物中占主导地位。它们的长度范围从几百个碱基对到最高可达 25 kb(例如Ogre)。LTRs 位于这些元件的两侧,长度从几百个碱基对到超过 5 kb 不等,并且以 5′-TG-3′ 开始,以 5′-CA-3′ 结束。
整合时,LTR逆转座子产生 4–6 bp 的靶位点重复序列(TSD)。
它们通常包含编码 GAG 的开放阅读框(ORF),GAG 是病毒样颗粒的结构蛋白,以及编码 POL 的 ORF。Pol 编码天冬氨酸蛋白酶(AP)、逆转录酶、RNase H(RH)和 DDE 整合酶(INT)。偶尔还会有一个未知功能的额外 ORF。LTR 逆转座子还包含用于包装、二聚化、逆转录和整合的特定信号。
非后生动物中的两个主要超家族 Gypsy 和 Copia 在 POL 中 RT 和 INT 的顺序不同。所有 LTR 目中的超家族使用类似的转座机制。
许多LTR逆转座子家族并不限于任何一个物种,至少在禾本科植物中是这样。那些基因组较大的植物,如玉米、小麦或大麦,可以包含成千上万个家族。然而,尽管它们多样性巨大,相对较少的家族构成了这些大型基因组中的重复部分主体。例如,小麦中的 Angela、大麦中的 BARE1、玉米中的 Opie 以及高粱中的 Retrosor6。人类中也存在大量的 LTR 逆转座元件家族,但大多数已不再活跃,并且数量适中。
Bel-Pao
BEL–Pao 元件在结构上与 Gypsy 或 Copia 元件相似。它们含有标准的 LTR,编码 GAG 和 POL 蛋白,并在插入时产生 4–6 bp 的TSD。然而,基于 RT 的系统发育,它们形成了一个独立的分支。迄今为止,它们仅在后生动物中被检测到。
Retrovirus
从进化上看,Retrovirus(逆转录病毒)和 LTR 逆转座子关系密切。逆转录病毒可能是由 Gypsy LTR 逆转座子进化而来,通过获得包膜蛋白(ENV)及一组额外的蛋白质和调控序列,进化成病毒生活方式。逆转录病毒主要限于脊椎动物,尽管某些果蝇 Gypsy 家族成员能够感染新的个体。因此,逆转录病毒可以被纳入我们的分类系统,尽管它们长期以来被归类为病毒,并且在关键方面与逆转座元件不同。例如,它们的进化受流行病学的影响,而不是生物系统发育。
ERV
通过使能够实现细胞外移动性的结构域失活或删除,逆转录病毒也可以转变为 LTR 逆转座子。它们不再具有感染性,依赖于生殖系的垂直传播进行繁殖。因此,我们将这些所谓的内源性逆转录病毒(ERVs)归为 LTR 亚类中的一个超家族。尽管它们的 pol ORF 与 Gypsy TEs 类似,但其 gag ORF 编码了胶体和基质功能(Gypsy gag 仅编码基质)。此外,它们仍然具有一个 ENV,包含表面和跨膜单位,这是逆转录病毒的特征。
DIRS 目

DIRS(Dictyostelium intermediate repeat sequence,双歧杆菌中间重复序列)目成员含有一个酪氨酸重组酶基因而不是 INT,因此不会形成 TSD。它们的末端结构也不寻常,类似于分裂的直接重复序列(SDR)或倒置重复序列。这些特征表明它们的整合机制与LTR和LINE TEs不同。然而,它们的RT将它们归为I类。该目成员已在各种物种中被发现,从绿藻到动物和真菌。
PLE 目

PLE(Penelope-like elements,Penelope 类元件)目最早在果蝇(Drosophila virilis)中被发现。尽管它们已在超过 50 个物种中被检测到,包括单细胞动物、真菌和植物,但在其他已测序的基因组中却没有发现,表明其分布不均。PLE 编码的 RT 与端粒酶更为相似,而不是 LTR 逆转座子或 LINE 的 RT,其内切酶则与内含子编码的内切酶以及细菌 DNA 修复蛋白 UvrC 相关。某些成员包含一个功能性内含子。成员还具有 LTR 样序列,可以是正向或反向排列。
LINE 目

LINEs(Long interspersed nuclear elements,长散在核元件)没有LTR,可以达到数千碱基的长度,并且存在于所有真核生物的类群中。它们被分为五个主要超家族:R2、RTE、L1、Jockey 和 I。每个超家族又进一步细分为多个家族。历史上,LINE 超家族曾被划分为17个“谱系”,然后再分为家族。尽管谱系分类不包括在我们的系统中,但它在 LINE 研究者中广泛使用。
自主的 LINEs 至少在它们的 pol ORF 中编码了一个 RT 和一个核酸酶以完成转座;RTE 超家族的成员只有这个 ORF,类似于原始的 LINEs。核酸酶可以是一个内切酶(对于 R2 超家族,位于 RT 的 C 末端)或一个无嘌呤或无嘧啶内切酶(对于 L1、RTE、I 和 Jockey 超家族,位于 RT 的 N 末端)。有时在 pol 的 5′端可以找到一个类似 gag 的 ORF,但其作用尚不明确。只有 I 超家族的成员包含 RNaseH。尽管 LINEs 通常在插入时形成 TSD,由于 5′端的截断,它们往往难以发现。截断可能是由于逆转录的提前终止。在其 3′端,它们可以显示出 poly(A) 尾、串联重复或仅为 A 富集区域。
LINEs 在真核生物中的丰度和多样性不同,但在许多动物中,它们比 LTR 逆转座子更为主导。L1 家族在人类中约有 105 个拷贝,占人类基因组的 20%。相比之下,在疟蚊(Anopheles gambiae)中,大约 100 个分化的家族仅占基因组的 3%。在植物中,LINEs(例如玉米中的 Cin4 和拟南芥中的 Ta11)相比于 LTR 逆转座子似乎很少见,尽管也有显著的例外(例如百合属中的 Del2)。大多数已知的植物 LINEs 来自 L1 和 RTE 超家族。然而,鉴于植物中缺乏系统研究,可能存在更多的多样性。
SINE 目

SINE(Short interspersed nuclear elements,短散在核元件)目起源独特。虽然它们是非自主的(见下文),但它们不是自主 I 类元件的缺失衍生物;相反,它们来源于偶然逆转座的各种 RNA 聚合酶 III(Pol III)转录物。与逆转录的假基因不同,它们具有内部 Pol III 启动子,允许它们被表达。它们依赖 LINEs 提供转座所需的反式作用因子,如 RT。有些“严格”的 SINEs 有一个独特且必需的伴侣,而另一些则是广泛适应者。
SINEs 较小(80–500 bp),并产生 TSD(5–15 bp)。含有 Pol III 启动子的“头部”定义了 SINE 超家族,并揭示了它们的起源:tRNA、7SL RNA 和 5S RNA。SINE 的内部区域(50–200 bp)是家族特异性的,且来源多样,有时来自 SINE 的二聚化或三聚化。其 3′端的起源,虽然有时可能是 LINE,但通常不明确。它可以是 A 或 AT 尾,类似于 LINE 元件。最知名的 SINE 是 Alu,在人类基因组中最少有 500,000 个拷贝。
Alu 属于 7SL。
II 类 DNA 转座子
ClassⅡ类转座子(DNA transposons)则使用“Cut and paste”(剪切-粘贴)机制来增加拷贝数。
与 I 类一样,II 类转座子同样古老,存在于几乎所有的真核生物中。通常它们的数量较少到中等,但一些如真菌中的 Pogo–Fot1 或小麦及其近缘物种中的 CACTA 已成功地殖民了这些基因组。II 类元件也在原核生物中以简单的形式存在,称为插入序列(IS),或作为更复杂结构的一部分被发现。
II 类元件包含两个亚类,区别在于转座过程中切割的 DNA 链数量,但它们都不通过 RNA 中间体移动。
Class II 类转座子分为两个亚类,它们的区别在于转座过程中供体位点是否保留原先位置。
亚类 1
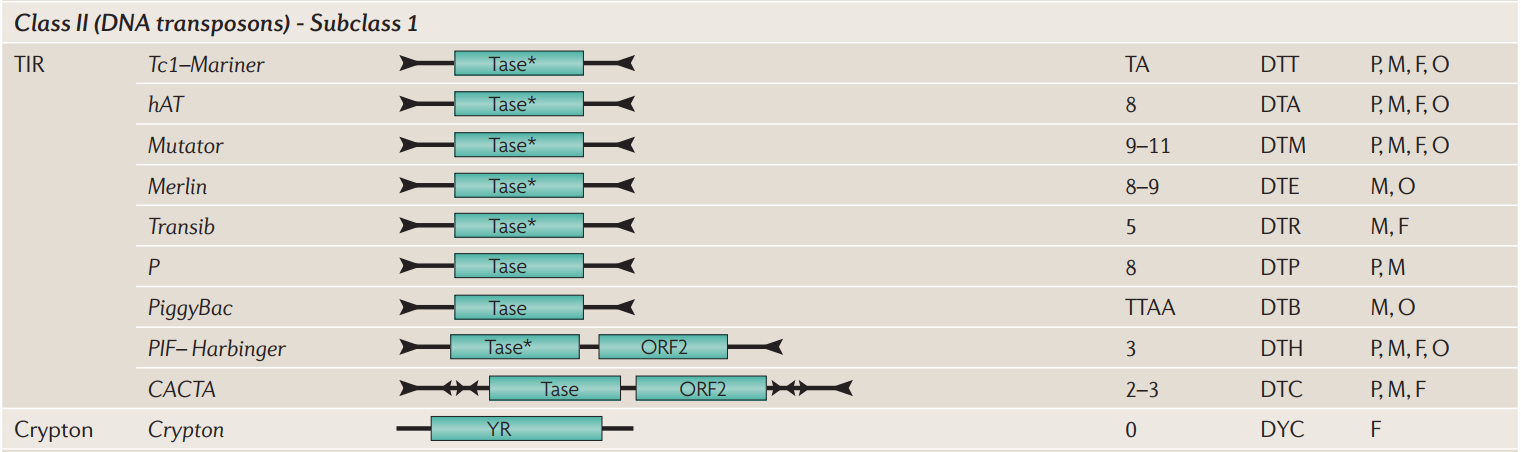
亚类 1 的转座过程不保留原始拷贝,如末端反向重复转座子(Terminal inverted repeats, TIR)和 Ctypton。
TIR 目
亚类 1 包括经典的“切割-粘贴”型转座子,它们属于 TIR 目,其特点是末端倒位重复序列(TIRs),长度可变。已知的九个超家族根据TIR序列和靶位点重复序列(TSD)的大小进行区分。这些 TEs 可以通过在染色体复制过程中从已经复制的位置转移到复制叉尚未经过的位置来增加其数量。或者,它们可以通过切割后修复缺口来在供体位点创建一个额外的拷贝。转座是由一种识别 TIR 并在每个末端切割双链的转座酶介导的。以前的分类基于转座酶中 DDE 催化核心的存在;然而,我们不再使用这一标准,因为它目前尚在变化中:在以前被认为不含DDE的群体中发现了 DDE 和类似 DDE 的基序。对于剩余的非 DDE 超家族(如P、piggyBac 和 CACTA),其催化结构域尚未得到很好的确定。它们可能含有催化天冬氨酸或谷氨酸残基,但在没有结构比对的情况下很难识别这些残基。
Tc1–Mariner
Tc1–Mariner 超家族普遍存在于真核生物中,具有两个 TIRs 和一个转座酶 ORF 的简单结构。它的众多家族强烈倾向于插入 TA 附近,产生 TA TSDs。
hAT
hAT 超家族的成员具有 8 bp 的 TSDs,相对较短的 TIRs(5–27 bp)及总长度少于 4 kb。该名称来源于三个描述较好的TE家族:果蝇中的 hobo、玉米中的 Ac-Ds 和金鱼草中的 Tam3。
Mutator
多样化的 Mutator 超家族也存在于所有真核生物界。尽管其 TIRs 可以延伸到数百个碱基对,有时却非常短或难以检测到。Mutator TEs 产生 9-11 bp 的 TSDs。Merlin 超家族的 TIRs 同样从几十个到几百个碱基对不等;这些 TEs 被 8-9 bp 的 TSDs 所包围。尽管编码 DDE 转座酶的完整功能 Merlin 元件超过 10 kb,但缺失衍生物可能仅有几百个碱基对。这个超家族的成员只在动物和真细菌中被描述。
Transib
Transib 超家族的转座酶含有上述的 DDE 基序;此外,它与参与 V(D)J 重组的 RAG1 蛋白有关。到目前为止,Transib TEs 只在果蝇和蚊子中被发现。最初在昆虫基因组中发现的 P 超家族现已知也存在于后生动物和藻类 Chlamydomonas reinhardtii 中。
P
P元件产生8 bp的TSDs。
piggyBac
piggyBac 超家族主要但不仅限于动物,插入偏向于 TTAA 附近。
PIF–Harbinger
PIF–Harbinger 超家族同样表现出对靶位点的偏好,但倾向于 TAA。这些 TEs 包含两个 ORFs,一个编码 DNA 结合蛋白,另一个编码 DDE 转座酶。
CACTA
CACTA超家族的TEs同时携带转座酶和第二个功能不明的ORF。在植物中,短TIRs以高度保守的CACTA(有时是CACTG)基序结尾,包围3 bp的TSDs;而在动物和真菌中,CCC取代了CACTA基序,产生2 bp的TSDs。TIRs通常包围复杂的亚末端重复阵列。
Ctypton 目
我们将 Crypton TEs 纳入亚类1的第二个目,尽管它们至今仅在真菌中被发现。它们含有一种酪氨酸重组酶,如某些噬菌体、IS 和 DIRS 样逆转座子,但缺乏 RT 域,这表明它们通过 DNA 中间体转座。它们的转座可能涉及圆形分子与 DNA 靶点之间的重组,需要切割两个 DNA 链,因此归入亚类1。它们缺乏TIRs,但似乎由于重组和整合产生了TSDs。
亚类 2

亚类 2 的 DNA TEs 的转座过程保留原始拷贝,通过复制而不是双链切割进行转座,与亚类 1 的机制截然不同。这些“复制-粘贴”型 TEs 通过复制而仅位移一条链进行转座。它们被归入 II 类反映了共同缺乏 RNA 中间体的特征,但不一定有共同的进化起源。
Helintron 目
Helitron 目中的元件似乎通过滚环机制复制,仅切割一条链,不生成 TSDs。Helitron 的末端由 TC 或 CTRR 基序(R 为嘌呤)和在 3′端前几对碱基的短发夹结构定义,尽管这似乎不是一个真正的诊断特征。自主的 Helitrons 编码一种 Y2 型酪氨酸重组酶,类似于细菌 IS91 滚环转座子中发现的重组酶,带有解旋酶域和复制启动活性。它们也可能编码单链结合蛋白或其他蛋白质。Helitrons 在玉米中得到了最好的表征,其中大多数是非自主衍生物。奇怪的是,许多玉米 Helitrons 携带了从宿主基因组捕获的基因片段。尽管 Helitrons 主要在植物中被描述,但它们也存在于动物和真菌中,占秀丽隐杆线虫基因组的 2%,占蝙蝠物种 Myotis lucifugus 基因组的至少3%。
Maverick/Polintons 目
Maverick(也称为Polintons)目中的 TEs 非常大,达到 10–20 kb,具有长 TIRs。它们编码多达 11 种蛋白质,但数量和顺序各异。一些蛋白质与各种 DNA 病毒的蛋白质显示出有限的同源性。Mavericks 编码 DNA 聚合酶 B 和一种与某些 I 类 TEs 中发现的整合酶相关的 c-int 类型 INT,但不含 RT,这表明它们通过不含 RNA 中间体的复制转座过程。这一过程可能通过单链切割后细胞外复制然后整合到新位点来进行。迄今为止,Mavericks 已零星地出现在各种真核生物中,但尚未在植物中发现。
参考
公众号 | BioArt植物 | 神奇的转座子:序言
公众号 | BioArt植物 | 转座子之母:芭芭拉·麦克林托克
公众号 | BioArt植物 | 神奇的转座子:转座子的分类
公众号 | BioArt植物 | 神奇的转座子:植物中转座子的分布

