在基因预测之前,一般首先会对全基因组进行重复序列鉴定和屏蔽。
1. 重复序列
基因组DNA的总含量与物种的进化复杂性无关,甚至矛盾,即所谓的C值矛盾。编码基因数量在不同物种中总体相似,而造成基因组大小显著差异的主要来自重复序列。植物基因组中重复序列通常占很大比例,分布在着丝粒、端粒及染色体各区域。根据序列排列方式可以将重复序列分为两类:一类是串联重复序列,重复单元首尾相连,成串排列;另一类为散在重复序列,其排列方式不是首尾相连簇集在一起,而是散布在不同位置,转座子(transposable element)即是典型的散在重复序列。
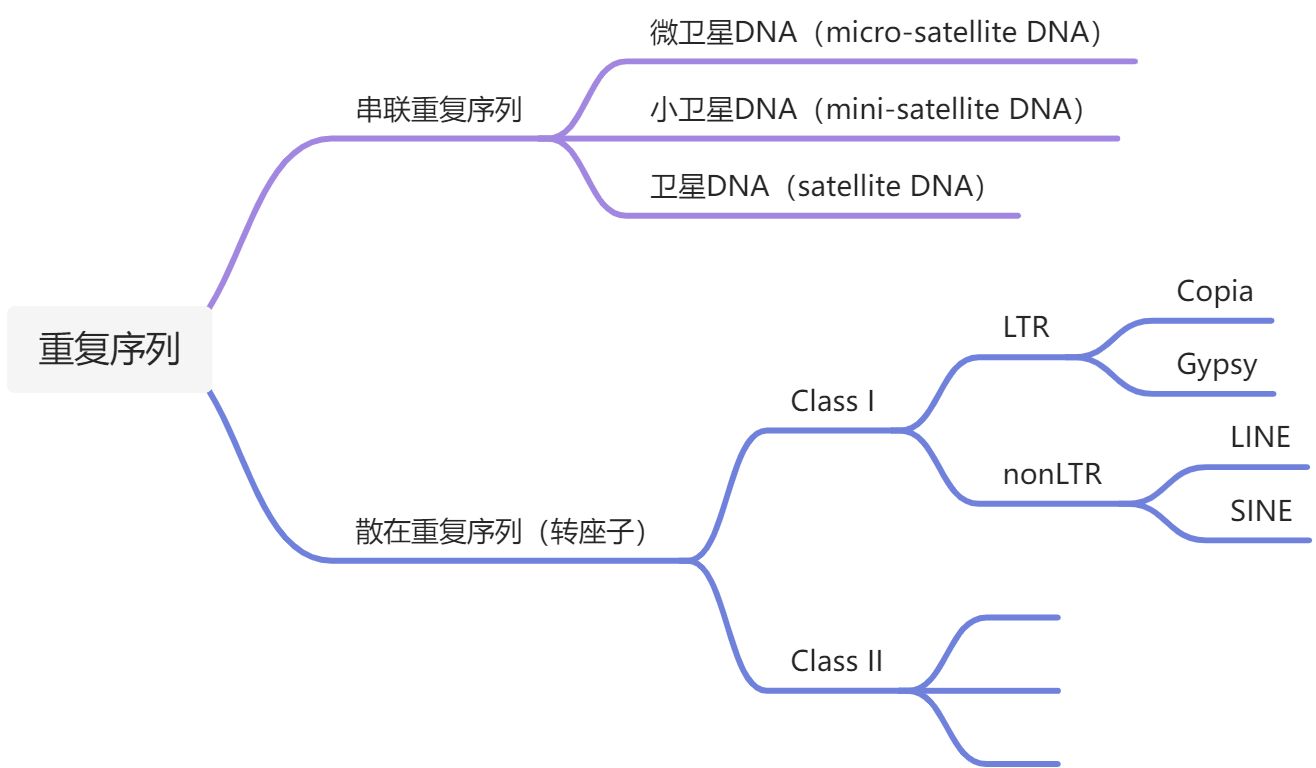
1.1 串联重复序列
串联重复序列按照其重复单元的长短可分为三类:
1.1.1 微卫星DNA
微卫星DNA(micro-satellite DNA)序列,又称短串联重复序列(short tandem repeats, STR)或简单重复序列(simple sequence repeats, SSR),是一类由几个核苷酸(一般为1~6bp)为重复单元簇集而成的长达几十个重复单元的串联重复序列。
微卫星DNA可分布在整个基因组的不同位置上,包括编码和非编码区域。这类重复序列最大的特点是长度的高度变异性,突变率比基因组其他部分高得多。同时,微卫星DNA两端的序列多是相对保守的单拷贝序列,因而可根据两端的序列设计一对特异的引物,扩增每个位点的微卫星DNA序列。微卫星DNA的丰富多态性、共显性、基因组位置上广泛分布等特性,使得微卫星DNA成为遗传标记受到人们的普遍关注。
在植物微卫星DNA中,AG/GT是最常见的,而在动物中,A和AC重复是最常见的。
1.1.2 小卫星DNA
小卫星DNA(mini-satellite DNA)序列通常是指7~100bp(多数为15bp左右)为重复单元的串联重复序列,长度多在0.5~30kb。
1.1.3 卫星DNA
卫星DNA(satellite DNA)通常包含富含AT的重复单元,一个重复单元长度通常在150~400bp,形成长度可达100Mb的串联重复序列。
卫星DNA位于染色体的各个不同区域,主要在着丝粒周围及亚端粒区域。重复单元长度多为135~195bp和315~375bp。卫星DNA在植物基因组中占比多的高达20%,拷贝数可高达10^6~10^7。一个物种中有多种不同的卫星DNA家族,一个家族也可能出现在不同物种中。卫星DNA具有很高的进化速率,多数卫星DNA是物种或属特异性的。
1.2 转座子
基因组上存在可移动的元件,是由美国科学家麦克林托克在玉米中发现的,后来这类元件被命名为转座子(transposable element)。
转座子依据其转座机制不同可以分为两大类:
反转座子(retrotransposon, Class I element):“复制-粘贴”机制
DNA转座子(DNA transposon, Class II element):“剪接-粘贴”机制
2. 重复序列鉴定
可以使用三种方法来鉴定重复序列:
2.1 基于重复序列数据库
所有物种都具有一个共同的祖先,其基因组中的重复序列在不同物种之间是具有相似性的。收集一些物种中已知的重复序列做成一个数据库,再将待分析物种的基因组序列和数据库中的重复序列进行比对,从而对重复序列进行鉴定。
基于重复序列数据库的方法鉴定重复序列的速度较快,。缺点是若数据库不完善,则鉴定的重复序列很少,适合于模式物种的重复序列分析(重复序列数据库中一般包含有模式物种较全的重复序列数据)。
2.2 基于重复序列的特定结构(signature)
LTR类型的重复序列具有一些特定的信号,例如其首尾部分较长的两端序列具有较高的相似性。基于重复序列的一些特点可以对其进行鉴定。
2.3 从头预测
通过将基因组序列和自身进行比较,从而找出在基因组上重复次数较多的序列,即为重复序列。该方法比较消耗计算。
参考
xuzhougeng | 基因组的重复序列注释的个人心得
xuzhougeng | 「基因组注释」MITE-Hunter鉴别基因组的MITE序列

