参考
Pfam数据库:
https://pfam.xfam.org/ (已失效)
https://www.ebi.ac.uk/interpro/entry/pfam/
https://www.ebi.ac.uk/interpro/download/Pfam/
CDD数据库
SMART数据库
HMMER
http://eddylab.org/software/hmmer3/
1. 介绍
高质量结构域数据库
HMMER
HMMER是基于隐马尔可夫模型,用于生物序列分析工作的一个非常强大的软件包,它的一般用途是识别同源蛋白或核苷酸序列和进行序列比对。
常用程序:
hmmbuild: 使用多重比对序列构建HMM模型
--amino: 蛋白质比对序列
--dna: DNA比对序列
--rna: RNA比对序列
hmmsearch: 寻找相似序列
-o: 直接输出结果到文件,不是标准输出
--tblout: tblout格式输出
--domtblout: domtblout格式输出
--pfamtblout: pfamtblout格式输出
hmmalignphmmerhmmscan
2. 下载hmm文件
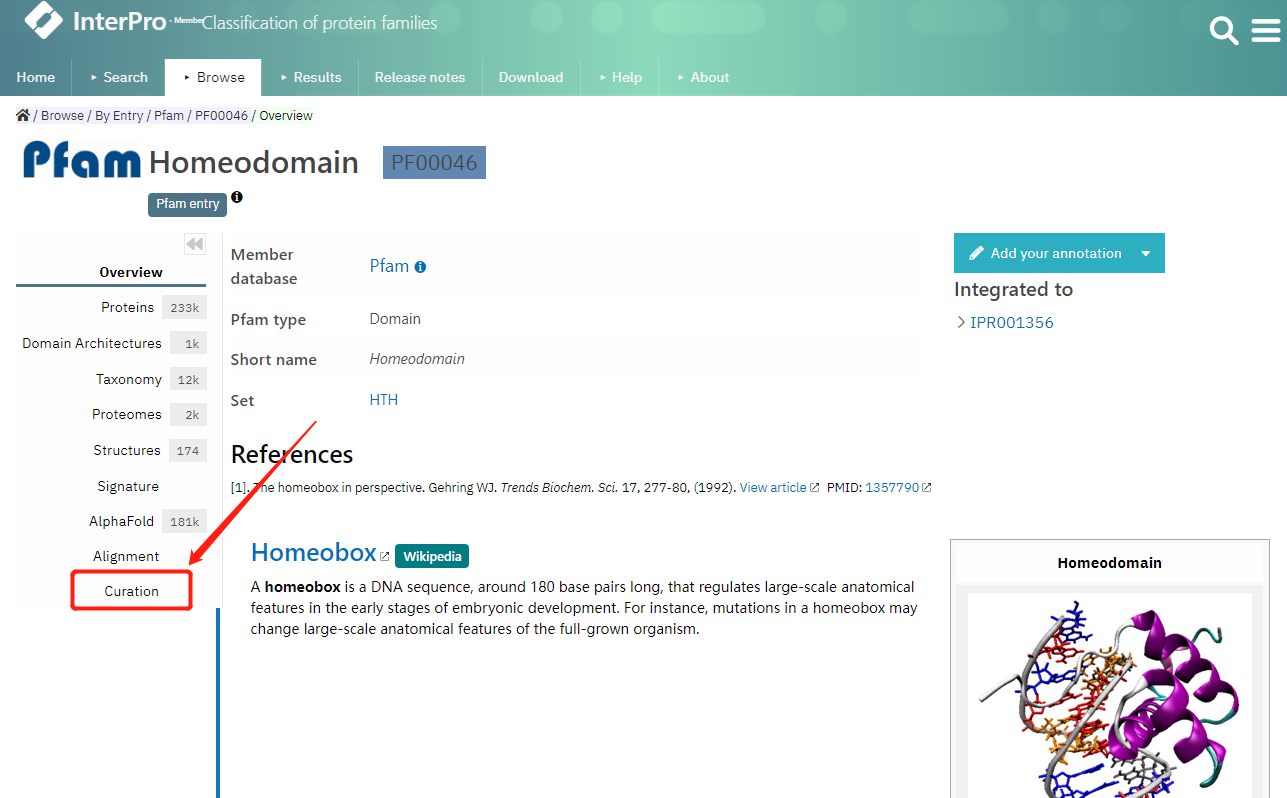
PF00046(https://www.ebi.ac.uk/interpro/entry/pfam/PF00046/)
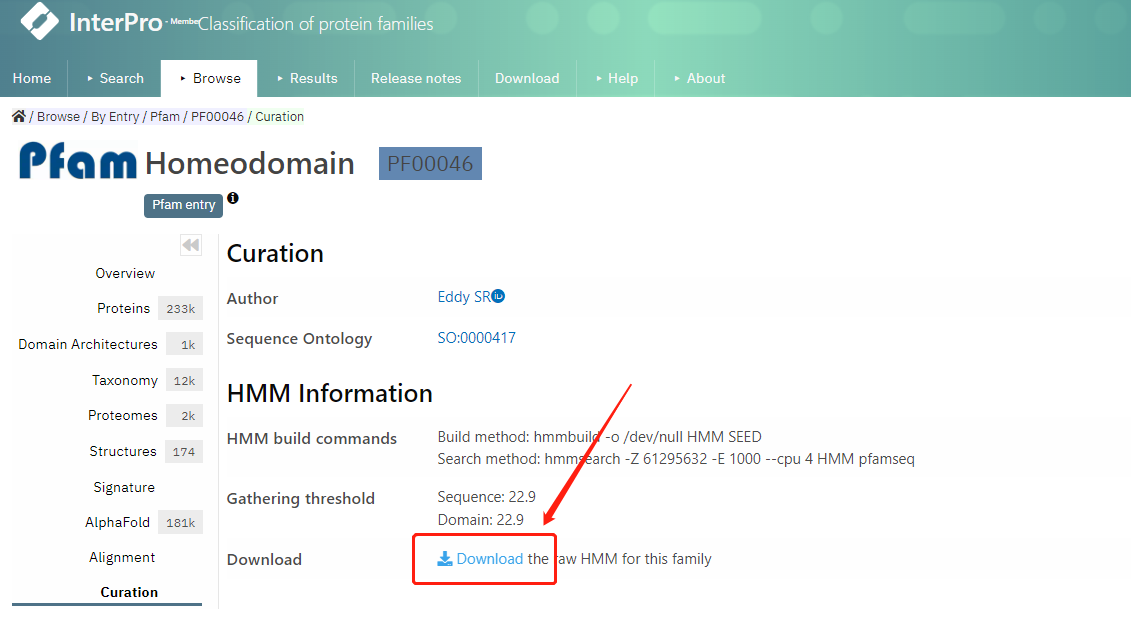
下载得到:PF00046.hmm.gz
3. hmmer鉴定
gunzip PF00046.hmm.gzhmmsearch PF00046.hmm pep.fa --cut_tc --domtblout PF00046.domtblout -o PF00046.hmmout# --cut_tc: 输出结果阈值设置# 基于E值进一步进行过滤awk '{if($7 < 1e-5 && $1 !~ /^#/) print $0}' PF00046.domtblout > PF00046.domtblout.filter# 提取预测的MAPK基因ID列表awk '{print $1}' PF00046.domtblout.filter | sort -u > Ft.MAPK.domtblout.filter.id

