- 参考
- 1. 成员数据库
- CATH-Gene3D">1.1 CATH-Gene3D
- CDD">1.2 CDD
- HAMAP">1.3 HAMAP
- PANTHER">1.4 PANTHER
- Pfam">1.5 Pfam
- PIRSF">1.6 PIRSF
- 1.7 PRINTS
- PROSITE profiles">1.8 PROSITE profiles
- PROSITE patterns">1.9 PROSITE patterns
- 1.10 SFLD
- SMART">1.11 SMART
- SUPERFAMILY">1.12 SUPERFAMILY
- TIGRFAMS">1.13 TIGRFAMS
- 2. 使用
参考
InterPro 官网:http://www.ebi.ac.uk/interpro/
InterPro provides functional analysis of proteins by classifying them into families and predicting domains and important sites. To classify proteins in this way, InterPro uses predictive models, known as signatures, provided by several different databases (referred to as member databases) that make up the InterPro consortium. We combine protein signatures from these member databases into a single searchable resource, capitalising on their individual strengths to produce a powerful integrated database and diagnostic tool.
InterPro 提供蛋白质的功能分析,将它们分类为家族,预测结构域和重要位点。为了以这种方式对蛋白质进行分类,InterPro 使用了被称为 signatures 的预测模型,这些模型由组成 InterPro 联盟的几个不同数据库(称为成员数据库)提供。我们将来自这些成员数据库的蛋白质 signatures 组合成一个可搜索的资源,利用它们各自的优势生成一个强大的集成数据库和评估工具。

1. 成员数据库
1.1 CATH-Gene3D
1.2 CDD
CDD,即 Conserved Domain Database,保守结构域数据库,主要用来对蛋白质或核酸序列进行功能和保守结构域注释。
CDD 数据库收集了许多基于多序列比对的保守结构域模型,这些模型以 PSSMs 来体现。使用 PRS-BLAST 能将查询序列比对到这些模型,从而获得注释结果。
CD-Search
Batch CD-Search
PRS-BLAST 本地注释
1.3 HAMAP
1.4 PANTHER
1.5 Pfam
1.6 PIRSF
1.7 PRINTS
1.8 PROSITE profiles
1.9 PROSITE patterns
1.10 SFLD
1.11 SMART
1.12 SUPERFAMILY
1.13 TIGRFAMS
2. 使用
2.1 网页版
可参考:
公众号 | 组学大讲堂 | 预测蛋白质保守结构域神器-InterPro工具使用详解
缺点:
每次最多输入 25 条蛋白质序列。
2.2 InterProScan 本地
下载地址:http://www.ebi.ac.uk/interpro/download/InterProScan/
https://mp.weixin.qq.com/s/DMRyDsS9B-CmHTPilCrqdQ
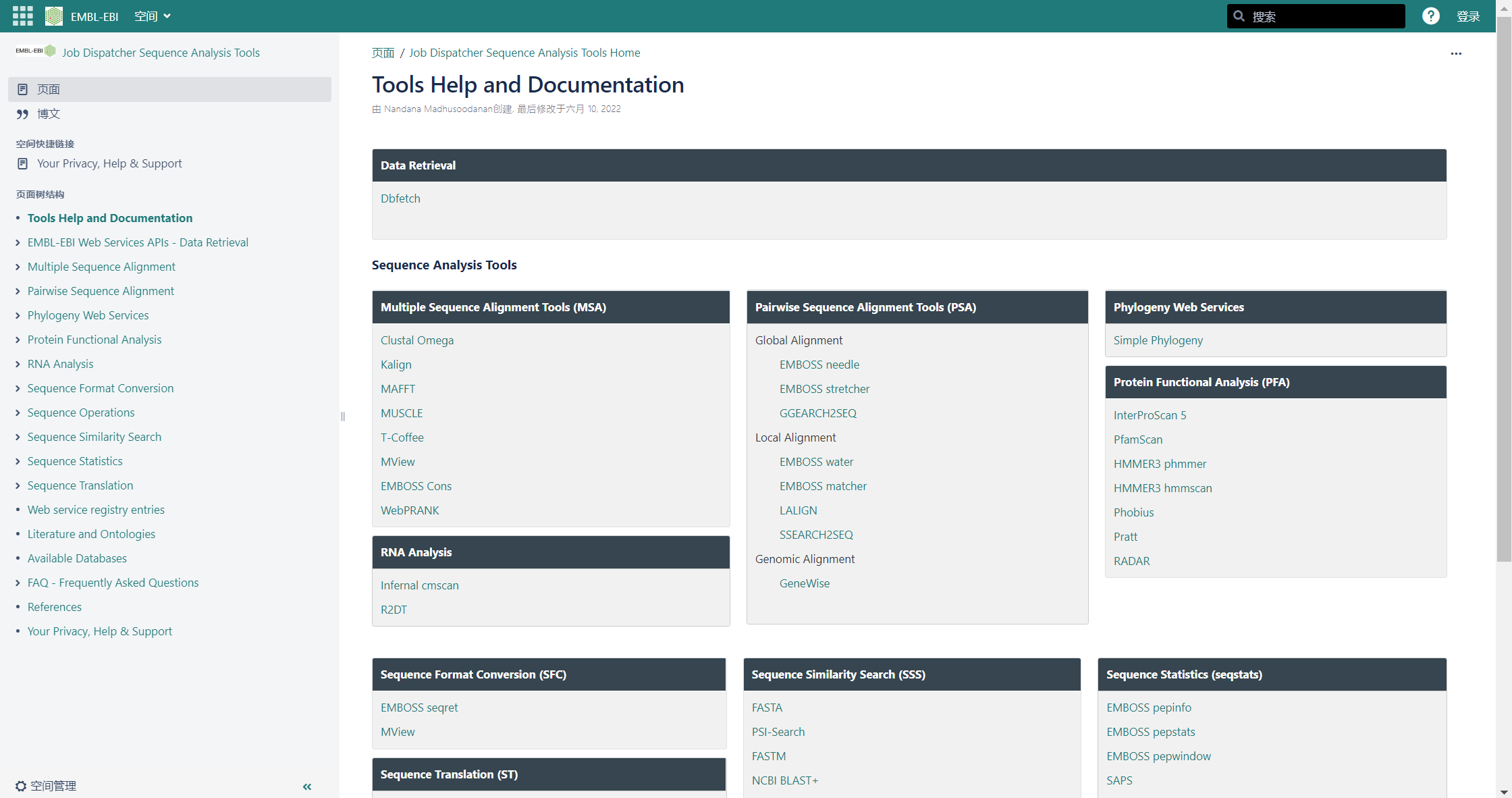
地址:https://www.ebi.ac.uk/seqdb/confluence/display/JDSAT/Tools+Help+and+Documentation

