- What does OrthoFinder do?
- Getting started with OrthoFinder
- Running OrthoFinder
- OrthoFinder Results Files
- Phylogenetic Hierarchical Orthogroups Directory
- Orthologues Directory
- Orthogroups Directory (deprecated)
- Gene Trees Directory
- Resolved Gene Trees Directory
- Species Tree Directory
- Comparative Genomics Statistics Directory
- Gene Duplication Events Directory
- Orthogroup Sequences
- Single Copy Orthologue Sequences
- WorkingDirectory
- Understanding Orthology
- Trees from MSA: “-M msa”
- Advanced usage
- Methods
- Command line options
- 参考
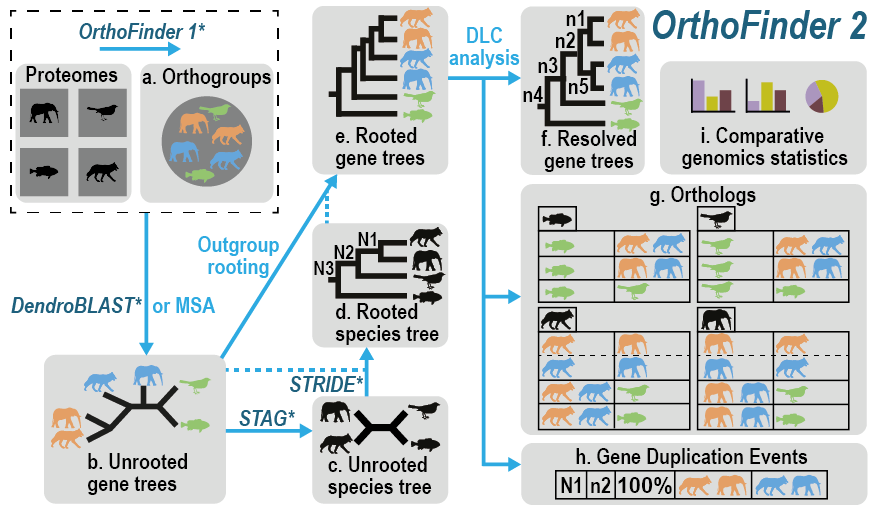
What does OrthoFinder do?
OrthoFinder是一个快速、准确且全面的比较基因组学平台。它能够找到同源基因组(orthogroups)和同源基因(orthologs),在所有同源基因组中推断有根基因树(rooted gene trees),并识别这些基因树中的所有基因复制/加倍事件(gene duplication events)。它还能为正在分析的物种推断一个有根物种树(rooted species tree),并将基因树中的基因复制事件映射到物种树的分支上。OrthoFinder还为比较基因组学分析提供了全面的统计信息。OrthoFinder使用简单,你只需要准备一组蛋白序列文件(每个物种一个),格式为FASTA。
有关更多详情,请参阅以下OrthoFinder论文。
Getting started with OrthoFinder
Installing OrthoFinder on Linux
conda install -c bioconda orthofinder
Running OrthoFinder
在示例数据类型上运行OrthoFinder:
OrthoFinder/orthofinder -f OrthoFinder/ExampleData
要在您自己的数据集上运行,请将 “OrthoFinder/ExampleData” 替换为包含您的输入FASTA文件的目录,每个物种一个文件。OrthoFinder 将搜索具有以下任何文件名扩展名的输入 FASTA 文件:
- .fa
- .faa
- .fasta
- .fas
- .pep
OrthoFinder Results Files
https://davidemms.github.io/orthofinder_tutorials/exploring-orthofinders-results.html
Phylogenetic Hierarchical Orthogroups Directory
从版本2.4.0开始,OrthoFinder通过分析根基因树推断出HOGs,HOGs即每个层次级别(即物种树中的每个节点)的同源基因组。这是一种比其他所有方法更准确的同源基因组推断方法,其他方法都基于基因相似性/图,也是以前OrthoFinder所使用的方法(已弃用的Orthogroups/Orthogroups.tsv文件)。根据Orthobench基准测试,这些新的同源基因组比OrthoFinder 2的同源基因组(Orthogroups/Orthogroups.tsv)准确性提高了12%。通过包含外类群物种(outgroup species),可以进一步提高准确性(在Orthobench上提高20%),这有助于解释根基因树。
确保OrthoFinder使用的物种树准确无误非常重要,以最大限度地提高HOGs的准确性。要使用不同的物种树重新分析,请使用选项 -ft PREVIOUS_RESULTS_DIR -s SPECIES_TREE_FILE。这仅运行最终的分析步骤“from trees”,速度相对较快。如果使用了外类群物种,请参考 “Species_Tree/SpeciesTree_rooted_node_labels.txt“,确定包含您所需同源基因组的N?.tsv文件。
- N0.tsv 是一个制表符分隔的文本文件。每一行包含属于单个同源基因组的基因。每个同源基因组的基因按物种组织成列,每个物种一列。额外的列给出了 HOG(分层同源基因组)ID 和从哪个基因树节点确定了 HOG(注意,这可能在包含基因的支系根部之上)。这个文件有效地替代了使用 MCL 进行马尔可夫聚类的 Orthogroups/Orthogroups.tsv 中的同源基因组。
- N1.tsv、N2.tsv 等:从对应于物种树 N1、N2 等支系的基因树中推断出的同源基因组。由于 OrthoFinder 现在在物种树的每个层次级别推断同源基因组,因此现在可以在分析中包含外围物种,然后使用 HOG 文件获取物种树内所选支系的同源基因组定义。
(分层同源基因组拆分:在分析基因树时,一个嵌套的分层组(除了 N0,即所有物种的最近共同祖先水平的 HOG)有时可能会丢失最早分化物种的基因,然后在第一个存在的基因之前复制。然后,两个最早分化的支系将成为拟同源基因,尽管证据表明它们属于同一个 HOG。对于大多数分析,通常最好将这些支系拆分成单独的组。可以使用选项 ‘-y‘ 请求此操作。)
Orthologues Directory
Orthologues目录包含一个子目录,每个子目录代表一个物种,其中包含物种成对比较的文件,列出了该物种对之间的同源基因。根据同源基因分化以来的基因复制事件,同源基因可以是一对一、一对多或多对多的(有关更多详细信息,请参阅“同源基因组、同源基因和拟同源基因”部分)。每个文件中的每一行都包含一个物种中的基因,这些基因是另一物种中的基因的同源基因,并且每一行都与包含这些基因的同源基因组相互参照。
Orthogroups Directory (deprecated)
现在应该使用 Phylogenetic_Hierarchical_Orthogroups/ 目录中的orthogroups。它们是使用有根基基因树进行鉴定的,并且比准确度提高了12%至20%。
- Orthogroups.tsv(已弃用)是一个制表符分隔的文本文件。每一行包含属于单个 orthogroup 的基因。每个 orthogroup 中的基因按照物种排列在一列中。现在应该使用 Phylogenetic_Hierarchical_Orthogroups/N0.tsv 中的 orthogroups。
- Orthogroups_UnassignedGenes.tsv 是一个制表符分隔的文本文件,其格式与 Orthogroups.csv 相同,但包含所有未分配到任何 orthogroup 的基因。
- Orthogroups.txt(遗留格式)是另一个文件,包含了 Orthogroups.tsv 文件中描述的 orthogroups,但是使用了 OrthoMCL 的输出格式。
- Orthogroups.GeneCount.tsv 是一个制表符分隔的文本文件,其格式与 Orthogroups.csv 相同,但包含了每个 orthogroup 中每个物种的基因数量。
- Orthogroups_SingleCopyOrthologues.txt 是一个包含了每个物种中恰好一个基因的 orthogroups 的列表,即它们包含了一对一的同源基因。它们非常适合用于物种间比较和物种树推断。
Gene Trees Directory
由所有具有4条及以上序列的orthogroup推断的一个有根的系统发生树(大多数系统发生树推断程序要求至少4条序列)。
Resolved Gene Trees Directory
使用OrthoFinder混合物种重叠/重复-丢失合并模型(hybrid species-overlap/duplication-loss coalescent model)对具有4条及以上序列的每个orthogroup进行了根化系统发生树推断。
Species Tree Directory
- SpeciesTree_rooted.txt 从所有orthogroups推断出的STAG物种树,包含内部节点处的STAG支持值,并使用STRIDE进行根化。
- SpeciesTree_rooted_node_labels.txt 与上述树相同,但节点被赋予标签(而不是支持值),以允许其他结果文件交叉参考物种树中的分支/节点(例如,基因重复事件的位置)。
Comparative Genomics Statistics Directory
Gene Duplication Events Directory
Orthogroup Sequences
Single Copy Orthologue Sequences
WorkingDirectory
Understanding Orthology
Trees from MSA: “-M msa”
Advanced usage
Methods
Command line options
Options for starting an analysis
Options for stopping an analysis
Options controlling the workflow
Options controlling the programs used
Further options

