DNA甲基化简介
DNA甲基化是指DNA序列上特定的碱基在DNA甲基转移酶的催化作用下,以S-腺苷甲硫氨酸(S-adenosylmethionine, SAM)作为甲基供体,通过共价键结合的方式获得一个甲基基团的化学修饰过程。DNA甲基化是最早被发现、研究最深入的表观遗传调控机制之一。这种甲基化修饰可以发生在胞嘧啶的C5位(m5C)、腺嘌呤的N6位(m6A)及鸟嘌呤的G7(m7G)位等位点。它们分别由不同的DNA甲基化酶催化,大多发生在基因启动子区,但是启动子附近的CpG岛区域通常不是甲基化的。基因启动子区的甲基化可导致转录沉默,即抑制基因表达。DNA甲基化过程是可逆的,而去甲基化通常意味着基因表达被重新激活。发生在CpG二核苷酸胞嘧啶上第5位碳原子的甲基化过程,其产物成为5-甲基胞嘧啶(5mC),是真核生物DNA甲基化的主要形式。
基因组中DNA甲基化的分布在不同物种甚至不同个体样本中表现出不同的模式。这种模式通常反映了DNA甲基化在基因表达、转座子抑制以及对发育或环境线索反应中的功能。以单碱基分辨率解析DNA甲基化的分布对于人们理解胚胎发育、细胞分化、遗传印记、转座子失活等表观遗传调控机制具有重要意义。WGBS实验原理
人们开发了多种方法来测定样本中的DNA甲基化水平。全基因组重亚硫酸盐测序WGBS(Whole-genome bisulfite sequencing)被视为是检测甲基化水平的“金标准”,其原理是用亚硫酸氢钠处理,将基因组中未发生甲基化的C碱基转换成U,经过PCR扩增后变成T,与原本具有甲基化修饰的C碱基区分开来,再结合高通量测序,与参考序列比对,即可判断CpG/CHG/CHH(H为A或C或T)位点是否发生甲基化。
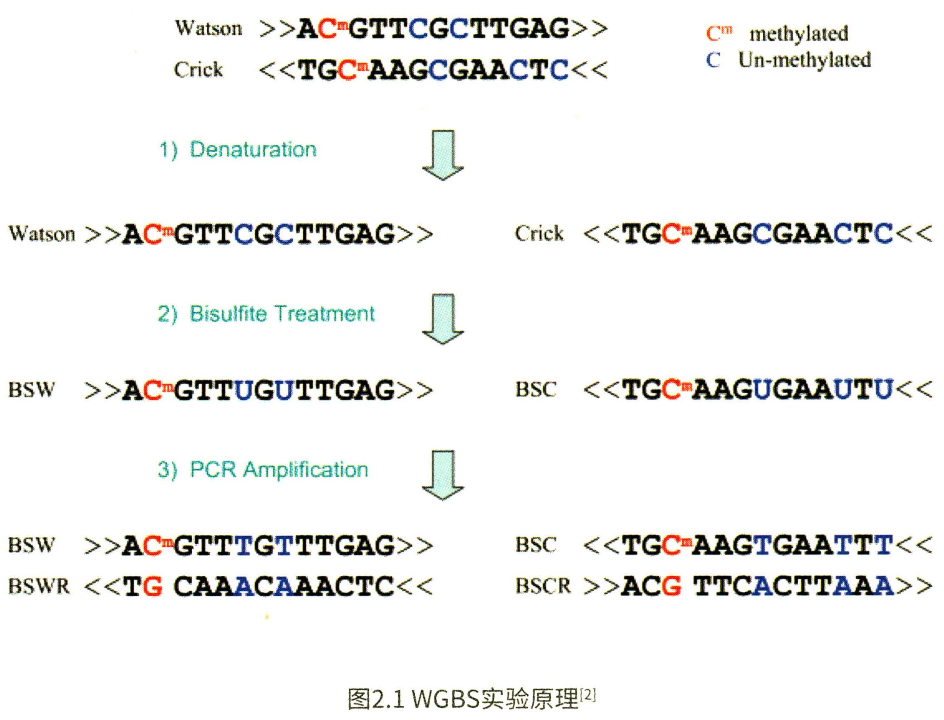
WGBS数据分析
数据质控与过滤
首先需要对获得的样本测序数据进行质量评估和过滤。随着读序长度的增加,测序错误率倾向升高;另外,读序上包含的引物会降低读序匹配到基因组上的准确率。因此,要对数据进行去接头、低质量读序等处理。对于利用二代测序技术获得的数据来说,已有成熟的软件进行质控。
WGBS使用二代测序平台,数据为fastq文件。因而WGBS的数据质控与过滤与其他二代数据一致。只要过滤掉接头与低质量碱基序列即可。方法可以采用Trimmomatic、Skewer、cutadapt等软件。测序质量可以使用FastQC进行检查。与常规二代测序结果A/T和G/C含量一致不同:WGBS的fastq文件中Forward序列大量C碱基转化成T碱基,而Reverse序列大量G碱基转化成了A碱基,从而会出现A/T分离和G/C分离的现象。
与参考基因组比对/去冗余/识别甲基化
质控后,需要将测序数据比对到参考基因组序列上。研究者基于短序列匹配算法,开发了专门处理重亚硫酸盐转换后的读序比对工具和算法。
通过比对结果可以获悉测序数据的深度、读序在全基因组上的分布及在基因组上的覆盖度情况。从读序的基因组位置中获得每个胞嘧啶的甲基化读序数和非甲基化读序数,然后计算某个胞嘧啶的甲基化水平,观察甲基化程度。
目前常用的两个WGBS分析软件Bismark与BSMAP的甲基化识别算法并不相同。这两种软件的基因组准备,数据比对,甲基化位点识别的方法也不同。
BSMAP的原理:首先,bisulfite sequencing的过程中可以将reads分为BSW、BSWR、BSC、BSCR四种类型。分别代表bisulfite处理的Waston链、bisulfite处理的Waston链PCR后互补链、bisulfite处理的Crick链、bisulfite处理的Crick链PCR后互补链。BSMAP采用“wild-card”策略进行比对,将参考序列构建一个HASH seed table,以seed序列为key,以基因组坐标为值。在seed table中包含了C位点的所有可能转换类型seed。为了进一步提高比对效率,BSMAP将碱基序列转换成了二进制表格A(00)、T (11)、C(01)、G(10),并按照表格方式判断是否匹配。
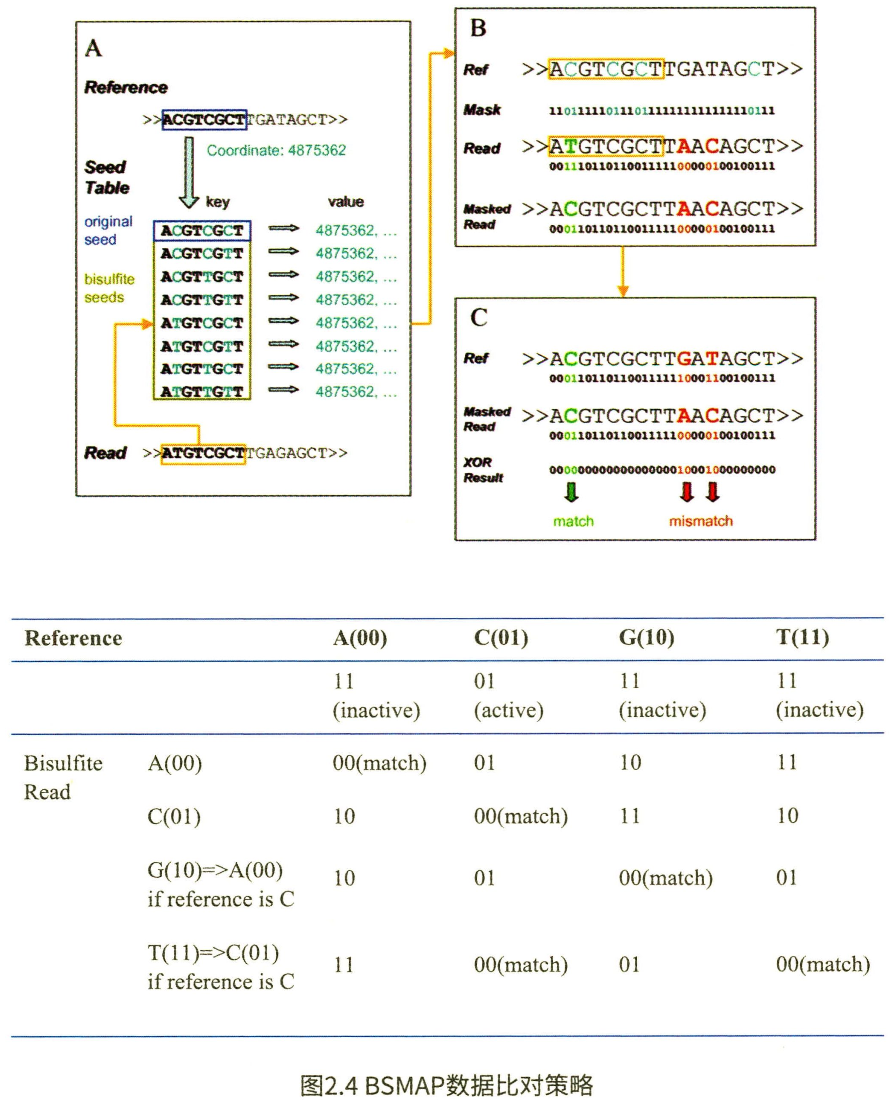
BSMAP的操作比较简单,并且不需要预先对参考序列进行建库,运行速度也较Bismark更快。但BSMAP是相对单纯的比对工具,其下游分析工具较少。
Bismark的原理为:基于非链特异性文库,Bismark将序列按C->T,G->A进行两种转换,同时也将基因组进行两种转换。通过对测序数据进行4轮比对,综合选取最佳比对结果判断甲基化情况。如果文库是链特异性的,因为测序结果两条链来源是确定的,仅需要进行两轮比对。判断时,根据参考基因组上C位点比对上的碱基类型来检测甲基化位点。若比对上的碱基是C,则发生了甲基化;反之,比对上的碱基是T,则没有发生甲基化。
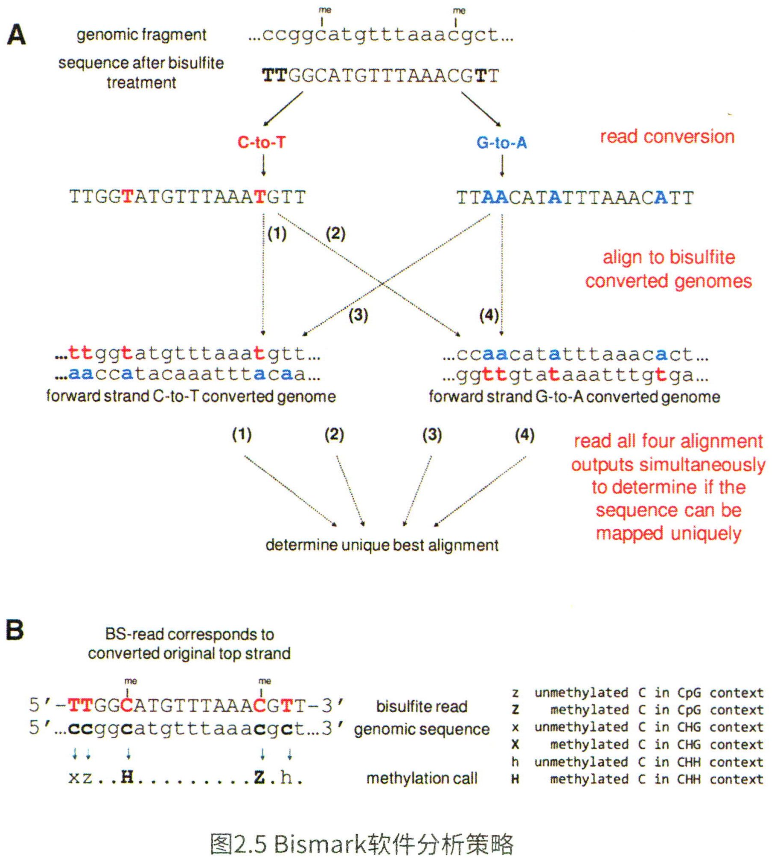
Bismark调用bowtie或bowtie2进行比对,所以需要对基因组的两种转换均进行建库。而由因为进行了多轮的比对,Bismark的运行时间较BSMAP要长。但Bismark的功能更为齐全,因而应用更为广泛。
甲基化水平及转化率计算
甲基化水平的计算采用以下公式:
$ Methylation level = methylated reads\ /\ (methylated reads + unmethylated reads) $
为了准确评估转化率(Bisulfite conversion rate),通常会在样品DNA中添加lambda噬菌体DNA作为spike-in。因此在软件分析时,需要在参考基因组中加入lambda DNA序列。因为lambda DNA是完全未甲基化的,因此结果中lambda DNA甲基化水平可以用于评估实验中bisulfite处理非甲基化C的转化效率。通常成功的WGBS实验转化率应该大于99%。
差异甲基化位点识别
差异甲基化区域(Differentially Methylated Regions, DMR)是在两组甲基化数据中存在甲基化水平差异的区域。虽然单个位点的甲基化可能与基因表达相关,造成疾病风险,但更多的报道认为甲基化的差异是通过几百上千个位点以区域形式影响基因的长时效表达调控。常用的DMR分析工具有DSS,methylKit,BSmooth等。在低覆盖度时,C位点的甲基化水平会偏高,且在重复样品间的波动较大。因此,在进行DMR分析时,推荐根据C位点的覆盖度进行数据过滤,仅保留测序深度大于等于5的C位点进行分析。
WGBS数据具有三个独特的特点可以用于知道DMR检测的严格算法设计;
(1)基因组上甲基化水平有很强的空间临近相关性,因而通常可以用全基因组的平滑曲线估计真正的甲基化水平;
(2)DMR检测的统计学分析应当考虑CpG位点的深度信息;
(3)应使用生物重复间的差异信息进行DMR分析的有效性检验。推荐使用DSS进行DMR分析。
DSS中实施差异甲基化检测程序系针对$ \beta $-二项分布的严格Wald检验。测试统计基于生物学差异(通过离散参数表证)及测序深度来实现。作为DSS实施算法的重要部分,离散参数估计通过贝叶斯层次模型的收缩估计器实现。DSS的优点是能在没有生物学重复的条件下进行,这是因为通过平滑处理,相邻的CpG位点可以被视为伪复制,基于此离散度仍可以在合理的精度下被估算。DSS对于一般实验设计也是有效的,这是通过具有反正弦函数的$ \beta $-二项回归模型来实现的。使用广义最小二乘法对变换后的数据进行模型拟合,与基于广义线性模型的方法相比,可以大大提高计算性能。DSS依赖于Bioconductor的bsseq软件包,该软件包对数据结构的定义清晰,且具有许多实用功能。
更多分析
通过合并相邻甲基化位点,还可以做更多的分析,如邻近基因区域功能注释,邻近顺式调控区域注释,区域保守性检测,区域SNP位点注释;也可以识别已知转录因子基序和在全基因范围对基序从头搜索。对于多样本,通过数据标准化后可以对各个样品甲基化水平进行比较,获取差异DNA甲基化区域。
三代测序检测甲基化原理及分析流程
WGBS以单核苷酸分辨率提供了胞嘧啶的甲基化状态,但实验过程相对繁琐,且短读长在比对到重复序列和单倍型方面具有明显的局限性。如今,长读长测序技术提供了一种更简单的文库制备流程,以PacBio单分子实时测序和Oxford Nanopore Technologies(ONT)纳米孔测序为代表的三代测序在提供长度长测序的同时还可以检测DNA甲基化修饰,而最新的兼顾长度长和准确性的高质量PacBio HiFi数据能够直接检测DNA 5mC,并具有区分单倍型的能力,这省去了额外的重亚硫酸盐转化实验流程,将为DNA甲基化检测技术带来一场革命。
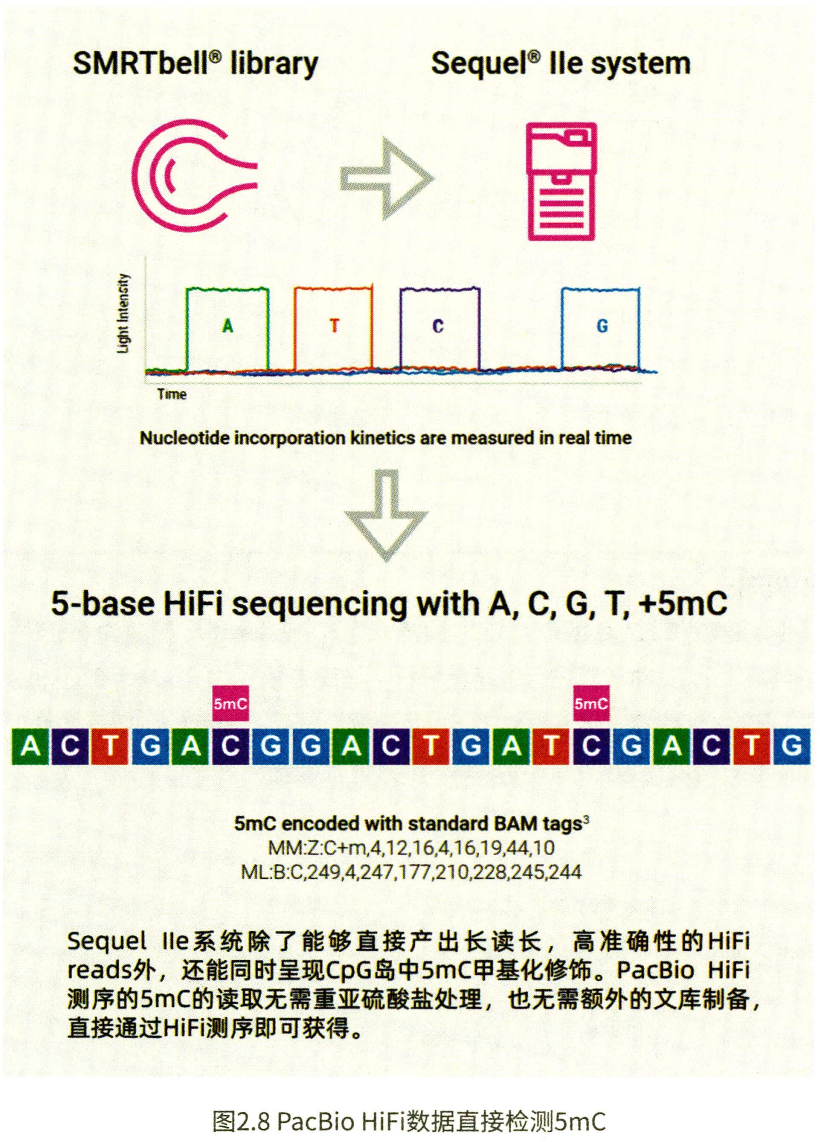
HiFi检测甲基化的原理
PacBio单分子实时测序技术会输出单分子的碱基荧光信号和聚合酶动力学信息,表观遗传修饰(如5mC)会影响聚合酶动力学碱基掺入的速度。数据上即呈现为荧光信号的延迟。利用HiFi技术(对每个单分子重新检测,数据内校正)得到兼顾长读长和准确性的高质量PacBio HiFi数据能够直接检测DNA 5mC,并具有区分单倍型的能力。
ONT纳米孔测序技术利用核算通过蛋白质纳米孔时电流的变化,利用深度学习技术对产生的信号进行编码,最终获得核酸序列信息。这个过程中表观遗传修饰也会影响其电信号的变化(即未修饰与修饰后的相同碱基信号不同)。因而可以利用这种变化分析核酸序列上的碱基修饰。利用纳米孔测序,研究人员可以单核苷酸分辨率直接鉴定DNA和RNA碱基修饰,包括DNA中的5mC、5hmC、6hmC、6mA和BrdU,以及RNA中的m6A。
HiFi检测甲基化的分析流程
基于HiFi的甲基化分析通常包括Subreads转ccs,使用primrose分析ccs数据分析中的5mC信号,数据参考基因组比对,基因组5mC位点识别的几个步骤。其中数据的参考基因组比对结果可以用于常规HiFi识别SV以及分型分析等工作。也可以使用分型分析的结果进行基因组5mC位点分型分析,进一步进行等位染色体差异甲基化位点分析。Revio平台输出即为包含5mC位点信息的HiFi数据,可以从数据比对步骤直接开始分析。

Subreads转ccs:ccs(smrtlink)
采用PB官方smrtlink软件的ccs工具可以将PB sequeII等平台输出的subreads文件转为ccs文件,通过调整参数即可得到酶动力学信号。Revio平台输出为转换后HiFi数据,此步略过。
分析5mC信号:primrose(smrtlink)
primrose也是PB smrtlink软件包内的工具。primrose采用卷积神经网络算法,利用ccs数据中的酶动力学信号预测序列中的CpG 5mC。Revio平台输出为转换后HiFi数据,此步略过。
数据比对:pbmm2
pbmm2是PacBio官方对minimap2进行的包装,使之适用于PacBio的原始输出数据。因其可以使用bam文件作为输入且输出结果保留5mC位点信息,采用该软件进行HiFi数据比对。注:minimap2需要以bam/sam作为输入文件,并使用-Y参数。
5mC位点识别:pb-CpG-tools
pb-GpG-tools是PB官方提供的5mC分析软件,该软件提供了“aligned_bam_to_cpg_scores”工具用于从HiFi比对结果中提取甲基化率。aligned_bam_to_cpg_scores可以使用包含MM和ML tags的比对后bam文件作为输入,分析基因组上的甲基化,也可以使用经过whatshap软件分型分析后带有HP tag的bam作为输入,输出分型的甲基化信息。
HiFi数据文件甲基化信息判断
如果数据文件是通过其他渠道(如网上下载)获得的。通常需要判断该数据处于哪个阶段,是否可以进行5mC位点分析。如下有几个判断方法。
(1)只有bam文件可以保留5mC位点信息,如数据为转录后的fastq文件,则不能进行5mC位点分析。
(2)Bam文件的header信息通常包含数据处理的各阶段操作信息。使用samtools查看数据的header信息,判断数据状态。如下bam文件header显示,数据经过了ccs处理并使用了—all—kinetics,随后使用了primrose。说明该数据可以直接进行基因组比对分型5mC位点信息。
(3)此外可根据bam文件每条reads信息的tags判断,可以进行5mC位点识别的HiFi数据必须包含“MM”和“ML”两个tags。
尽管WGBS是一种检测DNA甲基化的经典技术,多年来的研究极大地促进了人们对动植物表观遗传调控的理解。但随着技术的不断革新,三代测序PacBio和Nanopore的长读长在检测天然DNA碱基修饰方面有着独特的优势,虽然使用长读长工具分析表观遗传的数据准确性仍具有挑战性,但从各类样品中检测各种修饰碱基的能力来看,与短读长亚硫酸氢盐测序相比,长读长测序具有简单高效的明显优势,其应用定将也会越来越多。
参考
《植物基因组学》樊龙江
《生物信息学》樊龙江
公众号 | 万字详解 | 万字详解 | 植物DNA甲基化的调控机制和功能(一)
公众号 | 万字详解 | 万字详解 | 植物DNA甲基化的调控机制和功能(二)
公众号 | 万字详解 | 万字详解 | 植物DNA甲基化的调控机制和功能(三)
bilibili | DNA甲基化背景介绍
白墨 | 一文读懂DNA甲基化与BS-seq
知乎 | 基因组甲基化 基础知识梳理
BatMeth2
bismark
MethlDackel
methyKit
- 特征
DNA甲基化是最早发现的遗传修饰之一。大量研究表明,DNA甲基化能引起染色质结构、DNA构象、DNA稳定性及DNA与蛋白质相互作用方式的改变,从而控制基因表达。DNA甲基化常见于基因5’-CG-3’序列,在甲基转移酶的催化下,DNA的CG两个核苷酸的胞嘧啶被选择性地添加甲基,形成5-甲基胞嘧啶。
- 甲基化类型
5-甲基胞嘧啶、
甲基化位点可随DNA的复制而遗传,因为DNA复制后,甲基化酶可将新合成的为甲基化的位点进行甲基化。
甲基化的影响
DNA甲基化可引起基因的失活
- 甲基化测定
A survey of the approaches for identifying differential methylation using bisulfite sequencing data
重亚硫酸盐测序
基于限制性内切酶测序
靶向富集甲基化位点测序
Nanopore直接DNA测序
PacBio甲基化测序

