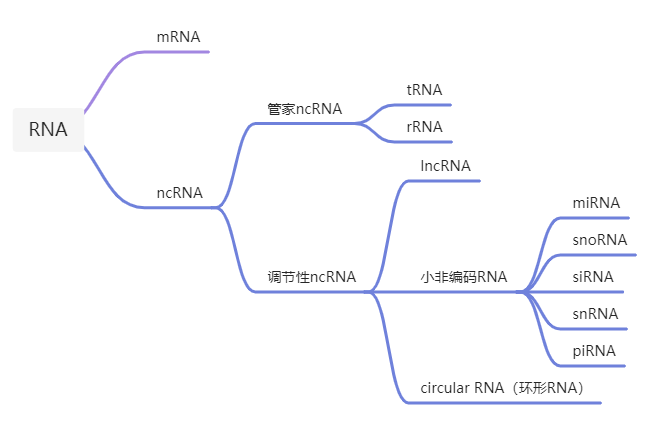
非编码 RNA 包括小非编码 RNA 和长非编码 RNA 两种类型,主要参与蛋白质编码基因调控。
内源性非蛋白质编码小 RNA 广泛存在于高/低等生物体内,通过对靶标 mRNA 直接剪接或抑制其翻译,主要在转录后水平对基因表达起调节作用。
已知的小 RNA 主要分为两大类:
- 微小 RNA
- 小干扰 RNA
在植物和动物体内,miRNA 与 siRNA 的产生机制和作用形式有所不同。
miRNA 是由具有发夹结构的初级转录本(pri-mRNA)经过一系列加工过程后生成
siRNA 是通过核酸内切酶对具有较好互补结构的长双链 RNA 前体进行加工形成的。
lncRNA(长非编码 RNA)是一类长度大于 200 个核苷酸并且不能翻译成蛋白质的转录本
部分 lncRNA 像 mRNA 一样具有 5’帽子和 polyA 尾巴,剪接之后形成成熟 RNA。
生物物理分析表明,lncRNA 可以折叠形成有功能的二级结构。有些 lncRNA 在不同的物种间相当保守,可能调节不同物种间共有的信号通路,使这些物种具有某些共同的生物学功能。此外,有些非保守的 lncRNA 的功能具有物种特异性,这可能受限与不同物种的环境选择压力和表型分离相关的进化。
由于 RNA 表达的时空特异性,导致传统的实验方法研究 RNA 的效率很低、成本较高,因此借助计算方法研究 RNA 是一个很好的补充。
在转录组水平上进行全转录组测序,从而开展可变剪接、编码序列单核苷酸多态性、基因表达情况等研究;
小分子 RNA 群体测序,通过分离特定大小的 RNA 分子进行测序,从而发现新的 miRNA 分子;通过去核糖体 RNA 并建立链特异性文库,进而鉴定新的 lncRNA 分子。
在转录组水平上,与染色质免疫共沉淀(ChIP)和甲基化 DNA 免疫共沉淀(MeDIP)技术相结合,从而检测与特定转录因子结合的 DNA 区域和基因组上的甲基化位点。
利用紫外交联免疫沉淀结合高通量测序(CLIP-seq),可以在全基因组水平揭示 RNA 分子与 RNA 结合蛋白的相互作用。
目前可以用 Illumina、BGI、Ion torrent 等二代测序平台进行小 RNA 测序。使用第二代测序技术对小 RNA 进行检测的基本步骤主要包括:
- 构建 DNA 模板文库,从总 RNA 中分离纯化出 20~30nt 的小 RNA 后,使用 T4 连接酶分别在 miRNA 的 5’端和 3’端连上接头序列,进行 RT-PCR 得到 70~80bp 的 DNA 片段;
- 将所得单链模板文库固定在平面或微球的表面;
- 通过桥式 PCR、微乳滴 PCR 或原位成簇对模板链进行扩增;
- 采集并记录 PCR 循环中的光学事件;
- 对产生的阵列图像进行时序分析,获得 DNA 片段的序列。
在植物中,小 RNA 以碱基互补配对的方式靶向 mRNA,导致 mRNA 的降解。为了大规模验证小 RNA 与 mRNA 的互作关系,常常会用到降解组测序(degradome sequencing)。降解组测序的原理是:在植物体内绝大多数的 miRNA 是利用剪接作用调控靶基因的表达,且剪接常发生在 miRNA 与 mRNA 互补区域的第 10 位核苷酸上。
参考
《植物基因组学》樊龙江
《生物信息学》樊龙江

