Kmer统计计算
在immunarch包中,要计算 kmer 出现的次数非常容易。我们可以直接使用getKmers()函数进行Kmer的统计计算:
kmers <- getKmers(immdata$data[[1]], 3)## Warning: `tbl_df()` is deprecated as of dplyr 1.0.0.## Please use `tibble::as_tibble()` instead.## This warning is displayed once every 8 hours.## Call `lifecycle::last_warnings()` to see where this warning was generated.kmers## # A tibble: 4,405 x 2## Kmer Count## <chr> <int>## 1 AAA 6## 2 AAD 5## 3 AAE 9## 4 AAF 2## 5 AAG 37## 6 AAI 2## 7 AAK 5## 8 AAL 4## 9 AAM 1## 10 AAN 13## # … with 4,395 more rows
同样的,我们还可以计算一批免疫组库中 kmer 的出现次数。为此,我们只需要向该函数提供一个免疫组库的列表。其中,NA 表示在样本中找不到这样的 kmer,具体由列名指定。
kmers <- getKmers ( immdata $ data , 5 )kmers## # A tibble: 172,926 x 13## Kmer `A2-i129` `A2-i131` `A2-i133` `A2-i132` `A4-i191` `A4-i192` MS1 MS2## <chr> <int> <int> <int> <int> <int> <int> <int> <int>## 1 AAAAG NA NA NA NA NA NA NA 1## 2 AAAAL NA NA NA NA NA NA NA NA## 3 AAAAW NA NA NA NA NA 1 NA NA## 4 AAADE NA NA NA NA NA NA NA NA## 5 AAADN NA 1 NA NA NA NA NA NA## 6 AAADT NA NA NA NA NA NA NA NA## 7 AAAEA NA NA NA NA NA NA NA NA## 8 AAAEN NA NA NA 1 NA NA NA NA## 9 AAAET NA NA NA NA NA NA NA NA## 10 AAAFE NA NA NA 1 NA NA NA NA## # … with 172,916 more rows, and 4 more variables: MS3 <int>, MS4 <int>,## # MS5 <int>, MS6 <int>
请注意,在默认情况下,getKmers()函数在计算 kmer 统计信息之前会过滤掉所有的非编码序列。您可以通过将.coding参数设置为 FALSE来同时使用编码和非编码序列:
kmers <- getKmers ( immdata $ data [[ 1 ]], 3 , .coding = F )kmers## # A tibble: 4,645 x 2## Kmer Count## <chr> <int>## 1 **G 1## 2 *~G 4## 3 *~L 1## 4 *AA 1## 5 *D~ 1## 6 *EE 1## 7 *G~ 1## 8 *GG 1## 9 *GP 1## 10 *HL 1## # … with 4,635 more rows
Kmer统计可视化
类似的,我们可以使用vis()函数来可视化kmer的统计信息:
kmers <- getKmers ( immdata $ data , 5 )vis ( kmers )
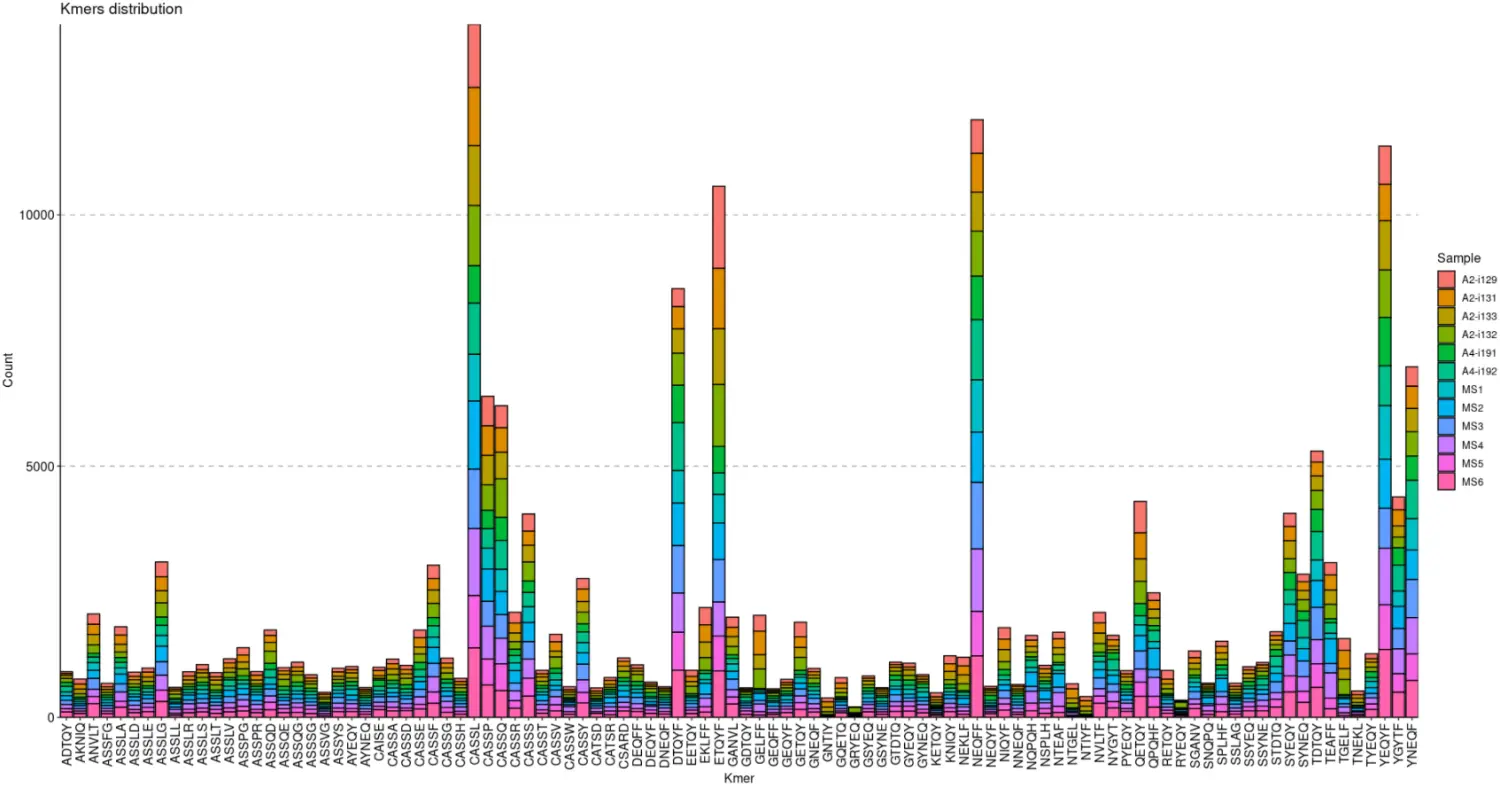
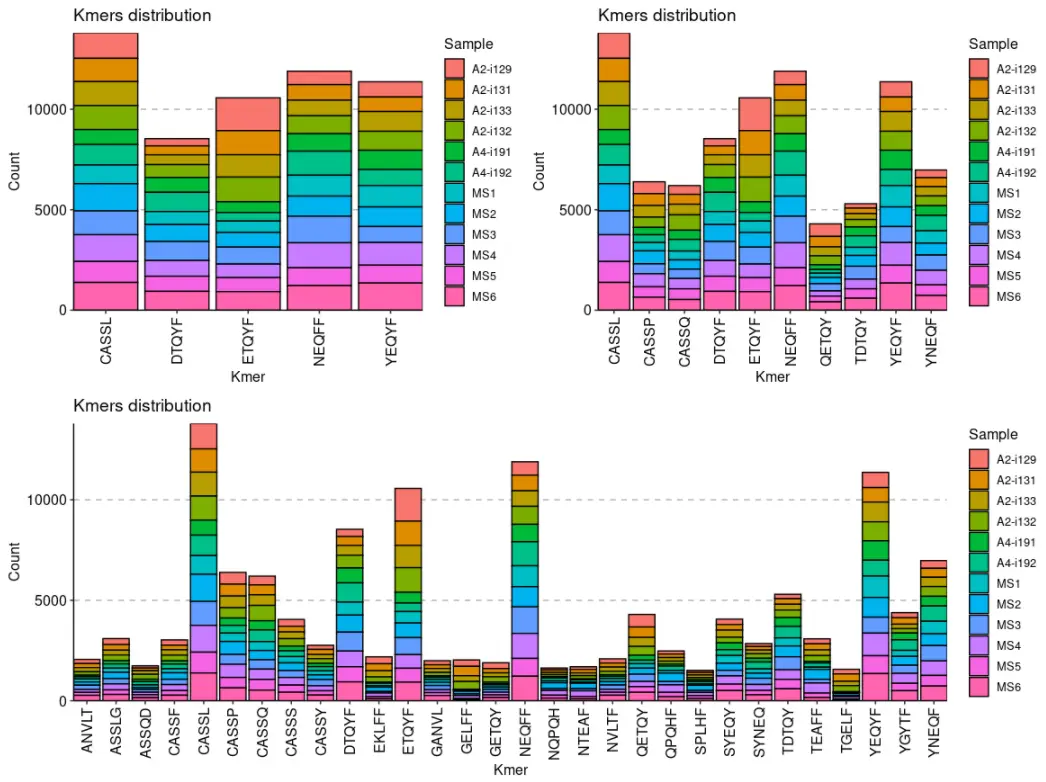
通过设置.head参数,可以指定要可视化的最丰富的 kmer 的数量。
p1 <- vis ( kmers , .head = 5 )p2 <- vis ( kmers , .head = 10 )p3 <- vis ( kmers , .head = 30 )( p1 + p2 ) / p3
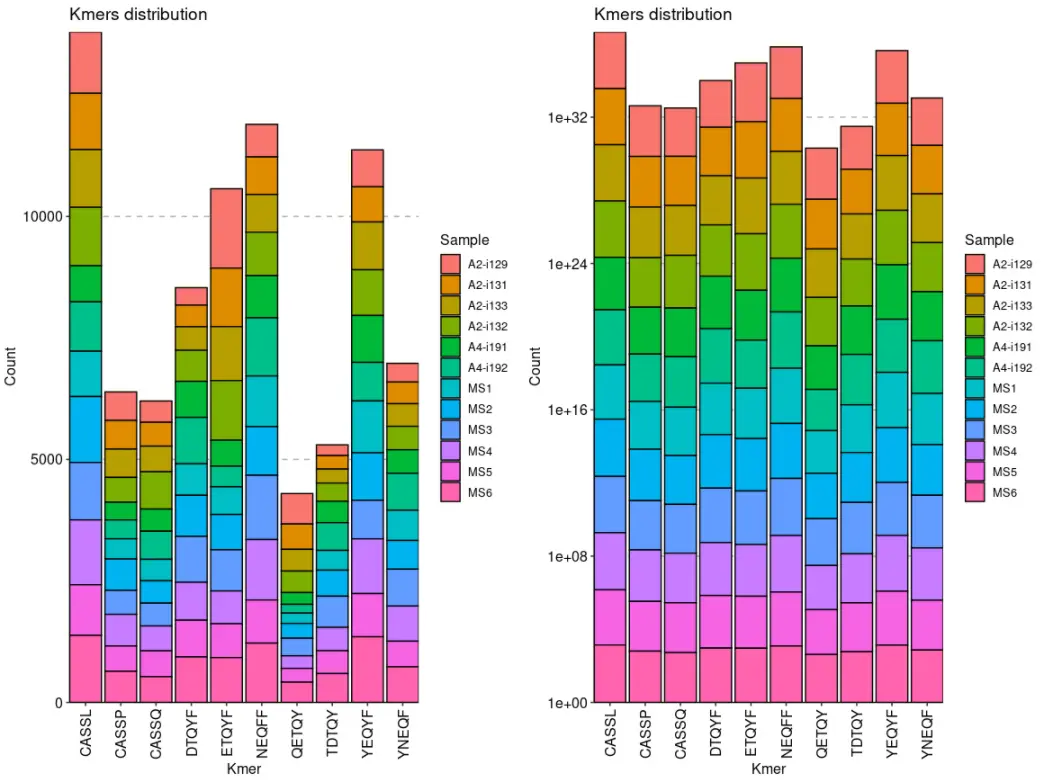
通过设置.position参数,我们可以更改调整柱的位置:“stack”, “dodge” and “fill”:
p1 <- vis ( kmers , .head = 10 , .position = "stack" )p2 <- vis ( kmers , .head = 10 , .position = "fill" )p3 <- vis ( kmers , .head = 10 , .position = "dodge" )( p1 + p2 ) / p3
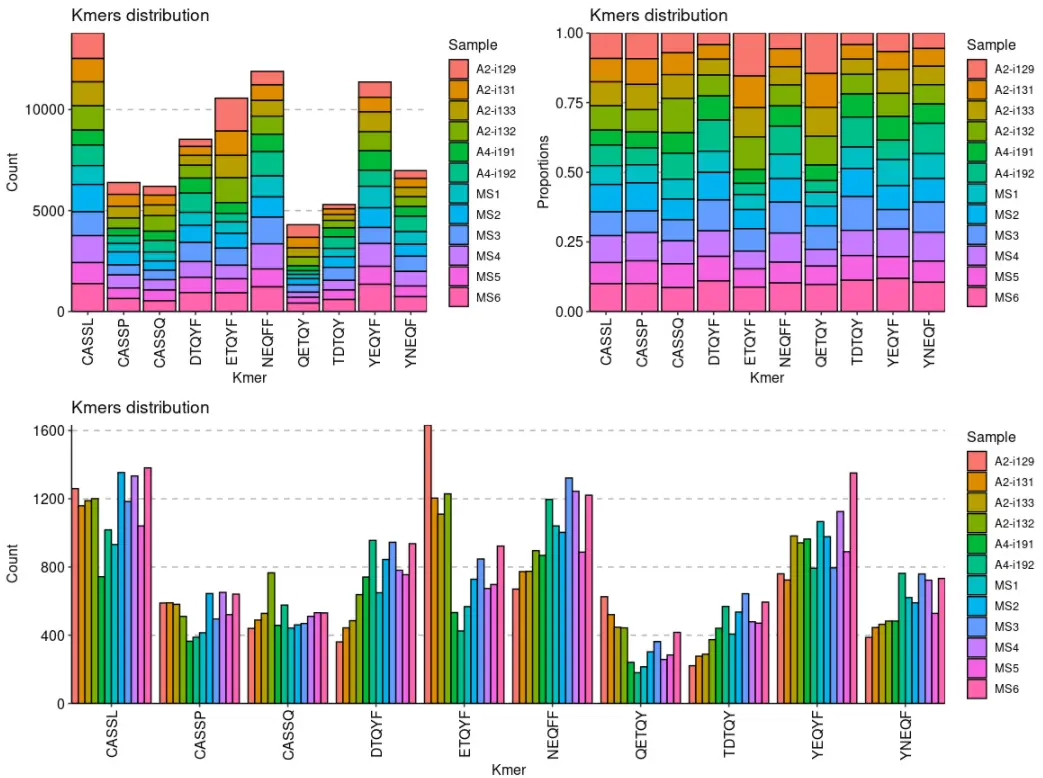
其中,选项“stack”将所有条形堆叠在一起,以便您可以查看 kmer 的完整分布。选项“fill”也将所有条形堆叠在一起,但同时对其进行标准化,以便您可以看到每个kmer的计数分布。选项“dodge”将不同样本的 kmer 条进行分组,以便您可以清楚地看到哪些样本总体上出现了更多的 kmer。
如果您的kmer计数的分布对于某些repertoire严重不平衡,则需要设置另外的参数.log。它允许使用y轴的对数变换,因此你可以在 kmer 计数中看到数量级的差异。
p1 <- vis ( kmers , .head = 10 , .position = "stack" )p2 <- vis ( kmers , .head = 10 , .position = "stack" , .log = T )p1 + p2
Motif分析
immunarch 使用常见的几种方法进行Motif序列基序分析,并使用不同类型的位置矩阵来表示Motif序列基序:
- 位置频率矩阵 (PFM) - 每个氨基酸在每个位置出现的频率的矩阵;
- 位置概率矩阵 (PPM) - 每个氨基酸在每个位置出现的概率的矩阵;
- 位置权重矩阵 (PWM) - 具有 PPM 元素对数似然的矩阵;
- 一个具有 PWM 中元素自信息的矩阵。
为了计算和可视化序列motif,首先,我们需要计算一个输入免疫组库的 kmer 统计数据,然后应用kmer_profile()函数来计算motif矩阵:
kmers <- getKmers ( immdata $ data [[ 1 ]], 5 )kmer_profile ( kmers )## [,1] [,2] [,3] [,4] [,5]## A 8955 8976 3791 3246 3159## C 6469 26 27 18 10## D 2129 2131 2131 2130 2085## E 3315 5868 5872 5870 5720## F 688 691 691 2600 9029## G 9194 9603 9603 9566 9326## H 405 405 405 680 659## I 750 1020 1107 896 852## K 721 1099 1100 1100 1044## L 3073 3078 4108 4097 4033## M 241 245 246 246 226## N 2421 2423 2424 2419 2355## P 2285 2564 2564 2559 2509## Q 2459 2466 7087 7086 7051## R 3487 3502 3504 3496 2917## S 14021 14029 13082 8128 3462## T 3408 5669 5671 6024 5797## V 1703 1927 1927 1561 1508## W 485 486 487 450 439## Y 2060 2061 2442 6097 6088## attr(,"class")## [1] "immunr_kmer_profile_pfm" "matrix"
目前我们不支持对多个样本进行motif序列基序分析,但我们正在努力。为了计算和可视化所有样本的序列基序矩阵,我们需要逐个处理它们,这可以在 for 循环中或通过lapply()函数轻松完成。
其中,参数.method指定要计算的矩阵:
.method = "freq"- 位置频率矩阵(PFM);.method = "prob"- 位置概率矩阵(PPM);.method = "wei"- 位置权重矩阵(PWM);.method = "self"- 自我信息矩阵。
kmer_profile ( kmers , "freq" )## [,1] [,2] [,3] [,4] [,5]## A 8955 8976 3791 3246 3159## C 6469 26 27 18 10## D 2129 2131 2131 2130 2085## E 3315 5868 5872 5870 5720## F 688 691 691 2600 9029## G 9194 9603 9603 9566 9326## H 405 405 405 680 659## I 750 1020 1107 896 852## K 721 1099 1100 1100 1044## L 3073 3078 4108 4097 4033## M 241 245 246 246 226## N 2421 2423 2424 2419 2355## P 2285 2564 2564 2559 2509## Q 2459 2466 7087 7086 7051## R 3487 3502 3504 3496 2917## S 14021 14029 13082 8128 3462## T 3408 5669 5671 6024 5797## V 1703 1927 1927 1561 1508## W 485 486 487 450 439## Y 2060 2061 2442 6097 6088## attr(,"class")## [1] "immunr_kmer_profile_pfm" "matrix"
kmer_profile ( kmers , "prob" )## [,1] [,2] [,3] [,4] [,5]## A 0.131172274 0.1314798811 0.0555303286 0.0475472030 0.0462728325## C 0.094757503 0.0003808464 0.0003954943 0.0002636629 0.0001464794## D 0.031185458 0.0312147534 0.0312147534 0.0312001055 0.0305409483## E 0.048557911 0.0859540934 0.0860126851 0.0859833892 0.0837861987## F 0.010077781 0.0101217244 0.0101217244 0.0380846358 0.1322562217## G 0.134673131 0.1406641375 0.1406641375 0.1401221638 0.1366066590## H 0.005932414 0.0059324144 0.0059324144 0.0099605970 0.0096529904## I 0.010985953 0.0149408956 0.0162152661 0.0131245514 0.0124800422## K 0.010561162 0.0160980826 0.0161127305 0.0161127305 0.0152924461## L 0.045013110 0.0450863496 0.0601737245 0.0600125972 0.0590751293## M 0.003530153 0.0035887445 0.0036033925 0.0036033925 0.0033104337## N 0.035462655 0.0354919510 0.0355065989 0.0354333592 0.0344958913## P 0.033470536 0.0375573101 0.0375573101 0.0374840704 0.0367516735## Q 0.036019277 0.0361218122 0.1038099284 0.1037952804 0.1032826026## R 0.051077356 0.0512970748 0.0513263707 0.0512091872 0.0427280318## S 0.205378722 0.2054959059 0.1916243097 0.1190584306 0.0507111573## T 0.049920169 0.0830391539 0.0830684498 0.0882391715 0.0849140899## V 0.024945436 0.0282265743 0.0282265743 0.0228654294 0.0220890888## W 0.007104249 0.0071188973 0.0071335452 0.0065915716 0.0064304443## Y 0.030174750 0.0301893978 0.0357702618 0.0893084709 0.0891766395## attr(,"class")## [1] "immunr_kmer_profile_ppm" "matrix"
kmer_profile ( kmers , "wei" )## [,1] [,2] [,3] [,4] [,5]## A 8.806550 8.8099289 7.5664346 7.3425192 7.303324## C 8.337399 0.3785116 0.4329594 -0.1520031 -1.000000## D 6.734032 6.7353868 6.7353868 6.7347096 6.703904## E 7.372865 8.1967251 8.1977082 8.1972167 8.159871## F 5.104337 5.1106138 5.1106138 7.0223678 8.818422## G 8.844549 8.9073414 8.9073414 8.9017720 8.865115## H 4.339850 4.3398500 4.3398500 5.0874628 5.042207## I 5.228819 5.6724253 5.7905114 5.4854268 5.412782## K 5.171927 5.7800476 5.7813597 5.7813597 5.705978## L 7.263504 7.2658494 7.6822924 7.6784241 7.655710## M 3.590961 3.6147098 3.6205864 3.6205864 3.498251## N 6.919459 6.9206506 6.9212459 6.9182670 6.879583## P 6.836050 7.0022525 7.0022525 6.9994363 6.970969## Q 6.941928 6.9460290 8.4690312 8.4688277 8.461684## R 7.445843 7.4520353 7.4528590 7.4495614 7.188342## S 9.453374 9.4541965 9.3533674 8.6667566 7.435462## T 7.412782 8.1469505 8.1474593 8.2345780 8.179163## V 6.411935 6.5902128 6.5902128 6.2863267 6.236493## W 4.599913 4.6028844 4.6058499 4.4918531 4.456149## Y 6.686501 6.6872007 6.9319194 8.2519557 8.249825## attr(,"class")## [1] "immunr_kmer_profile_pwm" "matrix"
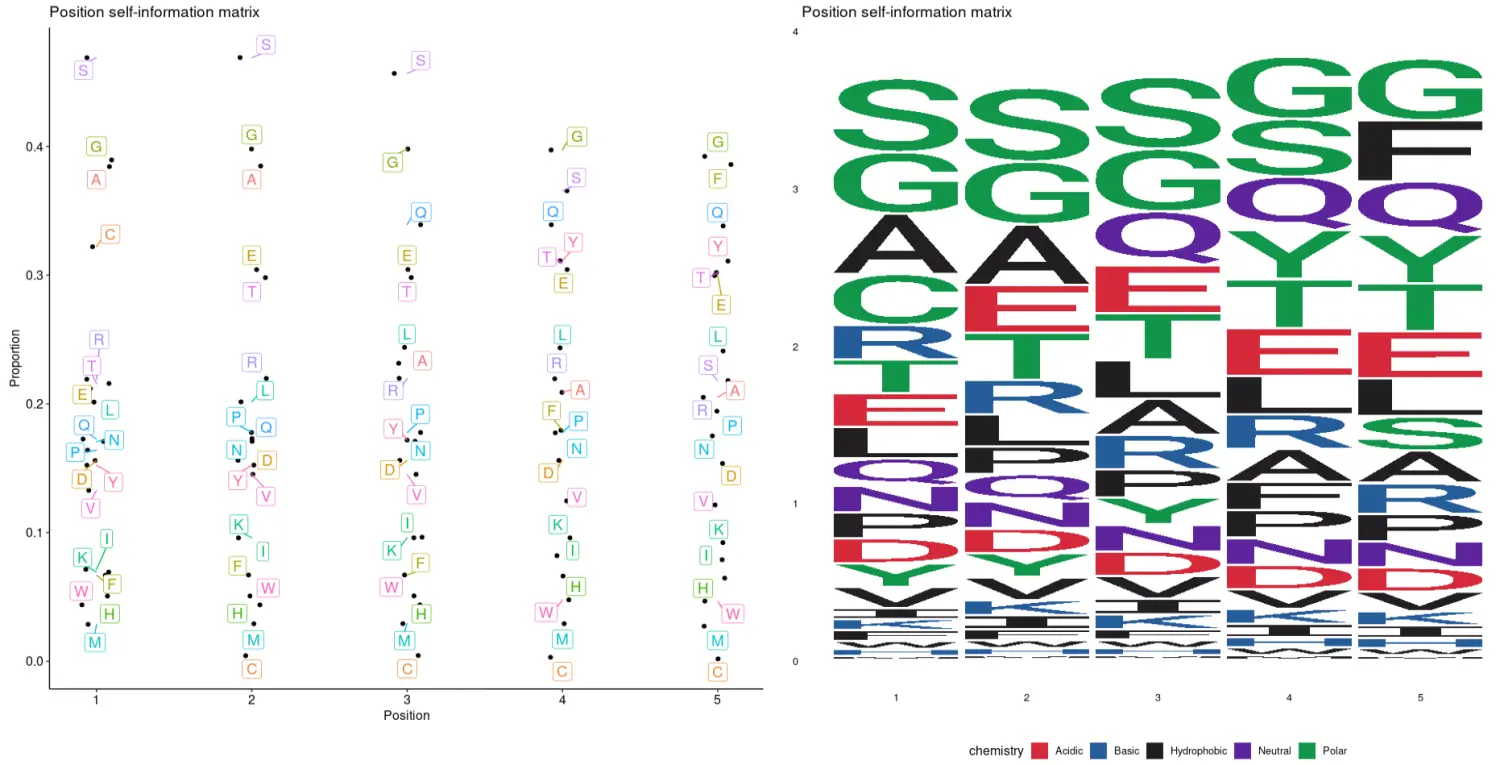
kmer_profile ( kmers , "self" )## [,1] [,2] [,3] [,4] [,5]## A 0.38439580 0.384852924 0.231593692 0.208945980 0.20515943## C 0.32213912 0.004325845 0.004470689 0.003134693 0.00186571## D 0.15602031 0.156124588 0.156124588 0.156072452 0.15371599## E 0.21191400 0.304302404 0.304425277 0.304363847 0.29971526## F 0.06684268 0.067070605 0.067070605 0.179555617 0.38600202## G 0.38953746 0.398033587 0.398033587 0.397280373 0.39232070## H 0.04388305 0.043883048 0.043883048 0.066233509 0.06462492## I 0.07149874 0.090610399 0.096424136 0.082049289 0.07892670## K 0.06933496 0.095895752 0.095961867 0.095961867 0.09222931## L 0.20136664 0.201588530 0.243987757 0.243566576 0.24110364## M 0.02875681 0.029148878 0.029246677 0.029246677 0.02727388## N 0.17084331 0.170942166 0.170991579 0.170744427 0.16756143## P 0.16403791 0.177824941 0.177824941 0.177583728 0.17516018## Q 0.17271556 0.173059094 0.339249150 0.339222412 0.33828469## R 0.21918174 0.219806921 0.219890176 0.219557010 0.19435586## S 0.46901135 0.469109844 0.456764807 0.365540136 0.21813673## T 0.21586646 0.298115914 0.298178816 0.309052134 0.30211178## V 0.13283645 0.145276593 0.145276593 0.124632326 0.12150152## W 0.05070375 0.050787142 0.050870488 0.047757003 0.04681920## Y 0.15239801 0.152450850 0.171879524 0.311245305 0.31097592## attr(,"class")## [1] "immunr_kmer_profile_self" "matrix"
Motif序列基序的可视化
同样的,我们可以使用vis()函数对计算出的序列基序矩阵进行可视化展示。有两种类型的图可供选择 - “sequence logo”和“text logo”。其中,参数.plot指定了绘图的类型:.plot = "seq"用于绘制“sequence logo”图和.plot = "text"(默认情况下)用于绘制“text logo”图。
kp <- kmer_profile ( kmers , "self" )p1 <- vis ( kp )p2 <- vis ( kp , .plot = "seq" )p1 + p2