Single cell RNA-seq analysis workshop
该系列课程由瑞典国家生物信息学基础设施(NBIS) 负责设置,为瑞典Phd学生,博士后,研究人员和瑞典学术界的其他工作人员开设的全国性研讨会。
本课程将通过演讲和实战练习涵盖了单细胞转录组(scRNA-seq)数据分析的基本步骤。涵盖的主题包括:
- An overview of the current scRNAseq technologies(当前常用scRNA-seq技术概述)
- Basic overview of pipelines for processing raw reads into expression values(原始测序数据比对转换为基因表达矩阵的分析流程概述)
- Quality control of scRNAseq data(scRNA-seq数据的质量控制)
- Dimensionality reduction and clustering techniques(数据降维和聚类分析技术)
- Data normalization(数据归一化处理)
- Differential gene expression for scRNAseq data(scRNA-seq数据的差异基因表达分析)
- Celltype prediction(细胞类型预测)
- Trajectory analysis(细胞轨迹拟时分析)
- Comparison of different analysis pipelines such as Seurat, Scran and Scanpy(不同分析工具流程的比较,如Seurat,Scran和Scanpy)
实战练习
在该实战练习中,我们使用了3种常用的scRNA-seq分析工具流程(Seurat,Scran和Scanpy),分别提供了不同分析工具的简短教程,我们可以任选自己熟悉的工具进行scRNA-seq数据分析。原则上,我们对这三种不同的分析工具执行相同的步骤,但是仍会存在一些细微的差异,因为并非在所有的分析工具中实现了所有不同的方法。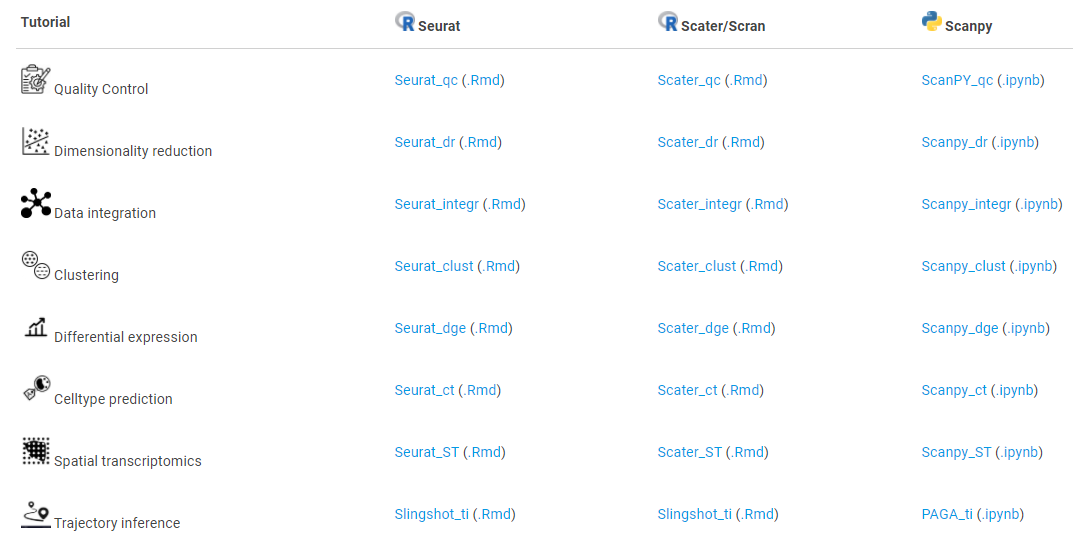
在本实战中,我选用了scRNA-seq数据分析中最常用的Seurat包进行练习演示。

