R studio 修改镜像
如果你是通过R studio 使用R,是可以直接通过偏好中的设置去修改镜像:
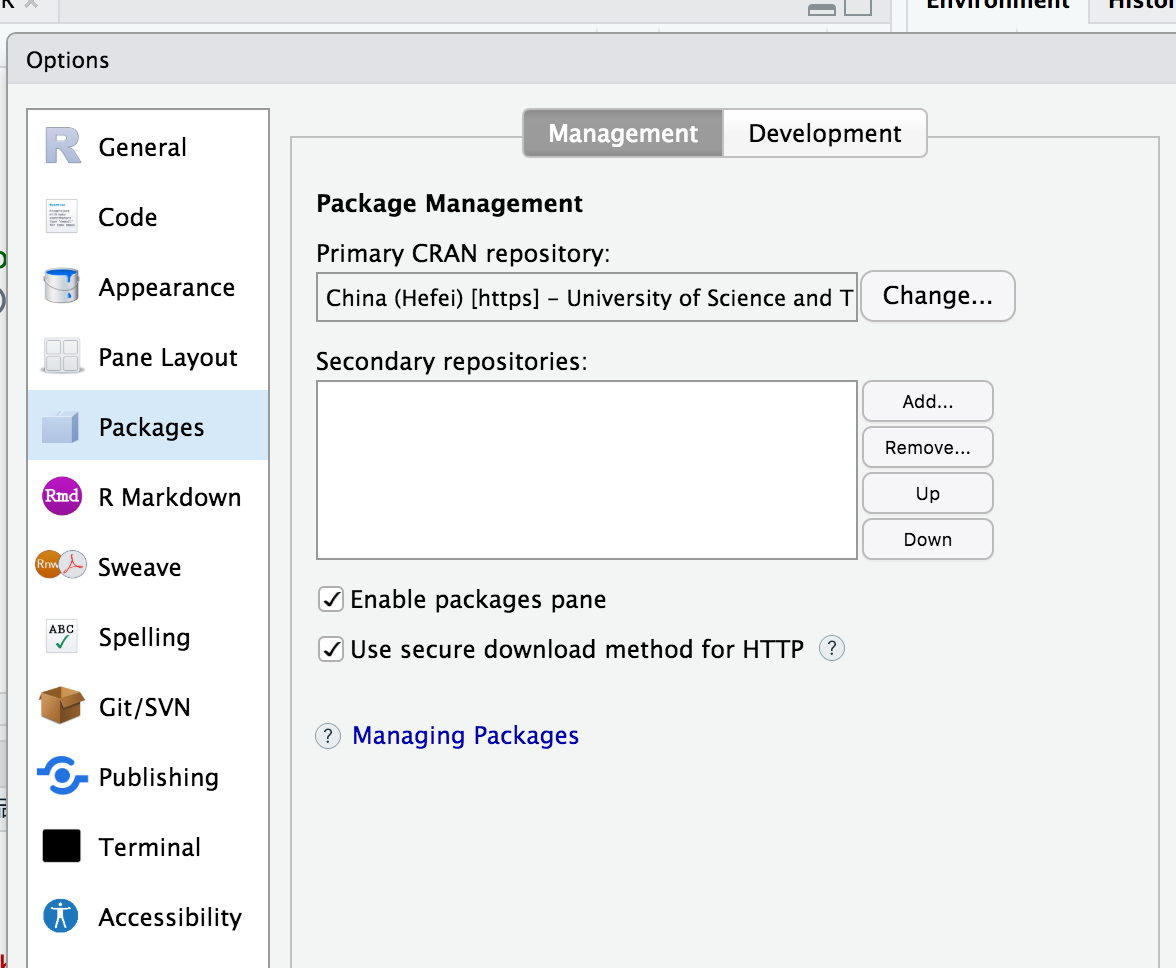
R 默认提供了非常多的镜像来源:

但有时候,我们可能需要通过biocmanager 安装包,就无法通过R studio 来修改了。
修改biocmanager 镜像
我们可以通过函数选择一下想要修改的镜像;
> chooseBioCmirror()Secure BioC mirrors1: 0-Bioconductor (World-wide) [https]2: Japan (Tachikawa) [https]3: Japan (Wako) [https]4: China (Peking) [https]5: Germany2 (Gottingen) [https]6: Germany2 (Gottingen)7: China2 (Nanjing) [https]8: China2 (Nanjing)9: (other mirrors)Selection:
选中需要的内容就可以直接修改了。
直接option 里修改
我们可以直接通过代码对options 中的镜像进行配置:
这里提供的是清华源的代码
local({r <- getOption( "repos" );# set CRAN mirror for users in Chinar[ "CRAN" ] <- "https://mirrors.tuna.tsinghua.edu.cn/CRAN/"; # CRAN的镜像地址options( repos = r )BioC <- getOption( "BioC_mirror" ); # set bioconductor mirror for users in ChinaBioC[ "BioC_mirror" ] <- "https://mirrors.ustc.edu.cn/bioc/"; # bioconductor的镜像地址options( BioC_mirror = BioC )})# 检查一下修改情况getOption("BioC_mirror")getOption("CRAN")
local 函数可以让我们的配置只在当前project 环境生效。
安装包技巧
判断式安装
我们可以利用requireNamespace 函数判断包是否安装在R 中,如果有则返回T,利用判断,看是否执行安装命令:
# 安装biocmanager(bioconductor)if (!requireNamespace("BiocManager", quietly = TRUE)) install.packages("BiocManager")# 安装devtools管理github上的软件包if (!requireNamespace("devtools", quietly = TRUE)) install.packages("devtools")
向量保存包名再安装
首先将需要的包保存在一个向量中:
bioPackages <-c("corrplot","ggrepel", #绘制相关性图形"stringr", #处理字符串的包"readr","tximport","dplyr", #处理salmon表达量的扩展包"FactoMineR","factoextra", #PCA分析软件"limma","edgeR","DESeq2", #差异分析的三个软件包"clusterProfiler", "org.Hs.eg.db", #安装进行GO和Kegg分析的扩展包"GSEABase","GSVA", #安装进行GSEA分析的扩展包"airway" # 包含数据集的bioconductor软件包
接着,通过apply 函数读取向量中的包名,并进行判断,如果在installed.packages() 不存在,则在available.packages() 的已知CRAN 的包中安装;如果不在CRAN 中,则从BiocManager 上下载:
lapply( bioPackages,function( bioPackage ){if(!bioPackage %in% rownames(installed.packages())){CRANpackages <- available.packages()if(bioPackage %in% rownames(CRANpackages)){install.packages( bioPackage)}else{BiocManager::install(bioPackage,suppressUpdates=F,ask=F)}}})
你可以在安装完成后通过library 验证一下是否安装成功,如果还没有,可能你的网络环境出了问题,或者这个包需要通过源文件安装,或是在github 上,需要使用devtools 安装。
你也可以不输出信息到屏幕进行加载:
suppressMessages(library(limma))

