数据的来源
- 一般有五种

R语言读取的格式

使用基础包(base)
也就是R 内置的数据读取与存储的函数。
数据读取一般两种情况:1)在R默认环境下。2)在其他目录下。
在默认工作目录下,直接在语句中输入文件名即可。
在其他目录下,则输入其所在位置的绝对路径。和终端指令不同的是,可以利用file.path() 使用”/“分隔每个目录。
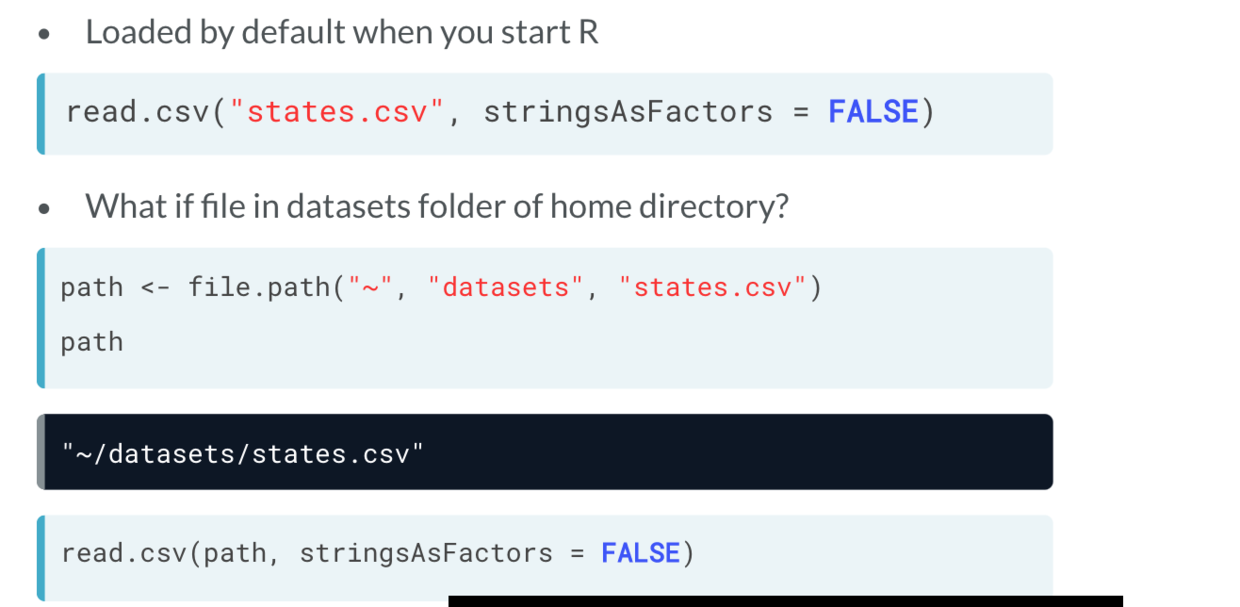
# Path to the hotdogs.txt file: pathpath <- file.path("data", "hotdogs.txt")
读取文件(flat data)
使用read.csv() 读取csv数据。
ps:可以通过 dir() 查看工作目录下的数据

# Import swimming_pools.csv: poolspools <- read.csv("swimming_pools.csv")
- 默认下,read.csv() 会将数据转化为factor 格式。
通过设定stringsAsFactors(默认TRUE)调整。
或者直接在全局设定options(stringsAsFactors = F)
# Import swimming_pools.csv correctly: poolspools <- read.csv("swimming_pools.csv", stringsAsFactors = FALSE)
相关参数
header = T #使第一行作为列名,默认Frow.names = 1 #使第一列作为行名comment.char = "!" #使! 开头内容不识别(注释掉),默认为#sep = "/" #指定分割符stringsAsFactors = F #默认T,F会使读取不识别字符串为因子。col.names = c("a","b","c") #指定读取的表格中的列colClasses = c("character","numeric","logical")) #指定读取的类型skip = 5 #读取表格略过第5行。n_max = 5 #选择前5行的内容。
其他参数
col_types = "clid" #和colClasses差不多,c 代表字符串,l代表布尔值,i代表整数,d代表浮点型。
不同类型的文件
read.csv() 读取csv文件,comma-separated value
read.table() 读取txt 文件。
read.delim() 也可以读取csv 文件。
一般来说read.table() 最为强大,而read.csv(), read.delim() 作为补充。
导出数据
可以很简单将df 数据框以exp.csv名导出到工作目录下。
write.csv(df,file="exp.csv")
txt 格式可以使用 write.table() 。
readr包
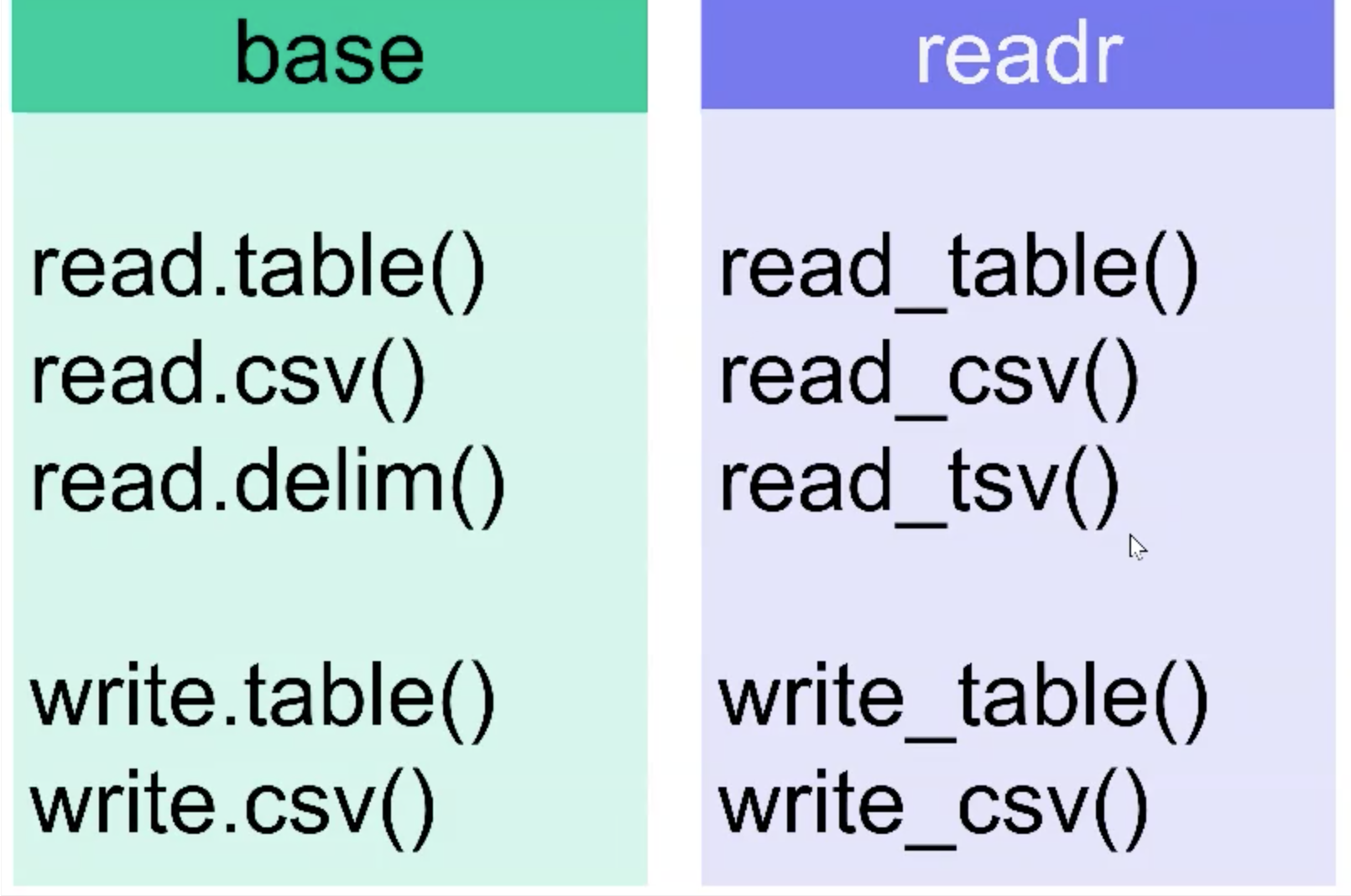
在基础包之上升级而来(便捷、智能)
使用data.table 包
data.table 使得导入table 类型文件更加方便。
fread函数
fread 是非常强大的读取table函数。
可以识别出如csv 文件是否文件第一行有列名称。

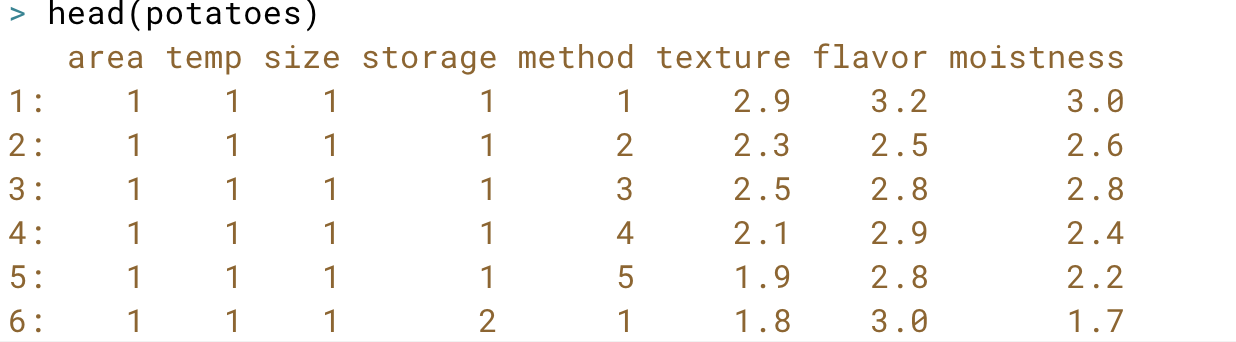
# load the data.table package using library()library(data.table)# Import potatoes.csv with fread(): potatoespotatoes <- fread("potatoes.csv")# Print out potatoespotatoes
两个参数:drop&select
fread 函数中有drop 与select 两个参数。可以对数据内容进行筛选。

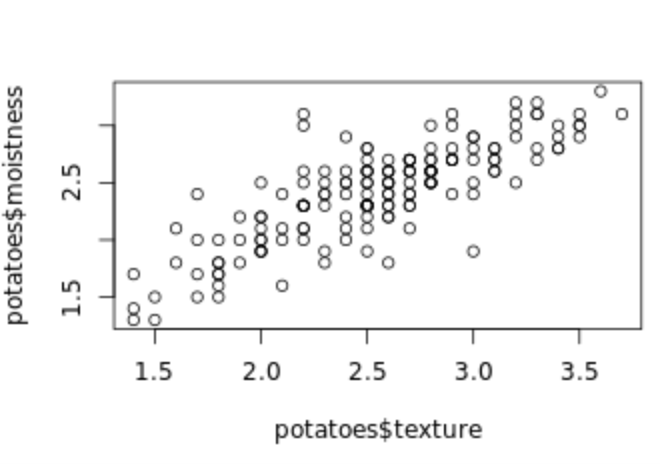
# fread is already loaded# Import columns 6 and 8 of potatoes.csv: potatoespotatoes <- fread("potatoes.csv", select = c(6,8))# Plot texture (x) and moistness (y) of potatoesplot(potatoes$texture, potatoes$moistness)
ps:plot 可以进行简单作图。
fread() 与 read.csv() 区别
主要区别在于,fread 的输出结果包括 data.table 与data.frame,而read.csv() 包括tbl_tf, tbl, 和data.frame。
rio包
主要用法:

直接将文件名作为参数,就会自动识别文件格式并进行相关的处理。
练习题
4-1
#1.读取ex1.txtex1 <- read.table("ex1.txt", header = T)#2.读取ex2_B cell receptor signaling pathway.csvread.csv("ex2_B cell receptor signaling pathway.csv")#3.读取GSE32575_series_matrix.txt,赋值给gse。gse <- read.table("GSE32575_series_matrix.txt", comment.char = '!',row.names = 1, header = T)#4.描述gse的属性str(gse)#5.将gse导出为新的txt和csv文件。write.csv(gse,"test1.csv")write.table(gse,"test2.txt")#6.将gse保存为Rdata并加载。save(gse,file="test3.Rdata")load("test3.Rdata")#练习4-1:#1.读取complete_set.txt(已保存在工作目录)set <- read.table("complete_set.txt",header = T)# 2.查看有多少行、多少列dim(set)# 3.获取行名和列名colnames(set);rownames(set)# 4.导出为csv格式write.csv(set,"test_set1.csv")# 5.保存为Rdatasave(set,file="test_set2.Rdata")# 6.加载class.Rdata,查看数据类型load("test_set2.Rdata");str(set)#高阶数据读取指南https://www.jianshu.com/p/4ea320c0dcc6

