COCOA package

COCOA全拼:Coordinate Covariation Analysis,中文译名:坐标协变分析
表观遗传学的关键挑战是确定个体之间表观遗传变异的生物学意义。研究人员提出了Coordinate Covariation Analysis (COCOA),这是一种使用跨个体的表观遗传信号的协变和区域集数据库来注释表观遗传异质性的计算框架。COCOA不但是用于DNA甲基化数据的工具,还可以分析具有基因组坐标的任何表观遗传信号。
COCOA是一种用参考数据注释样品间表观遗传变异的方法。可用于表观遗传数据,包括基因组坐标,如DNA甲基化和染色质可访问性数据。
GitHub:https://github.com/databio/COCOA
使用说明文档:http://code.databio.org/COCOA/articles/IntroToCOCOA.html
文章发表在:COCOA: coordinate covariation analysis of epigenetic heterogeneity,Genome Biology,07 September 2020
Basic workflow
- 输入文件要求
没仔细看完使用说明文档,但看到了输入必须是数量足够大的样本,那就是说这个软件比较适合用于大量的DNA甲基化芯片数据分析,对于相对昂贵的BS-seq来说可能不太适用,具体见说明文档。
一、COCOA包的安装
# To install from Bioconductor (recommended):if (!requireNamespace("BiocManager", quietly = TRUE))install.packages("BiocManager")BiocManager::install("COCOA")# COCOA may also be installed from Github:devtools::install_github("databio/COCOA")# or locally after downloading/cloning the source code:install.packages("path/to/COCOA/directory", repos=NULL, type="source")
二、分析流程
- Quantify inter-sample epigenetic variation
COCOA的第一步是量化表观遗传数据和目标变量之间的关联(即,用于监督分析的样本表型或用于非监督分析的主成分)。 此步骤的目标是获得每个表观遗传特征的分数,以表示其对目标变量的贡献程度。 我们将这些分数称为特征贡献分数。
- Annotate variation with region sets
COCOA的第二步是使用步骤1中的特征贡献分数来找出哪些参考区域集与目标变量的变化最相关。
- Permutation test
为了说明区域集的不同大小并评估结果的统计显着性,我们可以进行置换检验以估计p值。
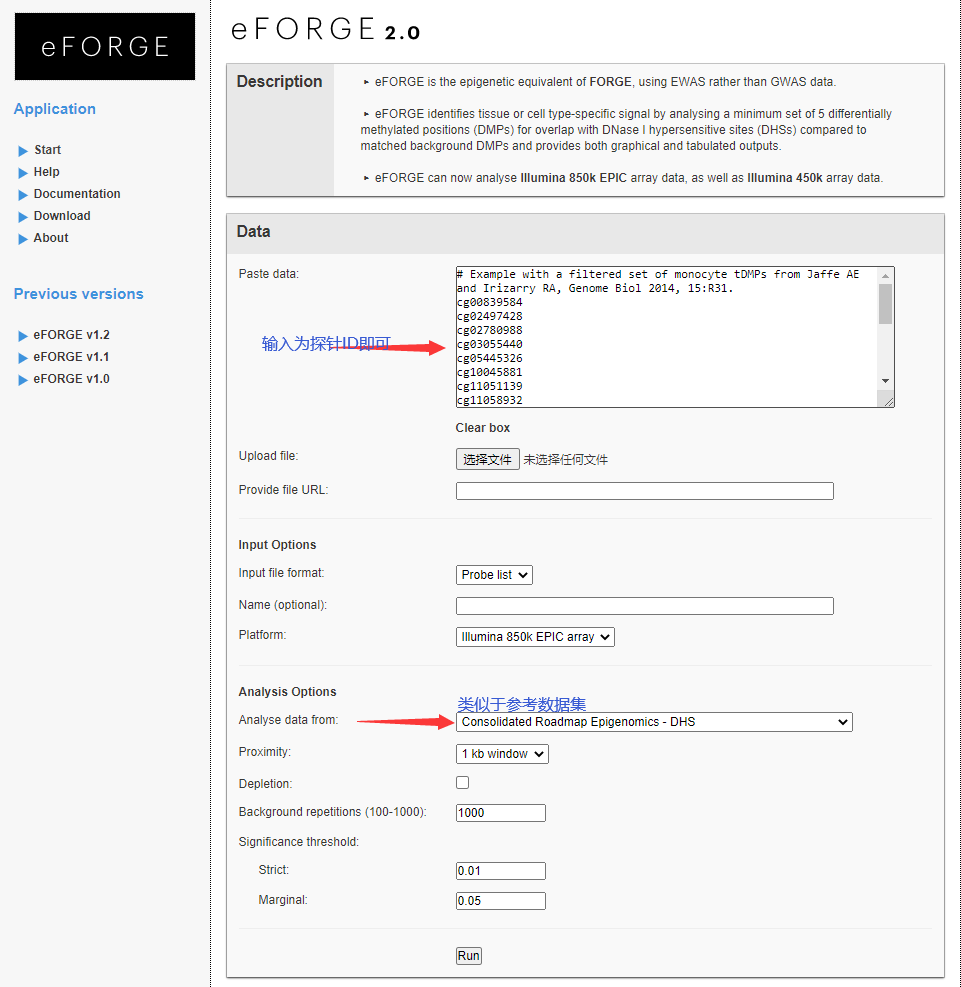
eFORGE
文章访问地址:eFORGE v2.0: updated analysis of cell type-specific signal in epigenomic data
A Tool for Identifying Cell Type-Specific Signal in Epigenomic Data,根据芯片探针来识别细胞类型
eFORGE2.0:https://eforge.altiusinstitute.org/