RRBSsim
标题:A comprehensive evaluation of alignment software for reduced representation bisulfite sequencing data
中文标题:用于减少代表性亚硫酸氢盐测序数据的比对软件的综合评估
DOI: 10.1093/bioinformatics/bty174
截止至2021.10.02,被八篇文章引用,但GitHub上已有两三年没有更新。
摘要:
动机: 下一代测序技术的快速发展为以单碱基分辨率研究全基因组 DNA 甲基化提供了机会。然而,未甲基化胞嘧啶的消耗给将亚硫酸氢盐转换的测序读数与大型参考比对带来了挑战。用于对齐甲基化读数的软件工具尚未得到全面评估,尤其是广泛使用的减少代表性亚硫酸氢盐测序 (RRBS),其中涉及 CpG 岛 (CGI) 的富集。
结果:
我们专门开发了一个模拟器 RRBSsim,用于对 RRBS 数据进行基准分析。我们在真实和模拟 RRBS 数据中对用于甲基化分析的七种比对算法进行了全面的比较。通过 RRBS 协议对 18 个肺肿瘤和匹配的邻近组织进行了测序。我们的实证评估发现,在 CGI 海岸或基因体上,低测序深度、中等甲基化水平的 CpG 位点的软件工具之间的甲基化结果不太一致。这些观察结果通过模拟得到进一步证实,模拟表明软件工具检测这些易受攻击的 CpG 位点的召回率通常较低,并且估计这些 CpG 位点中甲基化水平的精度较低。
在测试的软件工具中,bwa-meth 和 BS-Seeker2 (bowtie2) 目前是我们在召回率、精度和速度方面首选的 RRBS 数据对齐器。现有的对齐器不能有效地处理中等甲基化的 CpG 位点和 CGI 海岸或基因体上的那些 CpG 位点。对来自这些易受攻击的 CpG 位点的甲基化结果的解释应谨慎对待。
我们的研究揭示了甲基化数据固有的几个重要特征,RRBSsim 为推进基于序列的甲基化数据分析和方法学开发提供了指导。
可用性和实施: https://github.com/xwBio/RRBSsim 或 https://github.com/xwBio/Docker-RRBSsim 。
MethGET

DOI: 10.1186/s12864-020-6722-x
台湾学者开发的一个DNA甲基化与基因表达的关联分析的工具,发表的文章分数不太高,使用的人不多。
背景: DNA 甲基化是参与调控基因表达的主要表观遗传修饰。DNA 甲基化对基因表达的影响因基因组位置而异,并因界、物种和环境条件而异。为了确定 DNA 甲基化的功能调节作用,DNA 甲基化变化与基因表达改变之间的相关性至关重要。随着新一代测序技术的进步,全基因组甲基化和基因表达谱分析变得可行。当前用于调查这种相关性的生物信息学工具旨在评估 CG 位点的 DNA 甲基化。非CG甲基化与基因表达的相关性非常有限。一些生物信息学数据库允许相关性分析,
结果: 在这里,我们开发了一个生物信息学网络工具 MethGET(Methylation and Gene Expression Teller),专门用于分析全基因组 DNA 甲基化和基因表达之间的关联。MethGET 是第一个用户可以从任何基因组中提供他们自己的数据的网络工具。它也是基于全基因组亚硫酸氢盐测序数据将基因表达与 CG、CHG 和 CHH 甲基化相关联的工具。MethGET 不仅揭示了单个样本(单甲基化组)内的相关性,而且还对两组样本(多甲基化组)进行了比较。对于单甲基化组分析,MethGET 提供 Pearson 相关性和序数关联来研究 DNA 甲基化与基因表达之间的关系。它还对具有不同基因表达水平的基因进行分组,以查看特定基因组区域的甲基化分布。多甲基化组分析包括两组之间的比较分析和热图表示。这些功能可以详细研究 DNA 甲基化在基因调控中的作用。此外,我们将 MethGET 应用于水稻再生数据,发现基因体区域的 CHH 甲基化可能在组织培养过程中发挥作用,这证明了 MethGET 在表观基因组研究中的应用能力。
结论: MethGET 是一种 Python 软件,可关联 DNA 甲基化和基因表达。其网页界面可在 https://paoyang.ipmb.sinica.edu.tw/Software.html 公开获得。独立版本和源代码可在 GitHub 上获取,网址为 https://github.com/Jason-Teng/MethGET。
关键词: 生物信息学;相关性; DNA甲基化;表观基因组;基因表达; 二代测序;网络服务器。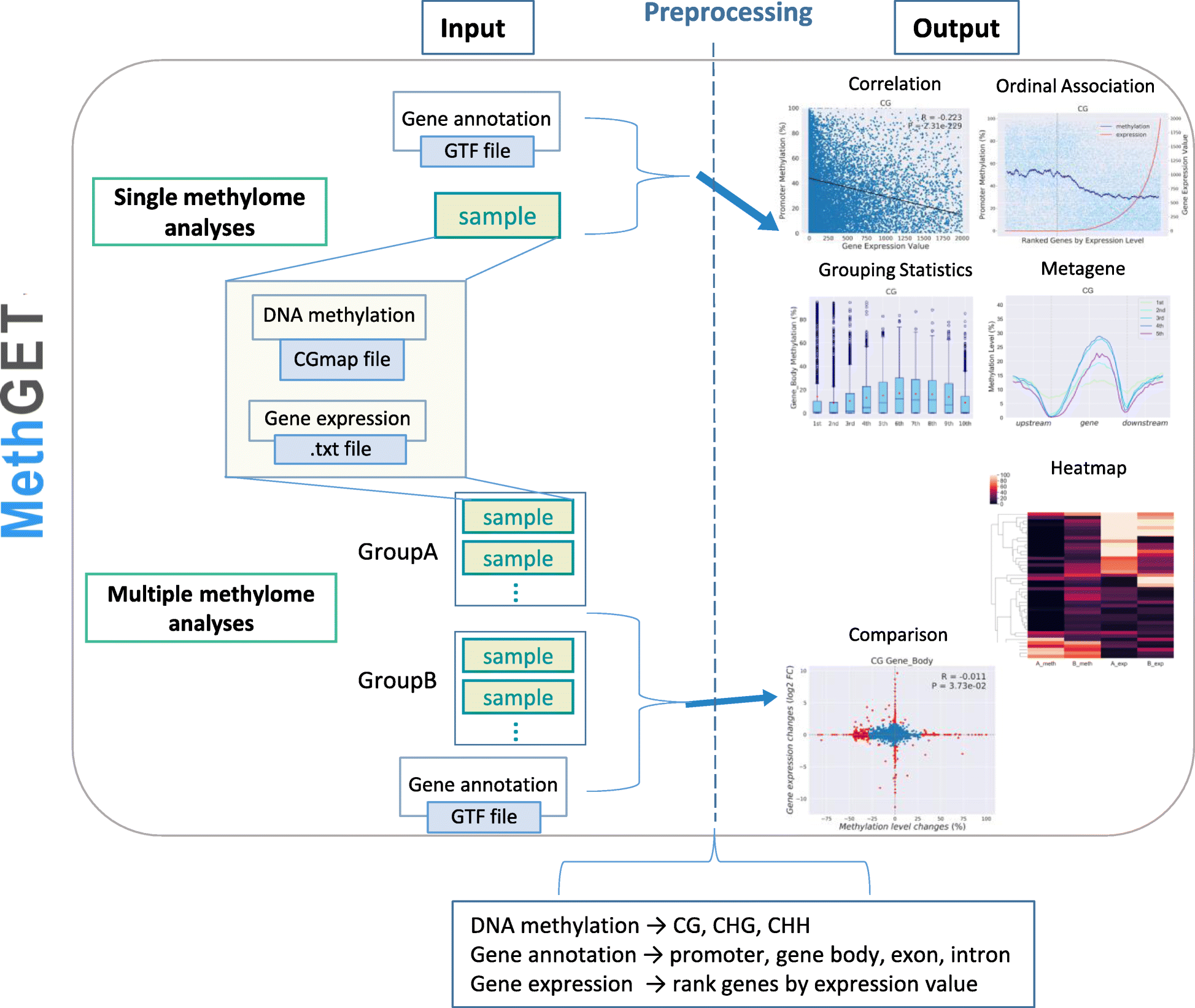
MethGET原理图。该图表显示了单甲基组分析和多甲基组分析的输入和输出

