中科院北京基因组研究生(国家生物信息中心):
https://bigd.big.ac.cn/
生物数据库汇总
相关数据库—重点
以下几个工作都是中国科学院北京基因组研究所国家基因组科学数据中心的成果:
MethBank
地址:https://bigd.big.ac.cn/methbank/
MethBank:a database of DNA methylomes across a variety of species,这个数据库主页包含很多自行开发的甲基化分析工具。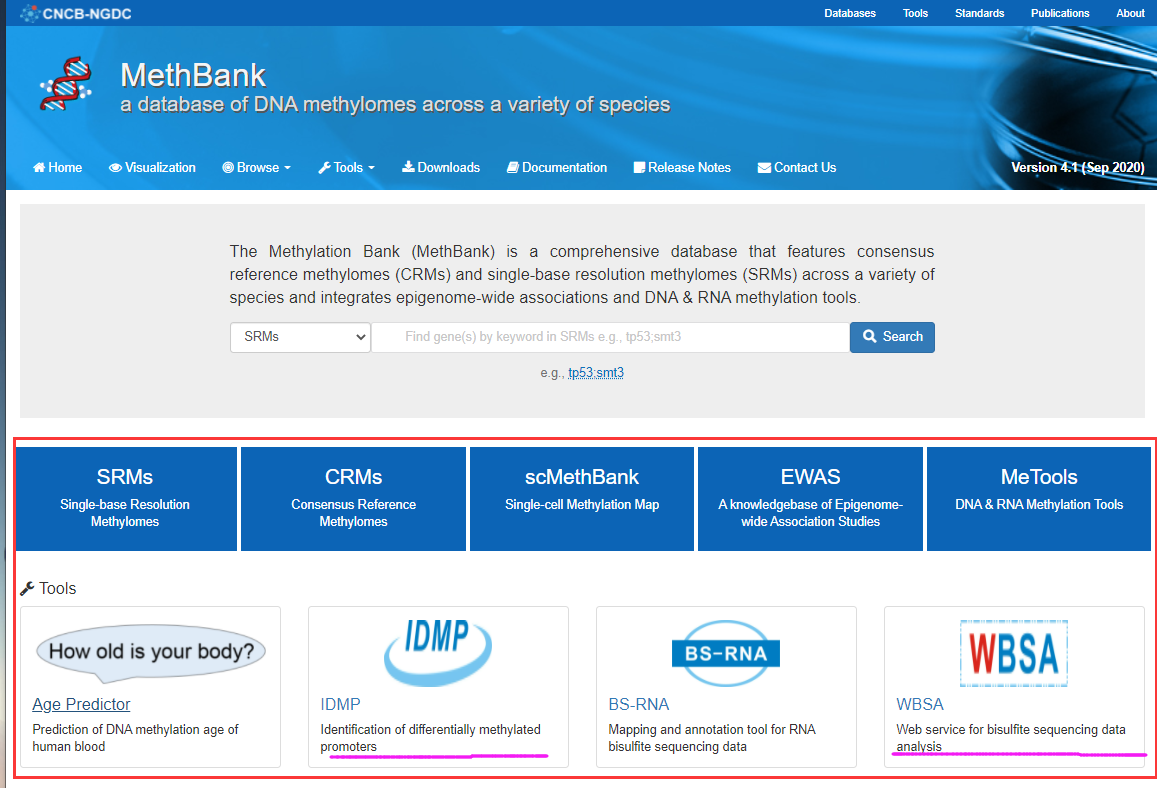
scMethBank
主页:https://ngdc.cncb.ac.cn/methbank/scm/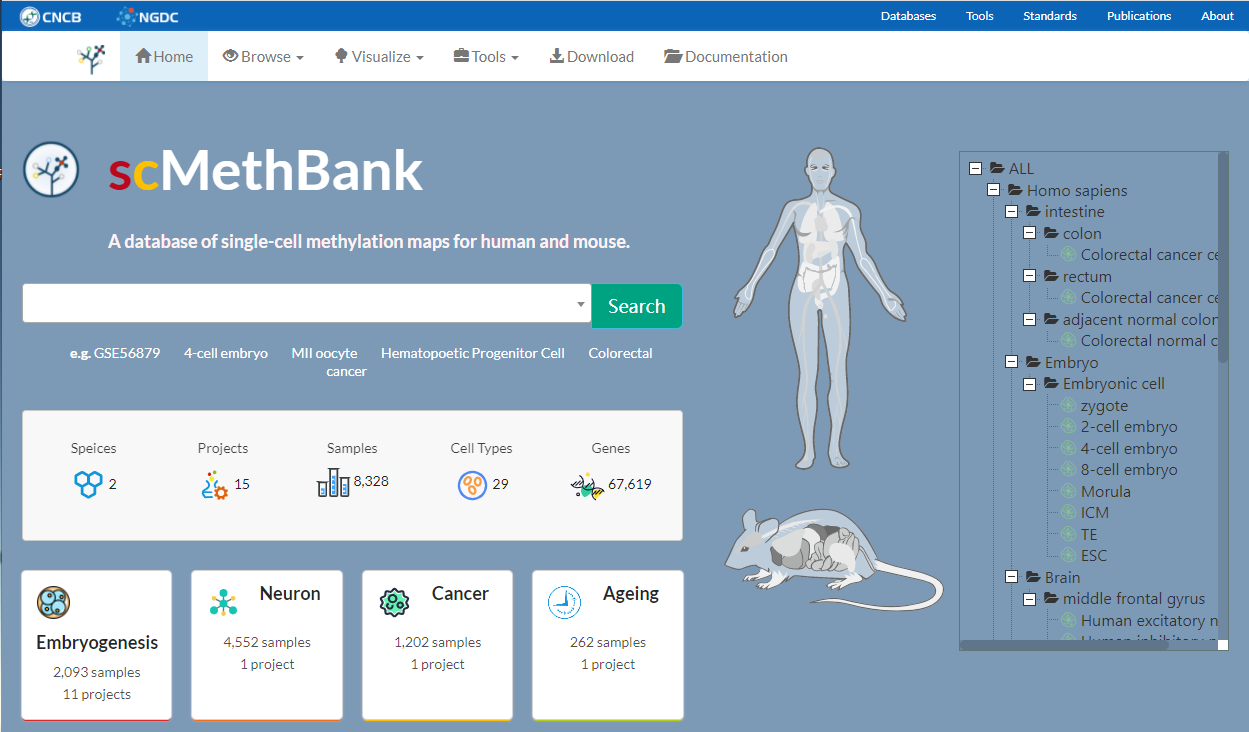
数据处理工作流程和 scMethBank 概述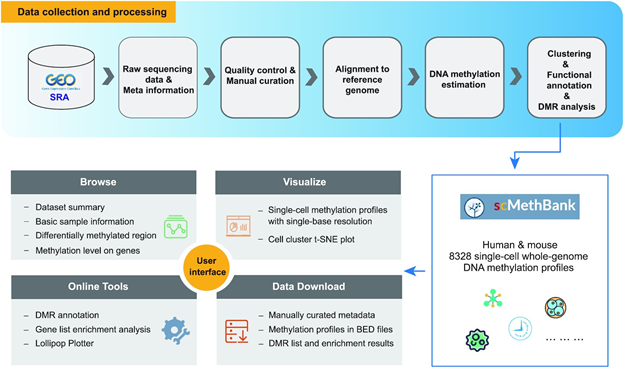
可视化的内容有:DMR,区域的甲基化水平可视化,单细胞的降维聚类等等;
支持用户上传DMR、gene list、bed文件 进行DMR注释、富集分析和绘制单位点分辨率的棒棒糖图。
建议阅读一下首页的document。
文章于2021.09.27发表在NAR上:scMethBank: a database for single-cell whole genome DNA methylation maps.
摘要:单细胞亚硫酸氢盐测序方法被广泛用于评估细胞状态的表观基因组异质性。在过去几年中,产生了大量数据并促进了对许多关键生物过程的表观遗传调控的更深入理解,包括早期胚胎发育、细胞分化和肿瘤进展。构建海量数据的功能性资源平台迫在眉睫。在这里,我们展示了 scMethBank,这是第一个开放访问的综合数据库,专门用于单细胞 DNA 甲基化数据和元数据的收集、整合、分析和可视化。scMethBank 的当前版本包括处理过的单细胞亚硫酸氢盐测序数据和来自 15 个公共单细胞数据集的 8328 个单细胞样本的精选元数据,涉及两个物种(人和小鼠)、29 种细胞类型和两种疾病。总之,scMethBank 旨在通过提供浏览、搜索、可视化、下载功能和用户友好的在线工具,帮助对细胞异质性感兴趣的研究人员在单细胞水平上探索和利用全基因组甲基化数据。
EWAS Data Hub
地址:https://bigd.big.ac.cn/ewas/datahub/index
EWAS Data Hub:A data hub of DNA methylation array data and metadata
主页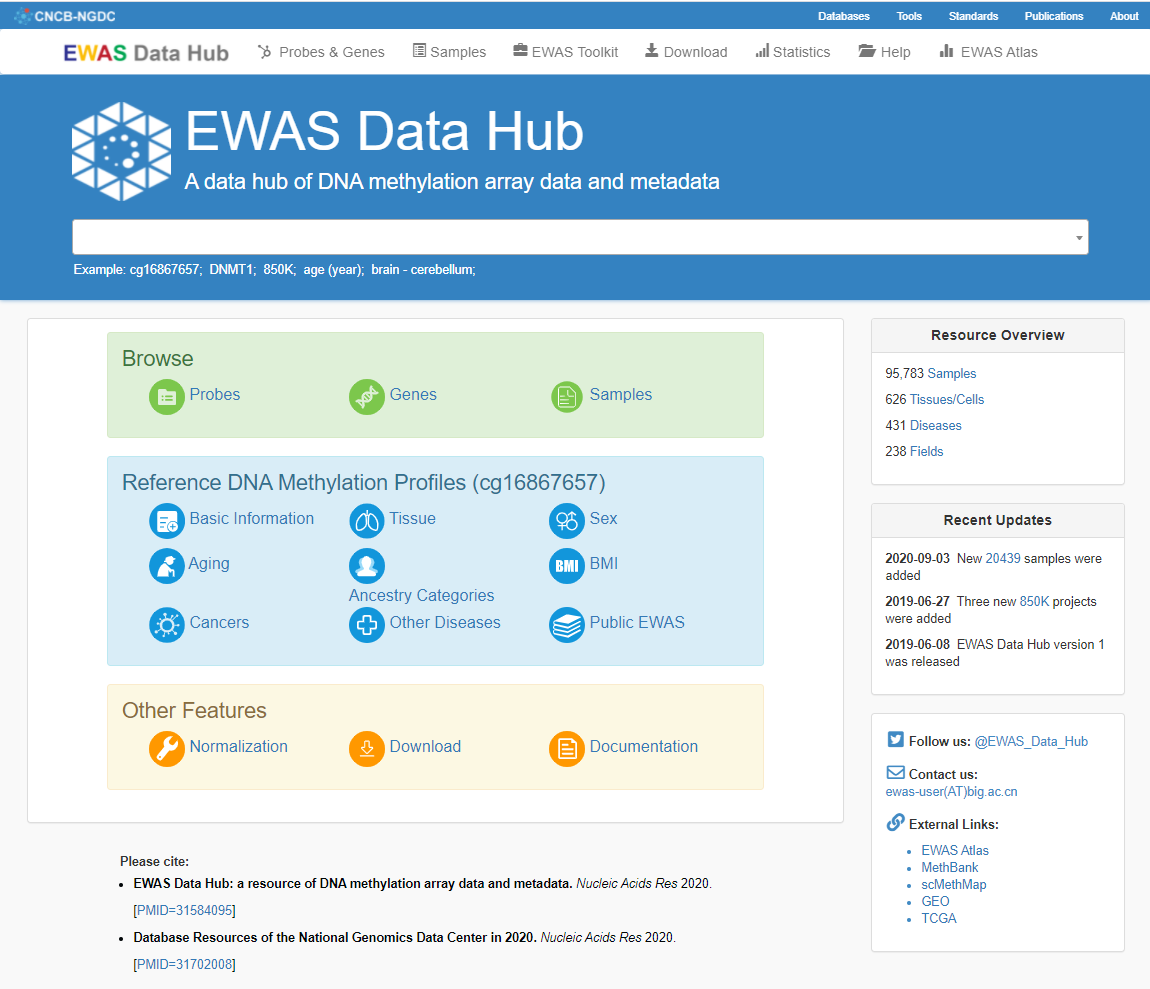
背景
表观基因组关联分析(Epigenome-wide Association Study,EWAS),即基于全基因组范围寻找组间的表观遗传学标记的差别或特征,目前是研究复杂疾病遗传学基础的一种有效方法。由于DNA样本的易获得性和DNA甲基化检测技术的不断成熟,大量的科研成果已经累积了大量的DNA甲基化芯片数据(主要是Infinium HumanMethylation450 (450K) 和 MethylationEPIC (850K) 芯片平台)。有效的数据整合和深入挖掘,将为复杂疾病或性状的表观遗传学深入研究提供更多重要的参考信息。但是,由于不同试验的研究平台,研究批次,包括样本的年龄性别等信息千差万别,直接使用这些数据肯定会导致大量的假阳性。为了解决这些问题,实现更好的甲基化数据整合分析,中国科学院北京基因组研究所国家基因组科学数据中心,开发了人类表观组关联分析数据库EWAS Data Hub。该研究成果于2019年10月在国际学术期刊《核酸研究》(Nucleic Acids Research)在线发表,标题为“EWAS Data Hub: a resource of DNA methylation array data and metadata”。
数据库内容
EWAS Data Hub整合了来自GEO、TCGA、ArrayExpress和ENCODE数据库的共计75,344个样本的DNA甲基化芯片数据(450K和850K芯片数据)和相应的元数据,涉及470种组织/细胞类型,306种疾病和178种样本相关变量。数据整合处理时采用了归一化方法来消除不同数据集的批次效应,为485,512个探针和36,397个基因提供了参考DNA甲基化图谱。
优点:数据来源多,样本多样
缺点:目前所有数据来源均为DNA甲基化芯片数据(主要是Infinium HumanMethylation450 (450K) 和 MethylationEPIC (850K) 芯片平台)。
使用示例
搜索某个基因:TCL1A
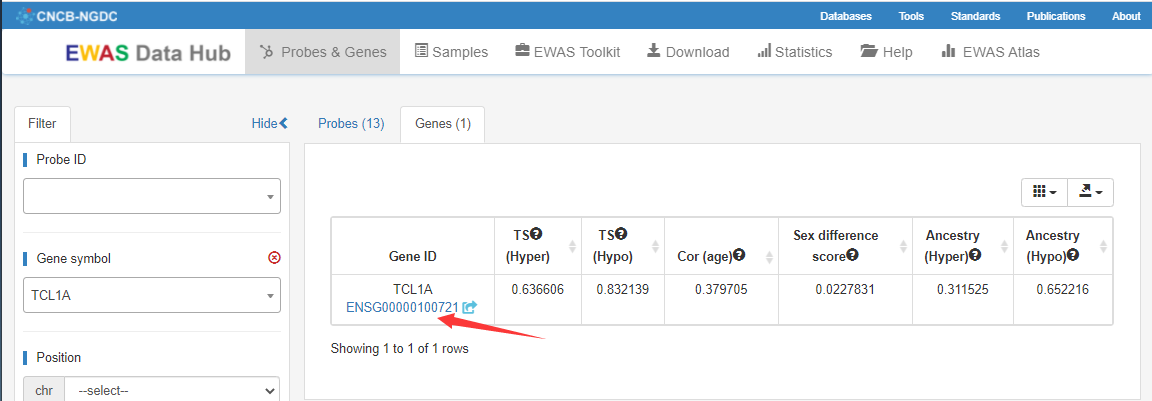
基础信息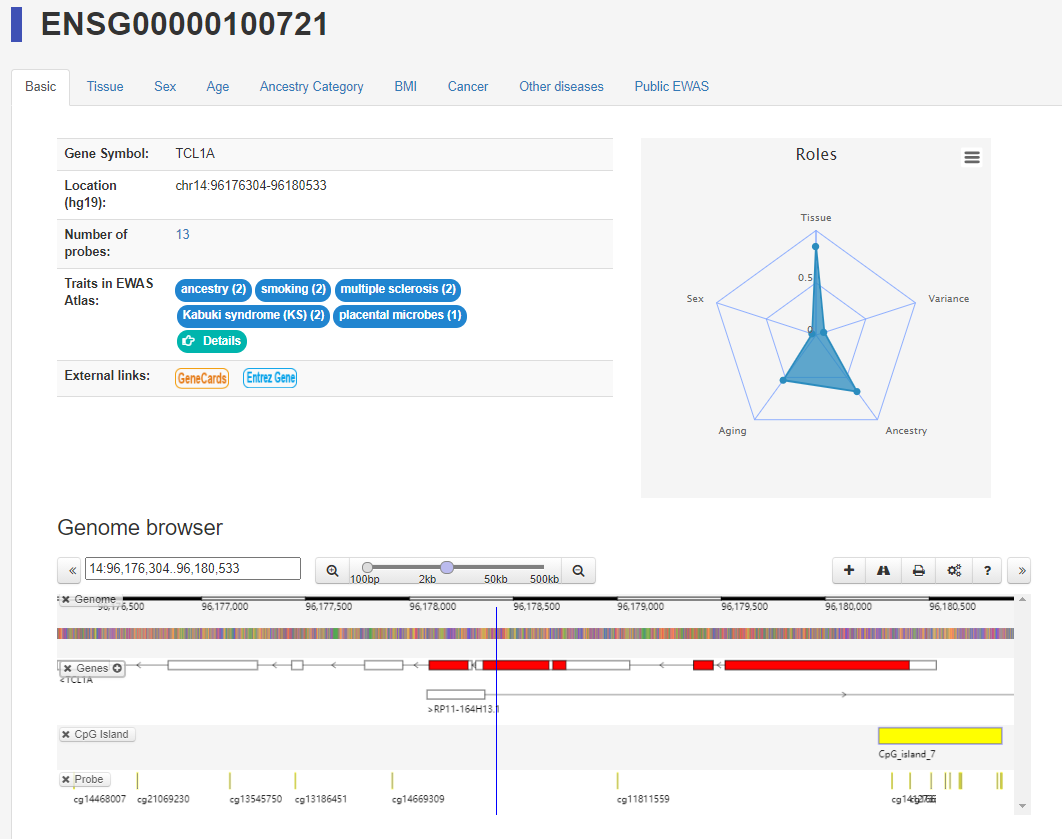
在组织/细胞类型的DNA甲基化情况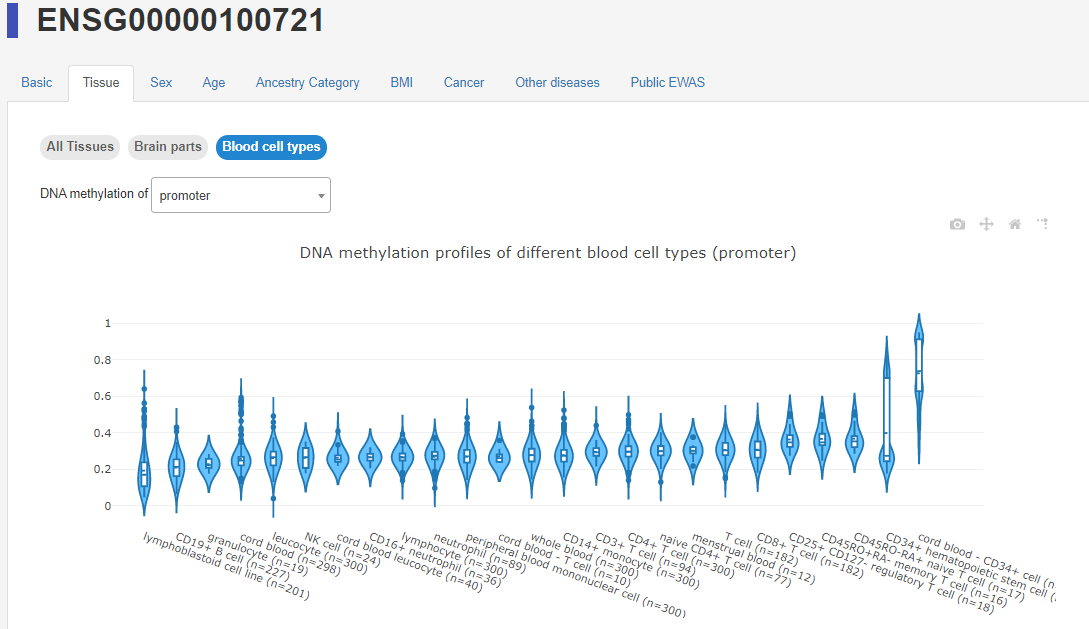
在癌症中的DNA甲基化情况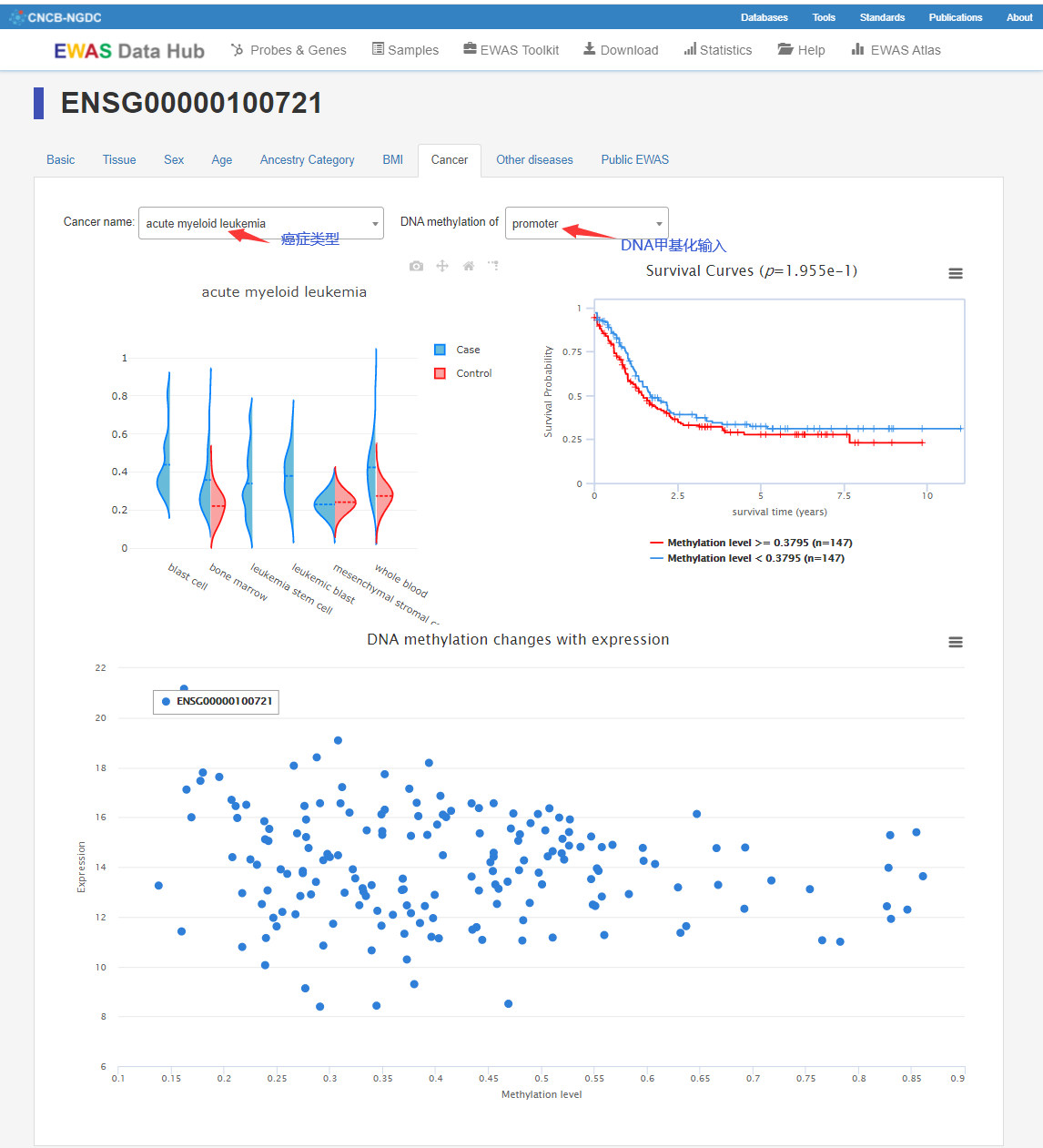
EWAS Atlas
地址:https://bigd.big.ac.cn/ewas/index
EWAS Atlas:A knowledgebase of epigenome-wide association studies
EWAS Open Platform
EWAS Open Platform:表观基因组关联研究资源开放平台
通过不断整合和更新中心已有EWAS资源,EWAS Open Platform提供从数据浏览与下载、在线分析与可视化到知识解释与验证的全面系统的资源和服务。
EWAS Open Platform包括:
- 标准化的数据信息库 (EWAS Data Hub);
- 人工信息提取的知识库(EWAS Atlas);
- 表观-特征关联在线工具(EWAS Toolkit)
MethHC
大家普遍反映该数据库经常登录不上去,提交请求后也会出现报错,嗯。。。看着用吧,所以有人推荐使用DiseaseMeth这个数据库作为查询肿瘤与甲基化关系的替代品。
1.0 版本
https://ngdc.cncb.ac.cn/databasecommons/database/id/120
Full name: A database of DNA Methylation and gene expression in Human Cancer
Description: The human pan-cancer methylation database, MethHC is a web-based resource focused on the DNA methylation of human diseases. MethHC integrated data such as DNA methylation, gene expression, microRNA methylation, microRNA expression, and the correlation of methylation and gene expression from TCGA (The Cancer Genome Atlas)
2.0版本
地址:https://awi.cuhk.edu.cn/~MethHC/methhc_2020/php/index.php
文章标题:MethHC 2.0: information repository of DNA methylation and gene expression in human cancer
文章地址:https://academic.oup.com/nar/article/49/D1/D1268/6018433
Tools
Age Predictor
Age Predictor
Age predictor is a online tool to calculate DNA methylation age of human blood. It has high accuracy (average absolute difference between predicted age and chronological age is around 3 years) and supports various types of data (raw data, processed data, GEO sample ID). Although the first version can only predict human blood DNA methylation age, it will support more tissues and srmSpecies in the future.
IDMP
IDMP
IDMP is a tool to identify the differently methylated promoters between two samples. Fisher exact test and false discovery rate (FDR) correction are used in IDMP. The IDMP software package is freely available from our ftp site: ftp://download.big.ac.cn/methbank/Tool
BS-RNA
BS-RNA
BS-RNA is an efficient, specialized and highly automated mapping as well as annotation tool for RNA bisulfite sequencing data. The annotation result of BS-RNA is exported in BED (.bed) format, including locations, sequence context types (CG/CHG/CHH, H = A, T, or C), reference sequencing depths, cytosine sequencing depths, and methylation levels of covered cytosine sites on both Watson and Crick strands. It is freely available from the website: http://bs-rna.big.ac.cn
WBSA
WBSA
WBSA (Web Service for Bisulfite Sequencing Data Analysis) is a convenient, stable, and efficient web service to analyze bisulfate sequencing data. WBSA comprises three main modules: WGBS data analysis, RRBS data analysis, and differentially methylated region (DMR) identification. The WGBS and RRBS modules execute read mapping, methylation site identification, annotation, and advanced analysis, whereas the DMR module identifies actual DMRs and annotates their correlations to genes. It could be accessed via http://wbsa.big.ac.cn

