前不久,我们实验室历时八年开发的全基因组 DNA 甲基化检测新方法 GPS 得以发表,揭示了很多我们不曾设想的规律。开发全新的技术困难重重,个中辛酸难以言表。借着文章发表的契机,我们系统回顾了文章从想法到现实的历程,以及 GPS 所揭示了重要规律,希望为大家提供一些借鉴,并鼓励前行者不要气馁。朝着正确的方向,终将柳暗花明。
全基因组 DNA 甲基化检测说起来容易做起来难
随着人类基因组计划的完成,生命科学研究进入了 “后基因组时代”,而表观遗传学是“后基因组时代” 的重要方向。DNA 甲基化是表观遗传学的核心组成部分,对于正常细胞功能维持、胚胎发育等生命过程至关重要,DNA 甲基化堪称人类基因组的另外一套密码。
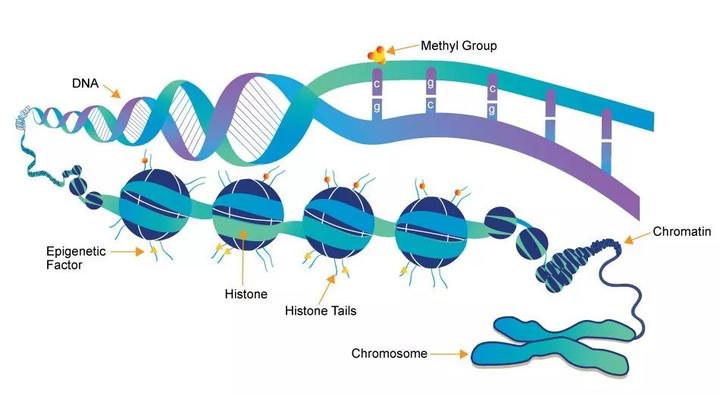
对一个人的所有细胞来说,每一个细胞的 DNA 几乎一样,但每一种组织细胞的表观基因组却差异巨大,而且随着时间的变化而不同,典型的例子就是通过检测外周血中特定位置的 DNA 甲基化竟然可用来预测一个人年龄。异常 DNA 甲基化已经被证明与人类许多疾病尤其是肿瘤的发生发展具有密切关系。因此,进行全基因组 DNA 甲基化的精准检测和分析无疑对探索肿瘤的发生发展和转移以及开发新的抗肿瘤策略具有重要意义。然而全基因组 DNA 甲基化测序可以说是表观遗传领域公认的 “土豪” 游戏,必须“不差钱”。2008 年美国 NIH 启动了“表观基因组的路线图计划(RoadMap Epigenome Project)”,计划用 5 年的时间绘制 50 种人类正常组织的表观基因组图谱,而预算的经费居然高达 6.6 亿美元。你没有看错,是 6.6 亿美元,平均一种组织细胞的花费大约为 1320 万美元,如果按当时汇率折合人民币接近 1 亿人民币。我们再看看欧洲的“千人表观基因组计划(1000 Epigenome Project)”,拟用 10 年时间花费 3900 万欧元完成 1000 人的表观基因组图谱,这个花费也挺吓人的。
为什么会花费这么多钱,因为构成生物体基因组仅仅由 A、T、C、G 四个碱基构成,当 DNA 被亚硫酸盐处理后,没有甲基化的 C 转变成了 T,构成 DNA 的序列就剩下了 A、T、G 三个碱基,其复杂度大幅降低,当测序完成后,你就会发现大部分序列没法与人类参考基因组比对(Map),通俗的讲就是 “找不到家门”,DNA 甲基化计算无从谈起,而确定重复序列区域的 DNA 甲基化状态更是全基因组 DNA 甲基化检测者的噩梦。最早的全基因组 DNA 甲基化检测的 Map 率只有 29%,也就是说你花费了 100 元钱,只有 29 元钱是有用的,那 71 元钱测到的序列因为“无家可归” 而被白白扔掉了。与此同时,人们也意识到那 71 元钱检测的序列其实是细胞内真实存在的,只是因为技术的限制被人为地忽略掉了。而这种检测最大的风险也可能是捡了芝麻丢了西瓜,因为那 71% 的真实信息本来也很重要,而你只能用 29% 的 DNA 甲基化信息得出结论,所得结论的有没有偏差,相信诸位心里都有判断。
但科学的进步是循序渐进的,丝毫不妨碍发高水平的文章,2012 年通过检测大猩猩和人类各 4 份精子样本的全基因组 DNA 甲基化也一样可以登顶 Cell 杂志。可想而知,全基因组 DNA 甲基化检测是多少人的梦想,同时你也会觉察到,有钱就可以任性。所以,开发出人人可以负担的全基因组 DNA 甲基化检测方法就显得尤为重要,使全基因组 DNA 甲基化检测真正从 “旧时王谢堂前燕”,进而“飞入寻常百姓家” 是许多表观人的奋斗目标。
GPS 因 “运” 而生
2009 年底,我全职回到复旦大学,组建了自己的实验室,鉴于国际上表观遗传学的快速发展,根据已有的研究结果设计自己的课题并不适合我们,追赶别人其实是一件很累的活儿。所以,我对实验室的研究方向定了一个基本的原则:别人已经做的,我们不做;别人能做的,我们也基本不做。我们要么就做别人不做的,比如我们的 NamiRNA 激活基因研究;要么就做别人做不好的,比如全基因组 DNA 甲基化检测。 我们希望让全基因组 DNA 甲基化不仅精准,而且费用足够低,让人人可以做。
GPS 方法开发纯属偶然。有一天,我在寻找线粒体 DNA 的甲基化信息,发现当时居然没有任何信息可用。尽管我从 2001 年起,在国外一直在从事 DNA 甲基化研究,要知道线粒体 DNA 仅有 16kb 左右,而当时,我们已经进入二代测序时代,如果连一个 16kb 的 DNA 都没有甲基化信息,那并不是说线粒体 DNA 甲基化不重要,而说明全基因组 DNA 甲基化检测一定存在什么问题,而这些问题无疑会阻碍该领域的发展。合理质疑是解决问题的前提,而要解决一个问题,首先要明白导致这个问题的根本原因。好在已经有文献告诉我们问题的关键是什么,在 Nature Method 和 Nature Review Genetics 有两篇综述认为 WGBS 测序最主要的问题是序列比对率低和比对准确性差两大问题。如果解决了这两个问题,就很容易打破 DNA 甲基化检测的瓶颈。
说干就干,我在实验室内下达了开展 DNA 甲基化检测召集令。首先,我将目前 DNA 甲基化检测所有方法的优缺点一一罗列,供同学们参考,如果有任何人能够提出全新的解决方案,实验室就给予不同程度的奖励。为了攻克这个难题,同学们想出了各种办法。最后大家认为既然 DNA 甲基化最主要的问题是比对的问题,我们就将重心放在如何提高比对率和准确性上。我们知道,含有 4 个碱基的基因组比对没有问题,我们何不借用双端测序的优势,让双端测序的一端是基因组原序列,另一端是转化后表观序列,那问题不就迎刃而解了。我们将这种全基因组 DNA 甲基化检测方法命名为 GPS(Guide Positioning Sequencing),即 “导航定位测序”,目前已经获得国内和国际专利。
这个大的策略一定下来,我们需要考虑的是如何实现这一设想。我们想到了 T4 DNA 聚合酶,在反应体系中没有 dNTP 的情况下,可以发挥 3’-5’ 外切酶的活性,当有反应体系中存在 dNTP 的时候可以发挥 5’-3’ 聚合酶的活性,不过在反应体系中,我们将 dCTP 换成甲基化的 dmCTP 就可以了。这样一来,所有的 DNA 片段 3’ 端在亚硫酸盐处理后还保持基因组序列,可用来定位;而 5’ 端就可以用来计算甲基化了。理论上,这种策略应该没有问题,我们大约用了一个月的时间证明这种策略没有问题,可以按照我们的设想 work,从而使复杂的全基因组 DNA 甲基化检测两大难题就这样变成了 “无心插柳”,化于无形。
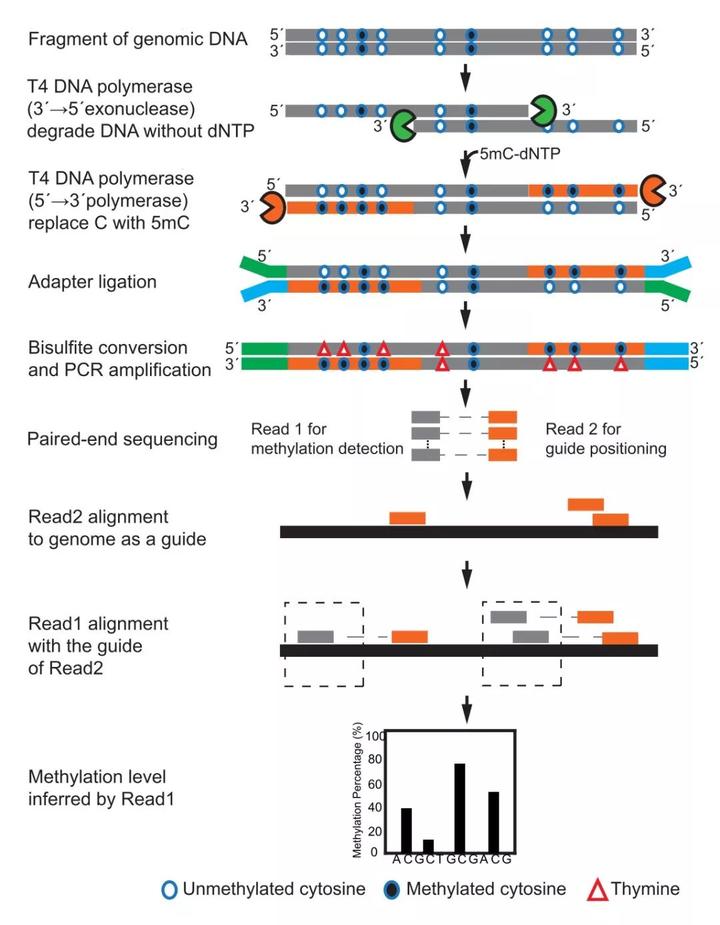
图 1. 导航定位测序(Guide Positioning Sequencing, GPS)工作原理
做研究的人都知道,一个全新方法的建立,说轻了就是自己和自己过不去,说重了就是自己给自己挖了个坑。要让一个全新的方法高效工作,谈何容易。我们要确定酶的用量,酶切的时间,酶切的温度,进而对酶的活性进行精准把控。如果把握不好,要么就将 DNA 全切光了,要么就切的很短,3’ 端无法定位。我还记得在这期间有一段艰难的时刻,我们想了好多办法,总是无法把握一个精准的度。实验结果反复无常,时好时坏,根本无法稳定下来。我们想了各种方法,总是以失败而告终。我可以说是绞尽脑汁,想的我实在头疼,刚好复旦大学工会组织职工休假,我就去扬州休息了两天,回来后接着想,终于将所有实验条件稳定下来。实验操作问题解决了,实验数据的分析又成了大问题,因为没有现成的软件可以用。我们只好根据我们的实验设计,自己编制分析软件。大约了用了 3 个月的时间,我们编制出第一版的软件。可是当我们比对测序数据时发现,200G 的数据需要计算机运行 3 周,这确实有点长,一般的实验室肯定吃不消。后来又编制了一版基于全新分析策略且需借助超级计算机的计算分析软件,200G 的数据大约运行 3 天,这解决了数据分析的大问题。但是,我们的本意是开发人人可以做的技术,可又有多少实验室可以用超算来分析 DNA 甲基化,怎么办?我们再次编制了一版全新的软件,目前在普通计算机上,200G 的数据运行时间为 3 天左右。现在,GPS 的实验条件和生物信息学分析软件已经全部优化好了。在 GPS 方法优化过程中,不难看出一个全新方法的产生和成熟确实需要花费很多的心思和精力,好在这些难题都留给了我们自己。我们实验室的理念是从我们实验室出去的方法,你按照 protocol 去尝试,就一定会 work 的很好,大家大可放心使用。

图 2. 研究论文于 2019 年 1 月 22 日发表在线发表于 Genome Research 上
GPS 优势想的见,看得见
GPS 检测全基因组 DNA 甲基化理论上简单,操作上易行,其优势不仅想的见,也看得见:
1)GPS 检测 DNA 甲基化的精确性:我们从人类参考基因组中随机生成了 100 万个 pair-end 读长并进行相应的改变,以模拟 pair-end 测序结果。由于已知这些片段确切的基因组位置,这样我们就可以通过 GPS 策略计算它们精确比对的概率,进而评估 GPS 检测 DNA 甲基化的准确性。如果使用 BSMAP 进行序列比对,其比对率仅为 66.2%,而 GPS 的比对率高达 82.3%,接近于用 Bowtie2 进行基因组的比对率 86.3%。在后续具体的实际测序和分析中,我们用焦磷酸测序实验也证明 GPS 具有极高的准确性,证明 GPS 具有精准检测的先天优势。
2)GPS 具有较高的比对率:在对肝细胞实际检测中,GPS 比对率为 80.9%,比 WGBS 的比对率高 15~20%,这主要是由于 WGBS 数据的先天的复杂度降低所致。如果这还不足以让您了解 GPS 方法的高效率,我给您一个目前 DNA 甲基化检测的现状。在人类参考基因组中 DNA 双链中有 1,170,378,405 个胞嘧啶(C)位点,以及 56,434,896 个 CpG 位点。以目前大家常用的基因芯片检测方法为例,如 450K 或 850K 芯片能够检测到的甲基化位点仅为 45 万个或 85 万个,占到人体基因组全部 CpG 的 0.8 到 1.5% 之间,占全部胞嘧啶的 0.04% 到 0.07% 左右,而 RRBS 能够检测的 CpG 位点大约为 1%,占全部胞嘧啶的比例大家可以自己算。WGBS 一般情况下能够覆盖全部 CpG 位点的 90% 左右,认为可以用来准确评估样本的 DNA 甲基化状态。我们来看一下 GPS 的检测效率,在肝细胞中,GPS 方法覆盖到了 54,853,393 个 CpG 位点,覆盖率高达 97%,同时也覆盖到了 1,123,233,333 胞嘧啶位点,覆盖率为 96%。从严格意义上来讲,只有全部确定了人体基因组中每一个胞嘧啶位点的甲基化状态才能算是绘制了人体细胞的表观基因组完整图谱,我们认为 GPS 方法至少帮我们绘制了第一张人类肝细胞的表观基因组图谱(也许有读者有不同意见,我们毕竟没有覆盖到 100%,但是我敢肯定,我们最接近这个目标)。
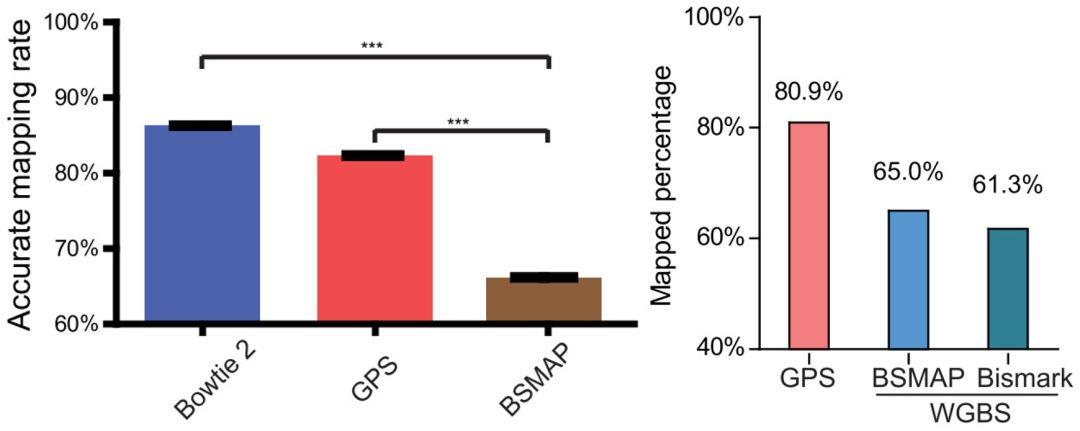
图 3. GPS 比 WGBS 具有更高的比对率
3)GPS 甲基化检测成本低:这主要基于 GPS 方法的比对率高,同时 GPS 测序数据比对只要超过 5 层,就能够比较精准的计算出 DNA 甲基化。而 WGBS 的精准检测甲基化一般情况下需要超过 30 层。目前 GPS 方法对一个样本的检测大约需要 200G 左右的测序数据,在 10X Illumina 测序平台上大约相当于 2 条 Lane 的测序数据,测序成本大约在 1.5 万元左右。况且你可以同时获得了基因组和表观基因组数据,一举两得。
4)GPS 检测甲基化没有序列偏好性:通过比较 GPS 测序和人类基因组功能区的分布情况,很清楚地看到,GPS 检测到的 DNA 甲基化位点在启动子区域和功能基因组元件上没有分布偏好性。与 WGBS 相比,GPS 对于重复序列、CpG 岛以及 GC-rich 区域(如启动子区域)的检测具有更高的效率。这些优势对全基因组的 DNA 甲基化精准检测非常重要,可以有效避免测序偏差导致结论的不确定性。例如,肿瘤细胞存在全基因组的 DNA 低甲基化现象,而偏偏 WGBS 倾向于检测 DNA 的高甲基化区域,而依靠 WGBS 来评估肿瘤细胞的全基因组 DNA 甲基化状态就会高估肿瘤细胞的 DNA 实际的甲基化水平,我们的结果也证明了这一点。
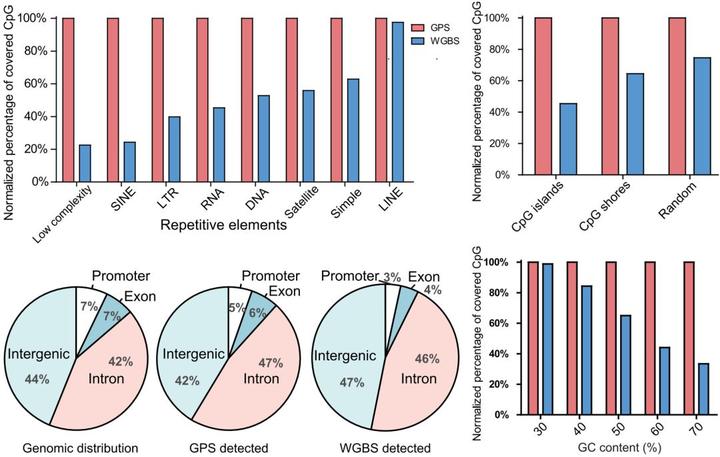
图 4. GPS 在全基因组范围的覆盖无偏好性,可覆盖重复序列和 GC-rich 区域
5)GPS 可以同时检测表观基因组和基因组学变异,特别适用于精准检测等位基因特异性的甲基化(Allele-Specific Methylation, ASM),而 ASM 检测可以有助于回答许多表观遗传调控的关键基础问题。例如,使用相同的数据量,GPS 鉴定了 1820 个 ASM,而 WGBS 只鉴定了 135 个。我们也验证了 97L 细胞系中的两个 ASM,它们定位于 CCDC97 和 TOP1MT 基因,这些区域富含转录因子和 DNaseI 高敏感位点。因此, GPS 更适用于研究基因组和表观基因组之间的交互作用(Crosstalk),而以前这些问题很难研究清楚。
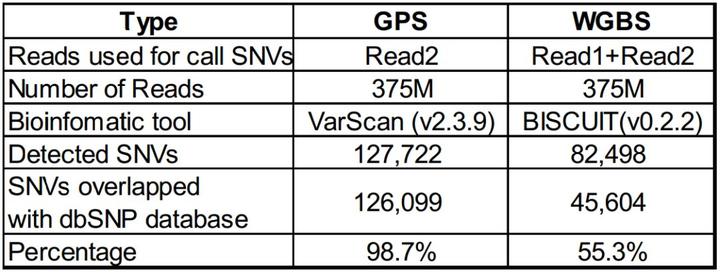
图 5. 相比于 WGBS,GPS 可以检测到更多的遗传变异
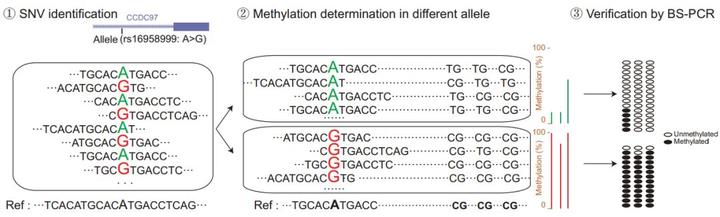
图 6. GPS 可检测等位基因特异性的甲基化
MeGDP,DNA 甲基化调控 “相反相成”
众所周知,DNA 甲基化与基因表达调控密切相关。基因启动子区域的高甲基化,基因表达降低,而启动子区域的低甲基化,则基因表达升高,这种调控规律深入人心。但令人尴尬的是,如果将一种特定组织细胞中所有启动子区域的甲基化状态与全部表达基因进行相关分析后,发现二者并没有显著相关性,是不是有点让人不知所措,就像局部的规律性并不能代表整体的规律性一样。此外,令人大跌眼镜的是某些基因的启动子区域高甲基化并不意味着这个基因表达一定降低,也就是说,有些基因启动子区域的高甲基化,这个基因反而是高表达的,由此看来,DNA 甲基化与基因表达调控并不是我们想象的那么简单。后来全基因组 DNA 甲基化的检测分析发现,基因体的甲基化与启动子区域正好相反,即基因体的高甲基化与基因的高表达有关,反之亦反。我们的 GPS 检测分析发现,这种规律也并不总是对的。例如,基因表达 FPKM 超过 20 时,基因体 DNA 甲基化不再与基因表达正相关。结果显示,FPKM 超过 20 的基因体甲基化程度更低,长度更短,更为保守,而且主要富集在代谢通路上。
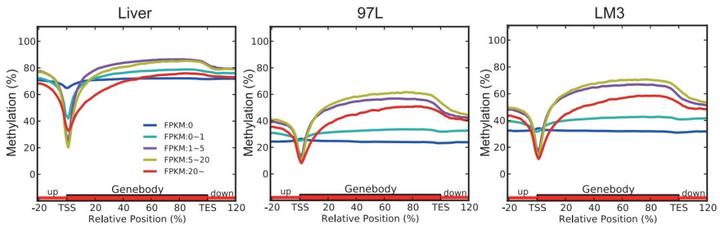
图 7. 基因表达与启动子区域以及基因体的 DNA 甲基化有关
那么问题来了,启动子和基因体 DNA 甲基化为什么会有截然相反的调控规律,它们之间有么有内在的联系并共同调控基因的表达?能否仅仅通过 DNA 甲基化检测来精准预测基因的表达情况,而这一点对于评估某些感兴趣的基因在特殊样本(如石蜡样本)中的表达无疑具有重要意义。
鉴于 GPS 检测每一个 CpG 位点甲基化的精准性,当我们用基因体和启动子区域的 DNA 甲基化差值(MeGDP, Methylation of Genebody Difference to Promoter)与基因的表达进行相关性分析时,我们惊喜地发现 MeGDP 与基因表达之间的相关性高达 0.67,提示 MeGDP 可以用来预测基因表达的情况。而如果利用 WGBS 测到的数据进行计算,得到相关系数仅为 0.33。在其他样本中应用 GPS,我们也能得到类似的结果,而 WGBS 结果则毫无规律性可言。可以这么说,MeGDP 的发现,并不是我们比别人聪明能干,而是得益于 GPS 对甲基化的精准检测。
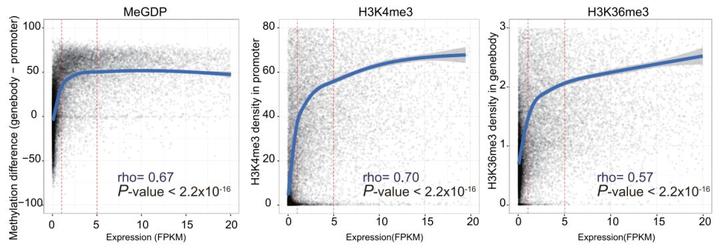
图 8. MeGDP、H3K4me3 和 H3K36me3 与基因表达均存在很强相关性
MeGDP,肿瘤免疫新框架和新靶标
你千万不要小看 MeGDP, 除了用于特殊样本中基因表达的预测,其重要性远不至此。大家都知道肿瘤的发生与免疫功能紊乱以及代谢异常密切相关,但表观遗传因素在这里发挥了什么作用不得而知。在肝癌细胞中,因 MeGDP 降低导致表达下调的基因主要富集在免疫与刺激反应以及代谢途径相关基因,而且 P 值非常低(做过 GO 分析的人都十分清楚,P 值代表了所富集基因的可信度),由此可见 MeGDP 可以更好地用来研究肿瘤相关基因的表达调控与肿瘤各种生物学行为的关系。
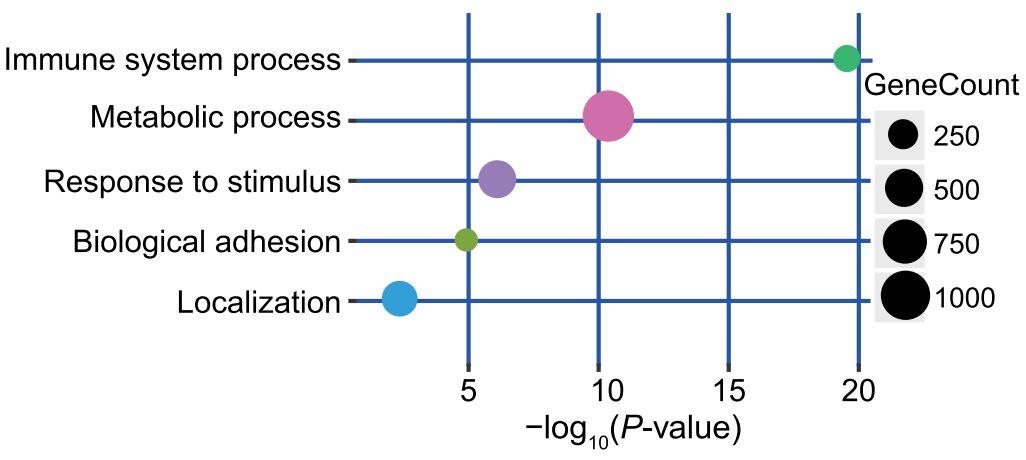
图 9. 在肝癌细胞中具有更低 MeGDP 且表达下调的基因富集在免疫系统以及代谢通路上
众所周知,肿瘤发生与免疫系统紊乱有极大的关系,免疫监视系统失衡是肿瘤发生的重要原因。这里面重要的概念就是肿瘤与免疫监视系统的相互作用(tumor-immune surveillance network)并且包含两层意思,一个是肿瘤细胞自身,一个是人体的免疫系统,到目前为止肿瘤如何逃避免疫系统还是一个谜。一般的理解,肿瘤与免疫监视系统相互作用的重点是会发在免疫系统上面,也就是肿瘤中的各种淋巴细胞异常,比如最近火热的免疫治疗正是针对这些不作为的免疫细胞。这让我这个免疫学的外行比较纳闷,研究肿瘤免疫,大家居然主要关注的是免疫系统,而不是肿瘤细胞自身;近来有些研究去寻找肿瘤新生抗原如 Neoantigen,理论很高大上,可是实际可应用的并不多。这里,我认为有必要重新认识并深入理解肿瘤免疫,尤其是从肿瘤细胞自身来重新诠释肿瘤免疫。站在表观遗传的角度,任何细胞都可能是 “免疫细胞”,或具有免疫细胞的特性,所以肿瘤细胞自身免疫相关基因的调控也是肿瘤免疫调控的重要组成部分。换一句话说,肿瘤免疫我们不仅要关注免疫系统,更需要关注肿瘤细胞内在的天然免疫系统基因的调控,而肿瘤细胞中内在的免疫相关基因的甲基化异常导致的基因沉默也许是肿瘤免疫逃逸的重要原因。
我们的研究结果表明,由于 MeGDP 导致的甲基化异常,肿瘤细胞中内源性的免疫相关基因被异常甲基化所沉默,导致肿瘤细胞对外界的各种治疗或免疫治疗没有反应。据此,我们推测,肿瘤的免疫耐受与免疫系统中的淋巴细胞也许没有必然的关系,而由肿瘤细胞自身的表观遗传学异常这个内因决定。在这个新的肿瘤免疫框架下,寻找预测肿瘤免疫治疗的新靶标就不会再 “山穷水复疑无路”,而一定会 “柳暗花明又一村”。
如果你还不相信这么复杂的问题,答案竟如此简单,那么我们依据该思路对 GPS 测序结果精细分析一定会让你茅塞顿开。目前 PD-1/PD-L1 抗体治疗 80% 肿瘤患者没有显著效果,没有人愿意成为这 80% 中的一员。如果患者花费巨资却发现免疫治疗效果有限或无效时,患者的心情可想而知。有人说钱能解决的问题都是最简单的问题,对肿瘤患者来说,也许最关键的问题是时间的问题,因为没有人耽误得起。所以,摆在临床医生和广大患者面前的一个重要且迫切的问题就是找出一个能够预测 PD-1 治疗有效性的标志物。现在的一些标志物,比如 PD-L1 的表达量,可是在临床的验证中并不好用。你也许会说肿瘤突变负荷(TMB)不错,但我要说 TMB 并不是免疫治疗特有预测标志物,也可以预测其他治疗方案的效果。总而言之,目前的预测免疫治疗的有效标志物,临床医生不满意,患者更不满意。近期 Cell 杂志报道,在黑色素瘤病人中,免疫监控相关的干扰素 IFNG 通路基因的突变或拷贝数丢失使得抗 - CALA4 反应失效。可惜的是,仔细查看文章发现 IFN 通路上 60 多个基因突变的概率太低,虽然很有意义,但确实临床应用十分有限。怎么办,该轮到我们的 MeGDP 粉墨登场了。我们通过对 IFN 通路 60 多个基因的 MeGDP 与基因表达分析发现,MeGDP 异常在肝癌细胞中确实可以导致 IFN 通路中的大多数基因表达下调(图 10 左中图),进而可能用于 PD-1 治疗效果的预测。如果想进一步破解 PD-1 治疗不佳的魔咒,我们认为 DNA 甲基化抑制剂有可能派上用场,而且已有研究证明,5AZA 确实与肿瘤细胞自身的免疫激活有关。我们也发现,在 5-AZA 处理后,免疫相关基因 EDNRB、ACP5 以及 BST2 都上调大约 2~75 倍(图 10 右图)。此外,我们也有理由推测肝癌细胞中的 MeGDP 的异常模式导致的免疫相关基因沉默也许是目前肝癌药物和其他疗法不佳的重要因素。
如果哪位老师愿意一起合作研究 MeGDP 与免疫治疗的关系,并打算筛选新的免疫治疗新靶标,我们将非常高兴,大家一起合作做点事。联系邮箱:wenqiangyu@fudan.edu.cn
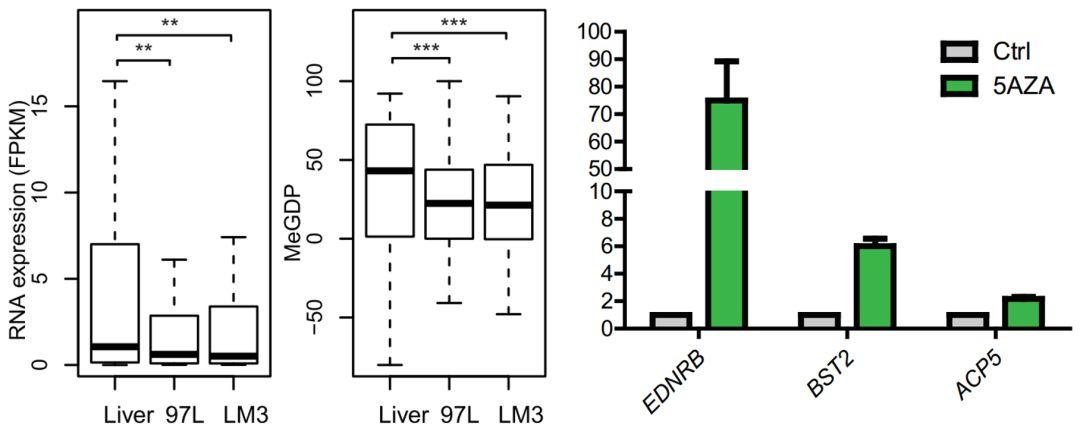
图 10. 肝癌细胞系中 IFNG 通路基因表达下调;5-AZA 处理后,免疫相关的基因上调。
MBS:DNA 甲基化调控不仅需要高度,更需要广度
MBS 的发现和定义纯属偶然。MBS 即 “Methylation Boundary Shift” 的缩写,中文的意思是甲基化边界漂移。谈到 DNA 甲基化对基因的调控,我们大多关注 DNA 甲基化高和低,因为这直接决定了基因表达的低与高。然而,当我们将正常肝细胞和肝癌细胞的甲基化测序数据比对到参考基因组上后,我们在 UCSC genome browser 上发现,与正常肝细胞相比,肝癌细胞中以 TSS 为中心的启动子区域 DNA 低甲基化范围在大多数情况下总是显示出更广阔的“V” 字形模式(wider opening)。这是巧合还是规律,我们一时也不能确定。但我们知道,用我们分辨率不高的肉眼看到的东西如果是真的,生物信息学分析可能会给我们一个满意的答案,结果我们毫无悬念的发现肿瘤细胞中启动子区域确实存在甲基化边界的漂移(图 11 左图)。
如图所示,MBS 现象在肿瘤细胞中非常清楚,毫不拖泥带水,我们自然要问,MBS 在肿瘤细胞中的出现,一定会有重要的生物学功能:
1)MBS 与组蛋白修饰离不得,见不得。与 H3K4me3 离不得,很清楚 MBS 所在区域与 H4K3me3 高度重叠;相反 MBS 与 H3K36me3 却是见不得,因为 MBS 与 H3K36me3 富集是互斥的。
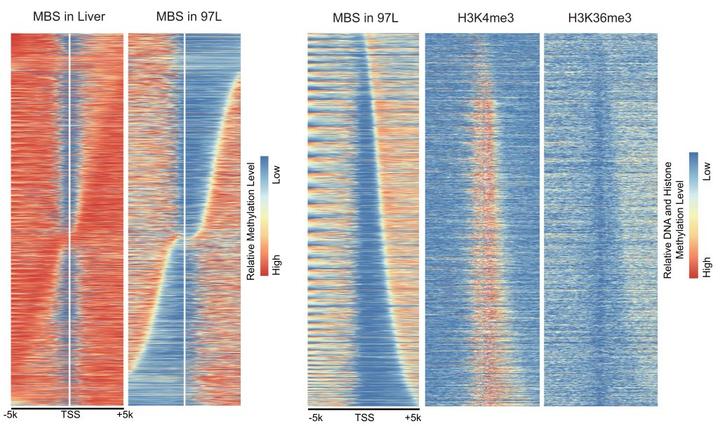
图 11. 97L 细胞中 MBS、H3K4me3 和 H3K36me3 的分布
2)MBS 与基因表达有关。我们的结果表明 MBS 向基因体方向的的漂移与基因的高表达密切关联。MYC 基因就是一个典型的例子,肿瘤细胞中 MYC 基因高表达,但其调控机制五花八门,而我们在这里清楚地看到 MYC 基因的启动子区域存在显著的甲基化边界漂移,说明 MBS 至少在一定程度上与肿瘤细胞中 MYC 基因的表达上调有关。
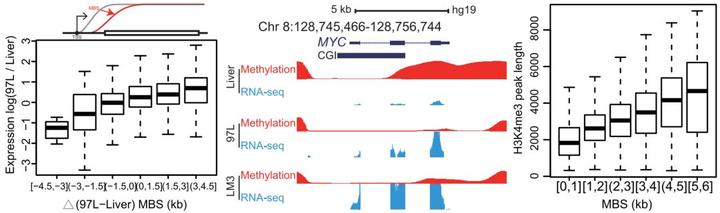
图 12. MBS 对基因表达的调控
既然 MBS 与基因表达有关,那么 MBS 是否有肿瘤的发生相关呢?通过对肝癌细胞中异常的 MBS 模式及相关基因表达进行分析后发现,这些基因富集在核糖体和细胞周期相关的通路。作为曾经的血液内科临床医生,我知道白血病细胞与正常造血祖细胞的形态学鉴定,很重要的一条标准就是细胞核中核仁的数量,核仁数量越多,是白血病细胞的可能性就越高,而核仁的增多离不开核糖体相关基因的高表达。如果按照传统的甲基化调控理论,你会认为 rRNA 基因不受表观遗传学调控,因为所有 rRNA 基因的启动子均是低甲基化。而 MBS 的发现告诉你,DNA 甲基化的边界漂移居然与肿瘤中 rRNA 的高表达有关,是不是有点脑洞大开的感觉。进一步分析发现,在 60 多个核糖体相关基因中,有 48 个核糖体基因的表达调控与 MBS 相关,而应用 WGBS 只发现了 7 个,再一次印证了 GPS 检测甲基化的精准性。可以想象,如果甲基化的检测准确性存疑,甲基化边界的漂移鉴定就变成了一项不可能完成的任务,或者你比较幸运,偶尔在 WGBS 数据中发现了 MBS,但因为在下一个样本中没法重复,也很难得到规律性的结论。我们在两个乳腺癌细胞系 MCF-10A 以及 MCF-10A-1H 中进行 GPS 测序,同样发现 MBS 及相似的调控规律,说明 MBS 调控具有普遍性。
MBS:增强子与细胞身份 “得” 与“失”
人类基因组中有数百万的增强子元件,其中 H3K27ac 是活性增强子的标签。既然启动子区域存在明显的 MBS,我们自然会想到,作为与启动子类似的顺式调控元件,增强子是否也受到 MBS 的调控。我们的答案是 “Yes”。在肝细胞和肝癌细胞中,H3K27ac 的峰宽也与 MBS 高度重叠(图 13 左),提示 MBS 与 H3K27ac 具有相关性,进而对基因表达产生影响。与正常肝细胞相比,肝癌细胞的 MBS 发生了显著的变化,进而引起增强子活性的选择性丢失或重新获得,这些增强子变化可导致相应的基因表达发生变化。我们惊讶地看到许多基因与细胞的身份相关,如肺发育、免疫细胞激活或其他组织特异性的基因。我们有理由相信,正是由于 MBS 异常模式导致肿瘤细胞增强子边界和活性变化,进而促使组织特异性基因表达上调或下调,引起细胞身份的“得” 与“失”,而这一切也许在肿瘤发生和转移过程中与肿瘤细胞特性的形成密切相关。
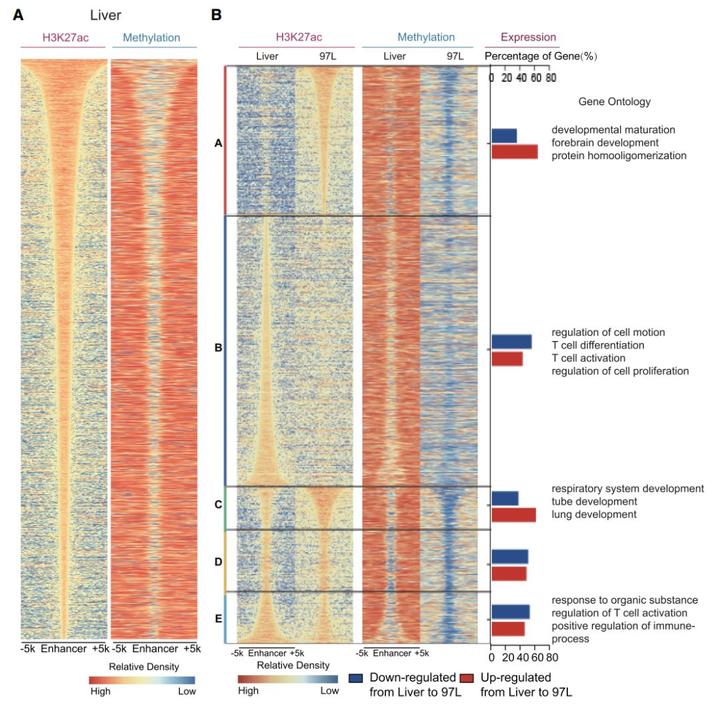
图 13. 增强子与 MBS 具有一致性
肿瘤转移,也许就是肿瘤细胞换了 “马甲”
转移是晚期肿瘤的显著特征,关于肿瘤转移的假说层出不穷,主要包含肿瘤干细胞理论、肿瘤微环境理论、上皮 - 间质转化理论、“种子 - 土壤”学说等 10 种。但经验告诉我们,对于绝大多数肿瘤而言,一旦转移,留给患者的时间就不多了。到目前为止,肿瘤转移的研究还停留在 “假说” 阶段。换句话说,假说依然是 “假” 的,并没有被证实,所以我们对于肿瘤转移依然束手无策。
图 14. 肿瘤转移的 10 种理论或假说
“同化共生”,是我们基于我们的研究结果提出的有关肿瘤转移的一个新概念。肿瘤细胞通过改变身份与特异性转移的器官相互适应,进而在转移的组织器官中与新的环境 “同化共生”,可能是肿瘤转移的新机制。物以类聚,人以群分,肿瘤的发生和转移也一样。肿瘤转移是肿瘤治疗失败的重要原因之一,而肿瘤特异性的器官转移机制并不清楚。例如肝癌容易发生肺转移,我们通过分析肝癌细胞 97L 和肝癌特异性转移到肺的 LM3 细胞的 DNA 甲基化模式和基因表达情况,发现肝细胞特异性的基因表达显著降低,而肺细胞特异性的基因表达上调,我们认为肝细胞身份丢失和肺细胞身份的获得是肝癌发生肺转移的重要原因。在肝癌发生和转移过程中,伴随异常的 DNA 甲基化介导的细胞身份的丢失和获得,使肺特异性基因表达增加,从而使肝癌细胞获得了肺细胞的身份,这有助于肝癌细胞在肺的环境中适应和生存,而这也许是肿瘤转移最重要的原因。简单的说,就是细胞换了个 “马甲”,从而实现了 “同化共生”。
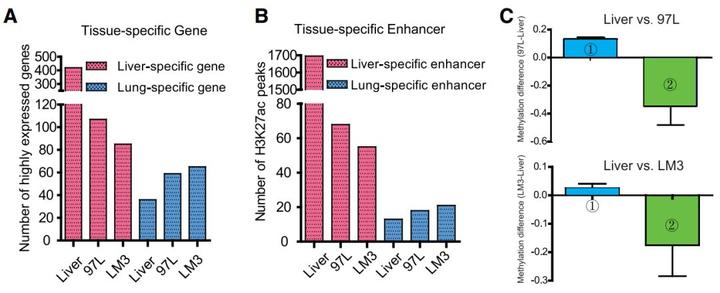
图 15. 肝癌发生过程中细胞身份的丢失
和我们预想的一样,在 97L 和 LM3 肝癌细胞系中,肝特异性高表达的基因的数目分别降低了 74% 和 80%。例如,在 97L 和 LM3 细胞中,肝特异性基因 ONECUT2 表达沉默,这与 H3K27ac 峰的丢失以及肝特异性增强子区域 DNA 甲基化的增加相一致(图 16D)。另一方面,在 97L 和 LM3 中观察到肺特异性基因 CKS2 的表达升高,这与 H3K27ac 峰的升高以及肺特异性增强子中 DNA 甲基化的降低相一致(图 16E)。在 LM3 中肺特异性的基因的表达可以帮助它们更好地在肺环境中适应和生存,即实现了 “同化共生”(Assimilated Symbiosis,我 coin 这么个词),而肿瘤细胞身份的丢失以及其他细胞身份的获得是肿瘤发生转移的前提条件和重要转折点。
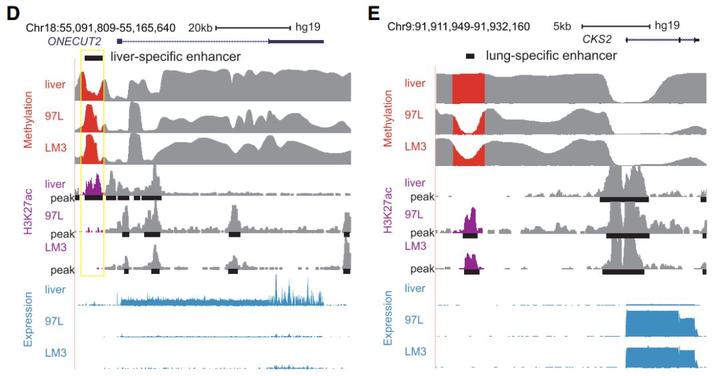
图 16. 肝癌发生和转移过程中肝 / 肺特异性基因的表达变化
同化共生背后的表观遗传因素可能是肿瘤发生特异性器官转移的重要分子机制,这为我们理解肿瘤转移提供了全新的视角。
后记
在这篇文章发表之际,我觉得我应该认真的总结一下,也需要写点什么,纪念一下自己,也纪念一下这 8 年逝去的光阴。GPS 甲基化测序一直是我们实验室的重头戏,我们投入了太多的人力、物力和我几乎全部的精力,这篇文章的图表超过 80 幅,工作量可想而知。在这 8 年时间里,我们实验室前后有 6 位博士或博士后投入研究,我们有过失败,反复的失败,但我们每一个人都扛下来了。该项研究前期在 Nature Biotechnology 审稿并根据审稿人的意见反复修改折磨 2 年多。用一位学生的话说,实验的失败其实并不算什么,面对审稿人反复折磨并在连续 3 次回答完全部问题后,又提出完全不同的新问题进行一遍又一遍的折腾,真是 “死的心都有了”。然而,正所谓不经风雨,难见彩虹。正是这种置于死地而后生的“炼狱” 式折磨,伴随着实验室的成长,也陪伴着我们每一位博士的成长,我也相信这一切也成就着我们每一个人的未来。这 8 年来,我们最大的收获也许是培养一批 “敢打敢拼” 博士生。 EpiRNAers 可以自豪的说,有 “EpiRNA Lab” 这碗酒垫底,以后还有什么酒过不去!而做别人不相信的研究,将是何等极致的人生体验。感谢他们!祝福他们!
撰文:徐鹏(复旦大学生物医学研究院博士生)
于文强(复旦大学生物医学研究院教授)
https://zhuanlan.zhihu.com/p/58086630

