循环细胞游离 DNA(cfDNA)在由细胞死亡后释放到血液中,是一种很有前途的候选生物标志物。在健康个体中,血液中的 cfDNA 来源于正常的细胞更新,而在患有癌症、自身免疫性疾病、移植反应和创伤患者中 cfDNA 水平升高。
DNA 甲基化具有细胞类型特异性。目前,有大量研究尝试利用 cfDNA 甲基化模式来分解 cfDNA 的起源组织。然而,已有的甲基化芯片覆盖率高,噪声相对较低,而 cfDNA 仅少量存在于血液中,必须从患者身上提取大量血液才能获得甲基化芯片所需的输入 DNA 量,因此,这无法适用于临床。此外,cfDNA 分解方法还会受到 DNA 甲基化位点(CpGs,)选择和参考细胞类型等的限制。
近日,加州大学洛杉矶分校 Noah Zaitlen 研究团队在Nature Communications上发表了题为 “Comprehensive cell type decomposition of circulating cell-free DNA with CelFiE” 的研究文章。为了解导致 cfDNA 在疾病患者中增加的原因,研究团队使用全基因组亚硫酸氢盐测序(WGBS)评估了 cfDNA 甲基化状态,并结合有效的期望最大化(EM)算法:CelFiE,能够准确地估计释放 cfDNA 的细胞类型和组织的相对丰度。与已有的分解方法不同,CelFiE 适应低覆盖率的数据,不需要 CpG 位点筛选,也能够检测出仅占总 cfDNA 一小部分的罕见细胞类型。

文章发表于Nature Communications
研究人员在模拟真实测序条件下,利用 cfDNA 混合物将 CelFiE 和其他方法进行了比较,包括最小二乘回归优化方法、“投影法”、优化的 “投影法” 和已发表的 cfDNA 分解工具 MethAtas。
MethAtlas 由 25 种组织和细胞类型组成,超过~ 6000 个 CpG 位点。为了确保公平比较,研究人员模拟了 25 种细胞类型和 6000 个 CpGs 与参考数据大小相匹配的数据。每个 CpG 的真实甲基化比例是从均匀分布中独立得出的,甲基化比例在 0% 到 100% 之间。对于 CelFiE 和投影法,是根据泊松分布模拟每个 CpG 的 cfDNA 读取深度,然后根据 cfDNA 混合物的细胞类型比例向量,将每个 CpG 的读取分配为一种细胞类型。
研究人员对 CelFiE 和所有比较方法进行了 50 次独立的模拟。在较低的读取深度下,CelFiE 的表现优于 MethAtlas 和线性最小二乘回归(图 1)。CelFiE 和投影优化方法在该条件下表现相似。与 CelFiE 不同,投影优化方法的主要限制是,不能估计缺失的细胞类型。
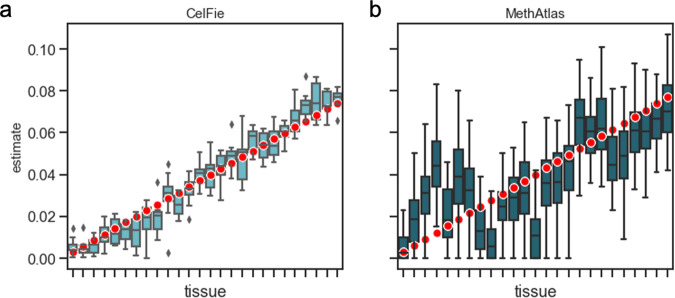
图 1 .CelFiE(A)和 MethAtlas(B)对模拟 cfDNA 混合物的分解。来源:Nature Communications
为进一步描述 CelFiE 的特性,研究人员改变了 CpGs 的数量(100、1000 和 10000),代表具有不同细胞类型信息量,并模拟了 10 种细胞类型,其中一种细胞类型是固定的,其余 9 个细胞类型比例是从一个独立的均匀分布,通过归一化,所有比例总和为 1。结果发现,随着位点数量的增加,CelFiE 分解 cfDNA 混合物的准确能力提高,尤其是对于数量较少的细胞类型。 随着测序深度的增加,固定细胞类型的估计甲基化比例与真实甲基化比例之间的相关性增加。(图 2)
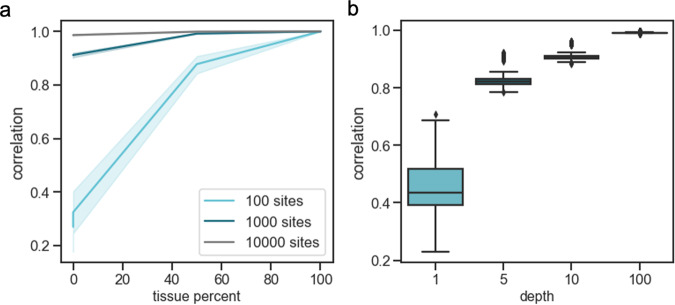
图 2.CelFiE 在模拟混合物上的性能。来源:Nature Communications
为评估 CelFiE 估计罕见细胞类型的能力,研究人员绘制了 cfDNA 混合物具有和不具有特定罕见细胞类型个体的 CelFiE 估计值(图 3a-d)。结果发现,随着深度和细胞类型比例的增加,CelFiE 区分罕见细胞类型的能力提高。 在深度为 5× 时, CelFiE 只能区分最丰富的固定细胞类型(5%),当继续增加深度时,CelFiE 能够检测到两组之间任意微小的差异。表明当读取深度较高时,CelFiE 可以准确地估计相对罕见丰度的细胞类型。
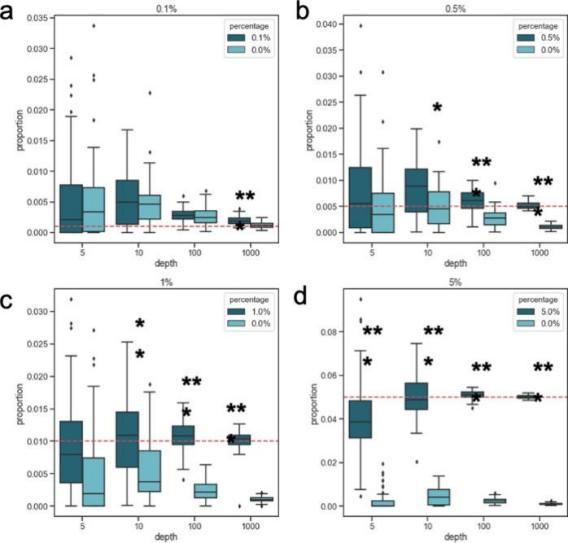
图 3. 具有罕见细胞类型 n=5(深蓝色)和不具有该细胞类型 n=5 的个体(浅蓝色)的细胞类型比例估计。来源:Nature Communications
同时,研究团队对 CelFiE 在估计未知细胞类型的能力进行了评估。通过模拟 t=10 个细胞类型,其中一种未知细胞类型被排除在参考数据之外,从均匀分布中提取已知细胞类型的剩余细胞类型比例,并将所有比例归一化总和为 1。研究团队模拟了 10 个、50 个、100 个、500 个和 1000 个个体的 cfDNA 读数。随着分解中人数的增加,CelFiE 的性能得到了改善。当添加额外的未知细胞类型时,需要更多的个体来准确估计。 (图 4)
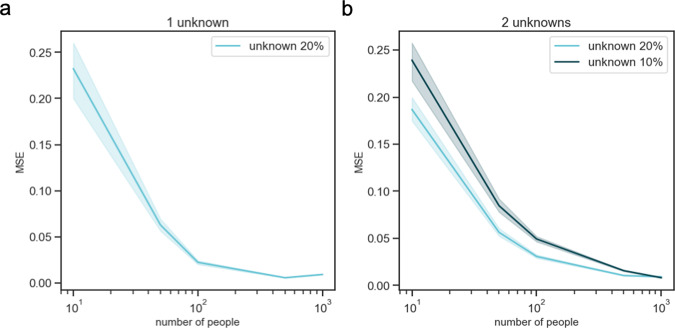
图 4. 参考文献中缺失细胞类型的 50 个独立模拟的 cfDNA 混合物的分解结果。来源:Nature Communications
最后,研究人员利用了 10 个 WGBS 数据集(小肠、胰腺、单核细胞、胃、胫神经、巨噬细胞、记忆 B 细胞、脂肪、中性粒细胞和 CD4+T 细胞),评估了 CelFiE 对 WGBS 真实数据生成的模拟混合物的分解能力。结果显示,CelFiE 在 WGBS 样品上表现良好(图 5a)。
此外,研究人员分析了 CelFiE 对含有大量未知成分的混合物的估计能力。首先,研究人员从参考样本中屏蔽了最丰富的细胞类型,即 CD4+T 细胞样本,对 100 人进行了 50 次模拟(图 5b)。随后,从参考组中屏蔽了两个参考样品,CD4+ T 细胞和小肠。结果发现,当有两种细胞类型缺失时,CelFiE 分解复杂混合物的能力下降,但估计的 WGBS 甲基化值与实际值的相关性仍然很高(图 5c)。
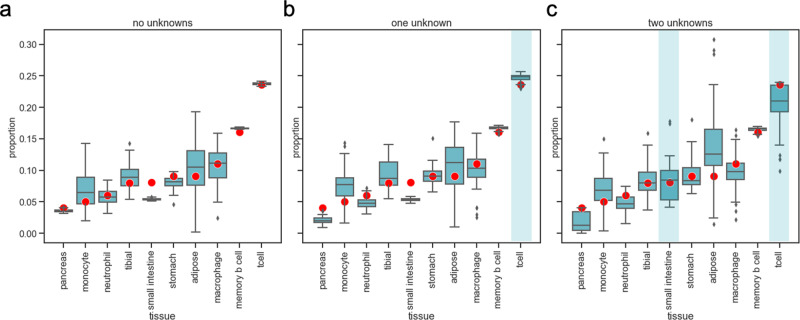
图 5.CelFiE 对模拟实验中随机选择的 50 个个体在真实 WGBS cfDNA 的细胞类型比例估计。来源:Nature Communications
在真实的临床情况中,CelFiE 正确估计了孕妇的胎盘 cfDNA 来源细胞类型(图 6)和肌萎缩侧索硬化症(ALS)患者的骨骼肌 cfDNA 来源 **细胞类型。**
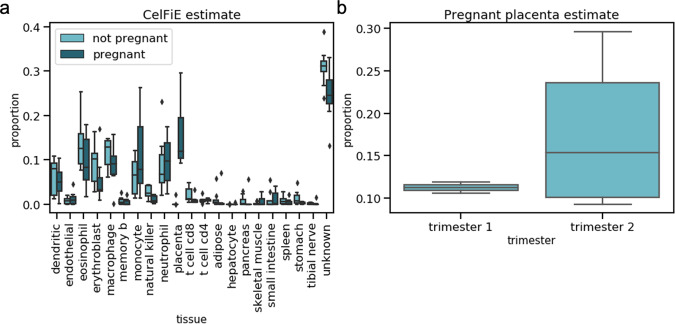
图 6. 来自孕妇和非孕妇对照的 cfDNA 的分解估计。来源:Nature Communications
综上所述,CelFiE 可以准确地分解 cfDNA 样本中测序覆盖率较低的 cfDNA 混合物,具有估计罕见和多种未知的细胞类型的能力。在临床应用情况中,CelFiE 的评估能力也得以验证,表明 CelFiE 有利于生物标志物发现和退行性疾病进展监测,量化复杂疾病中的细胞死亡情况。
参考文献:
Caggiano C, Celona B, Garton F, Mefford J, Black BL, Henderson R, Lomen-Hoerth C, Dahl A, Zaitlen N. Comprehensive cell type decomposition of circulating cell-free DNA with CelFiE. Nat Commun. 2021 May 11;12(1):2717.
·**END**·

热文 **推荐 **
GRAIL 癌症早筛产品 Galleri 最新检测数据公布:性能稳定,第二季度或将面世
基于 ctDNA 测序绘制晚期乳腺癌基因组图谱,揭示转移性乳腺癌耐药性产生机制

喜欢别忘了点 “在看” 呦!
https://mp.weixin.qq.com/s/Z0XGODVXfgp-eY4SvTjSTw

