
随着高通量低成本的生物组学技术的不断进步与发展,疾病相关的多模态多维度的组学大数据呈指数级增长,公共数据库中各种生物分子网络也逐步积累与完善。这为多角度多尺度刻画人类复杂疾病提供了丰富的数据来源与先验生物知识。通过对疾病的基因组、转录组、蛋白组、表观组等多层次组学数据的系统整合,才能更好地发现疾病相关的标记物,全面地挖掘其潜在机制并基于此展开精准诊疗。癌症是一种异质性极高的复杂疾病,同种癌症病人间也存在着巨大的差异,如何整合病人相关的多组学多模态数据,对其临床表型特征进行预测(如生存时间、分子分型、药物响应等),挖掘影响临床表型的内在生物机制是临床研究中极大的挑战,同样也是未来的一个研究热点方向。
近日,中科院计算研究所赵屹老师团队在Computational and Structural Biotechnology Journal发表文章 “DeepOmix: A scalable and interpretable multi-omics deep learning framework and application in cancer survival analysis”(2021 IF:7.2)DeepOmix 是一个可扩展可解释用于多组学数据整合的深度学习模型,如图 1 所示,输入为单一或多个组学的特征矩阵。 由于细胞中的基因并不是彼此独立行使功能,而是以信号通路或功能模块为单位进行工作,因此在该算法模型中引入了基因功能模块层,融合基因模块的生物先验信息(文章中使用的是信号通路基因集)用于整合来自样本的多组学特征信息,并将之应用于各类型癌症生存状态的预测。该研究通过训练模型可对病人的生存期进行预测,并得到样本数据在功能模块层的低维表示,通过统计分析可挖掘影响预后的基因功能模块。此外,DeepOmix 不仅可用于生存预测分析,也可用于预测药物反应等多种临床指标。
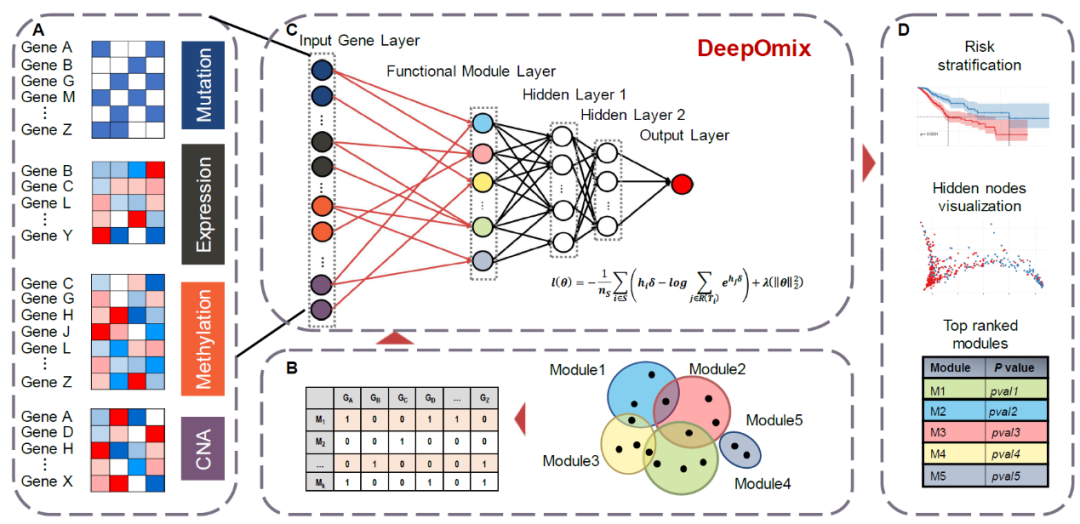
图 1. DeepOmix 的综合框架图
该研究所用数据是收集 TCGA 中多个癌种的多组学数据,以生存状态为训练目标进行交叉实验验证。DeepOmix 对生存周期的预测性能显著优于其他 5 种常用的基于组学数据预测生存周期的算法(图 2),包括基于传统机器学习的 BlockForest、glmBoost、IPFLasso 和基于深度学习的 DeepSurv 和 DeepHit。
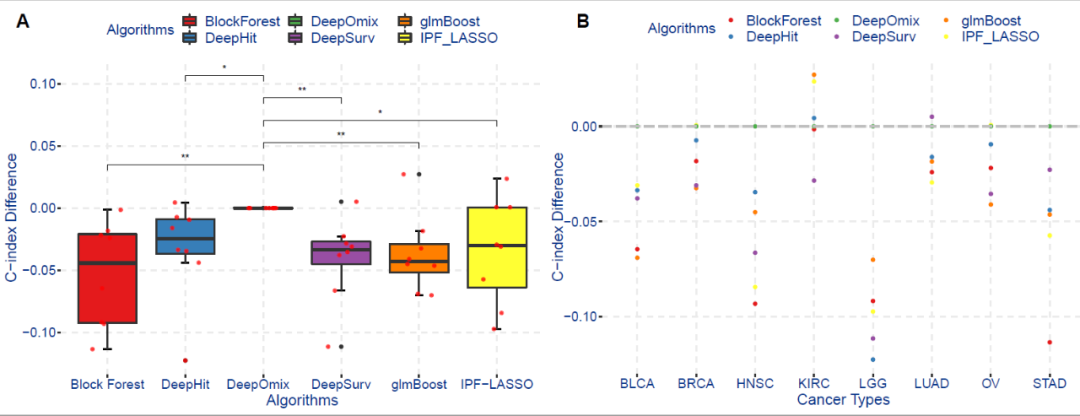
图 2. DeepOmix 与多个机器学习方法的 10 倍交叉实验验证的生存预测性能比较(基于八类癌种的多组学数据)
为了挖掘与预后相关的基因模块,该研究又以低级脑胶质瘤(LGG)多组学数据为例,将 DeepOmix 用于预测其生存状态,并基于输出层将样本进行分组,并在不同组别中根据基因功能模块层的低维表示分布差异的显著性对节点进行排序,排名前十位的信号通路(图 3)中几个信号通路均已有文献报导与脑胶质瘤或其他肿瘤密切相关。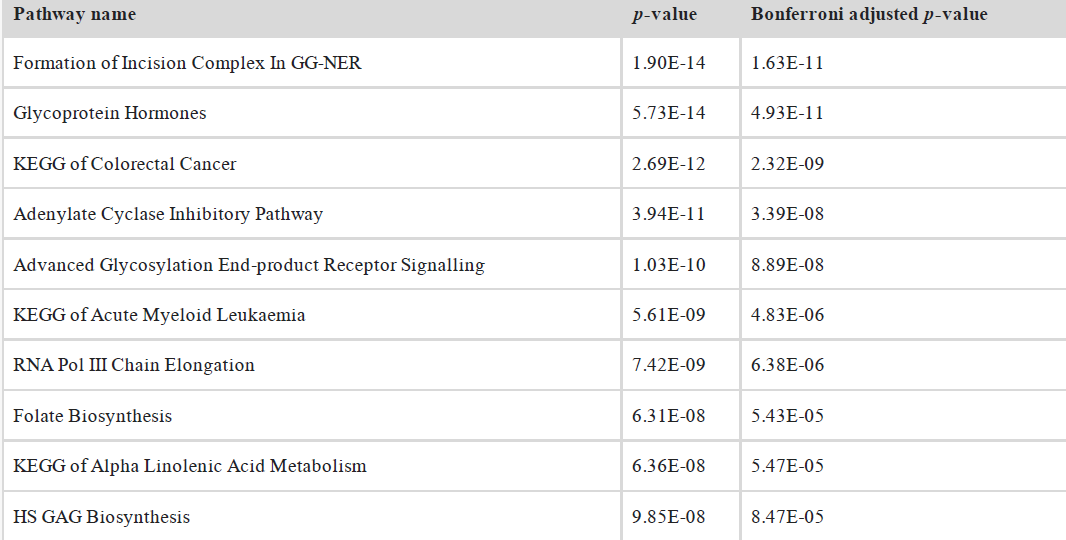
图 3.DeepOmix 在脑胶质瘤中发现的相关信号通路
综上,DeepOmix 为一个多组学整合的深度学习模型框架。随着生物技术的不断进步与样本实验数据的积累,DeepOmix 的可扩展性会随着不同研究目标和空间转录组等新型组学数据的深入而陆续更新。安装使用可访问https://github.com/CancerProfiling/DeepOmix。
热文推荐

