团队背景介绍
研究方向关键词:DNA methylation (DNAm), Cell-type heterogeneity, single-cell omics, statistics
大佬的文章产出很多,每年都有稳定产出
2017 ChAMP [Tool update]
文章访问地址:ChAMP: updated methylation analysis pipeline for Illumina BeadChips 
一、DNA甲基化芯片数据分析简介
DNA甲基化芯片:Illumina 850k EPIC array, Illumina 450k array。
DNA甲基化芯片的分析流程
由于 450k 和 850k 芯片使用了同样的技术,因此在分析上两者的相似性很高,许多生信软件针对 850k 芯片也做出了更新。
芯片分析的第一步是质控和标准化,450k 和 850k 芯片均采用了 Infinium I 和 Infinium Ⅱ 两种探针,导致不同类型探针的位点其 β 值分布不同,该问题是标准化步骤的重点,常见的标准化方法有多种包括:BMIQ、SWAN、Funnorm、Noob、PBC等等。
分析的第二步是去批次效应,批次效应是芯片技术常见的问题,针对于 DNA 甲基化芯片的去批次效应方法主要包括:ComBat、RUV。
分析的第三步通常是计算与表型关联的位点,这一步的关键之处在于矫正细胞的异质性。由于不同细胞的 DNA 甲基化组是特异的,而往往用于研究的组织都是不同细胞的混合体,各样本不同的细胞比例会影响结果。
矫正细胞的异质性通常分为有参法和无参法。广泛用于全血的有参法比较成熟,而缺乏参考甲基化组的其它组织则更依赖于无参法。常见的无参法主要有 RefFreeEWAS、MeDeCom、EDec等。
基于测序技术的 DNA 甲基化分析往往是以基因组区间为单位,鉴定的是差异甲基化区域(DMR),850k 芯片的推出导致可分析的位点更为密集,DMR 分析也成为了可选择的一步。
第四步是下游的富集分析。针对上述分析流程,有多个软件可以进行一条龙的分析,如 ChAMP、minfi、RnBeads2、missMethyl,本期 BioLinkX 生信半月谈主要对目前广泛使用的 ChAMP 包进行使用介绍。
2017.11 [review] DNAme的统计和综合分析
文章访问地址:Statistical and integrative system-level analysis of DNA methylation data
2018 CellDMC (contain in EpiDISH) [Tool]

This package contains a reference-based function to infer the proportions of a priori known cell subtypes present in a sample representing a mixture of such cell-types. Inference proceeds via one of 3 methods (Robust Partial Correlations-RPC, Cibersort (CBS), Constrained Projection (CP)), as determined by user.
【翻译:EpiDISH 包含一个功能,即基于参考数据集来推断出现在代表这种细胞类型的混合物的样本中存在的先验已知小区亚型的比例。 用户可以选择3种算法中的一种进行不同甲基化程度的细胞类型识别,这三种算法分别是 Robust Partial Correlations(RPC),CIBERSort(CBS),Constrained Projection(CP)。】
文章发表在:Identification of differentially methylated cell types in epigenome-wide association studies
GitHub地址:https://github.com/sjczheng/EpiDISH
生信技能树的软件测试:https://cloud.tencent.com/developer/article/1828884(建议先看看这个)
值得注意的是,作者还开发了网页版的 EpiDISH,访问地址:https://www.biosino.org/EpiDISH/
文章访问地址:EpiDISH web server: Epigenetic Dissection of Intra-Sample-Heterogeneity with online GUI
一、How to use
You will find the webpage quite self-explaining. You can just follow the order in the navigator(or download the pdf example file):
- Data preparation: Upload your beta value matrix, POI vector(optional) and covariates matrix(used in CellDMC; optional).
- Infer CT fraction: Select a mode and reference(s) to infer cell-type fractions. Check the results with interactive figures and save results in pdf and txt files.
- Run CellDMC: Run CellDMC with previsouly inferred CT fractions to identify differentially methylated cell types.
二、思考
1、如果要从样本中识别不同的细胞类型,需要使用到软件自建的参考数据集,EpiDISH包自建的数据集目前包含以下几个:
DummyBeta.m Dummy beta value matrixLiuDataSub.m Whole blood example beta value matrixcentBloodSub.m Whole blood reference of 188 tsDHS-DMCs and 7 blood cell subtypescentDHSbloodDMC.m Whole blood reference of 333 tsDHS-DMCs and 7 blood cell subtypescentEpiFibFatIC.m Reference for breast tissuecentEpiFibIC.m Reference for genenric epithelial tissue
但是假如你的样本与这些参考数据集的生物学意义上没有任何交集,意思是样本/组织/细胞来源差异非常大,那是否就没法进行细胞类型注释了?意味着这个软件对你就没用了呢?
有空测试一下这个包示例流程
# EpiDISH:https://github.com/sjczheng/EpiDISHBiocManager::install("EpiDISH")library(EpiDISH)
2020 EpiSCORE [Tool]

一、背景介绍
EpiSCORE这个工具的文章于2020年9月发表在Genome Biology上,链接:EPISCORE: cell type deconvolution of bulk tissue DNA methylomes from single-cell RNA-Seq data. Genome Biol 21, 221 (2020)
GitHub:https://github.com/aet21/EpiSCORE
值得注意的是,两年后,基于此工具进行的研究发表在Nature methods上:A pan-tissue DNA methylation atlas enables in silico decomposition of human tissue methylomes at cell-type resolution. Nat Methods 19, 296–306 (2022).
二、软件功能简介
EpiScore利用组织特异性单细胞RNA测序图谱构建相应的组织特异性DNA甲基化参考。
报道:http://www.sinh.cas.cn/xwgg/kyjz/202009/t20200904_5686519.html
中国科学院上海营养与健康研究所Andrew Teschendorff研究员指导的研究团队开发了一种名为“EPISCORE”的新型算法,用以从单细胞RNA-Seq中分析获得给定组织类型中的各种细胞类型,并从中发现蕴含与疾病发生相关DNA甲基化变化的特定细胞类型。EPISCORE算法利用了组织特异性单细胞RNA-Seq图谱数据(比如Human Cell Atlas Consortium)的强大功能和高分辨率特性,基于细胞类型特异性标记物的DNA甲基化差异特性,有效地将组织特异性mRNA表达图谱转化为单细胞分辨率的组织特异性DNA甲基化图谱。利用EPISCORE算法对肺癌数据开展研究,成功揭示了肺癌内皮细胞中的DNA甲基化表观遗传改变,提示表皮细胞向间充质细胞的转化过程是癌细胞入侵和转移的基础。
摘要:EpiSCORE是一个R包,用于构建组织特异性DNA甲基化参考矩阵,可随后与基于参考的细胞类型反卷积算法结合使用,以
(i)在相应的体细胞中获得细胞类型分数估算值存在全基因组DNAm谱的组织样本,以及
(ii)在一般Epigenome-Wide-Association研究的背景下推断细胞类型的特定差异DNA甲基化信号。
EpiScore特别适用于针对复杂的实体组织,无法通过实验生成代表组织内所有主要细胞类型的合适的DNAm参考矩阵。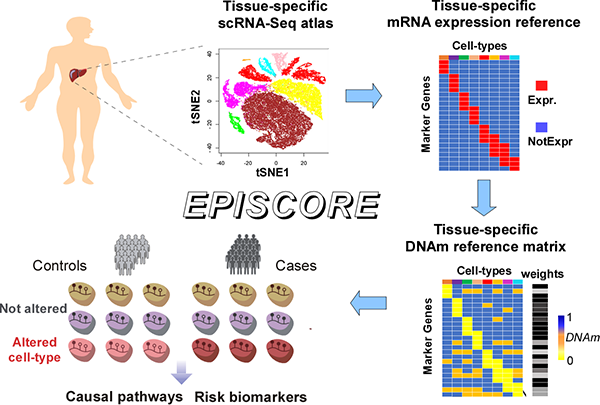
图. EPISCORE算法流程图
三、延伸阅读
从GitHub上别人的提问中可以发现当前版本还不支持WGBS, RRBS。见:https://github.com/aet21/EpiSCORE/issues/2


