一、表观遗传学的组蛋白修饰
染色体一个核小体由两个H2A,两个H2B,两个H3,两个H4组成的组蛋白八聚体和147bp缠绕在外面的DNA组成。组蛋白有很多修饰形式,包括组蛋白末端的乙酰化、甲基化、磷酸化、泛素化、ADP核糖基化等等,这些修饰都会影响基因的转录活性。而组蛋白H3是修饰最多的组蛋白。
组蛋白甲基化和乙酰化主要发生在它们的N-末端尾部并且可以影响基因的转录。大量研究表明,组蛋白乙酰化主要与基因激活有关,而甲基化取决于其位置和状态,与抑制或激活有关。组蛋白乙酰化主要发生在H3、H4的N端比较保守的赖氨酸位置上,是由组蛋白乙酰转移酶和组蛋白去乙酰化酶协调进行。特定基因部位的组蛋白乙酰化和去乙酰化是以一种非随机的、位置特异的方式进行。乙酰化可能通过对组蛋白电荷以及相互作用蛋白的影响,来调节基因转录。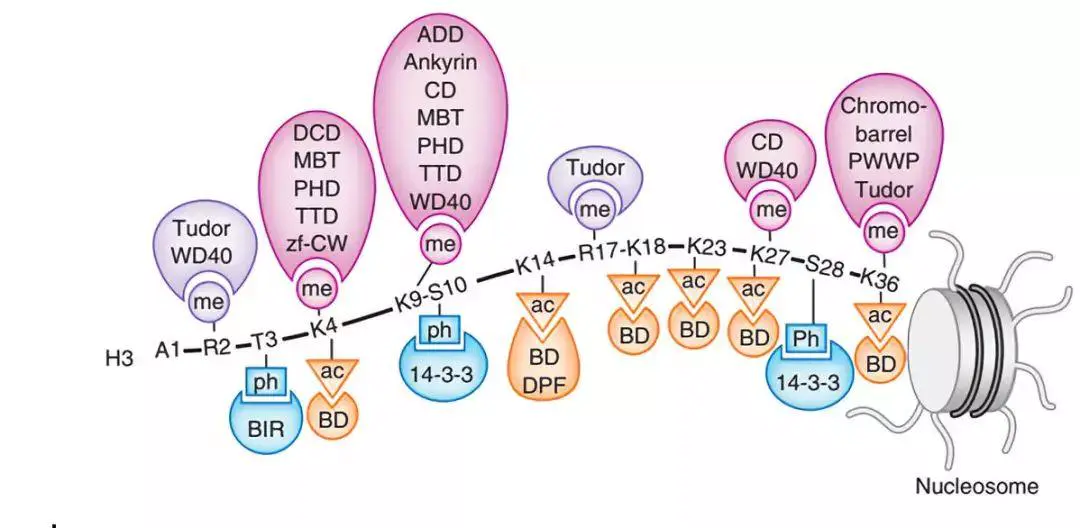
Figure 1 :Readers of histone PTMs. Recognition of themethylated(me) lysine, methylated (me) arginine, acetylated (ac) lysine andphosphorylated (ph) serine and threonine residues ofthe N-terminalhistone H3 tail by indicated readers.
图片来源:Perceiving the epigeneticlandscape through histone readers. Nat Struct Mol Biol,19, 1218-1227. E.L.Greer, and Y.Shi (2012)
组蛋白甲基化的位点是赖氨酸和精氨酸。赖氨酸可以分别被一、二、三甲基化,精氨酸只能被一、二甲基化。研究表明,组蛋白精氨酸甲基化是一种相对动态的标记,精氨酸甲基化与基因激活相关。相反,赖氨酸甲基化似乎是基因表达调控中一种较为稳定的标记。例如,H3第4位(H3K4)的赖氨酸残基甲基化与基因激活相关,而第9位和第27位赖氨酸(H3K9,H3K27)单甲基化与基因激活有关,三甲基化与基因沉默相关。
基因组包含大量的非编码DNA调控元件,包括沉默子、绝缘子、启动子和增强子,在基因表达中起重要作用。
启动子是RNA 聚合酶识别、结合和开始转录的一段DNA 序列,它含有RNA 聚合酶特异性结合和转录起始所需的保守序列,一般位于转录起始位点的上游。
增强子是指能够使基因转录频率明显增加的 DNA序列,是关键的调控元件,可以影响基因转录,而与其方向或距离无关,增强子通常可以远离其调节目标数千个碱基对。增强子有别于启动子处有两点:[1]增强子对于启动子的位置不固定,而能有很大的变动;[2]它能在两个方向产生相互作用。一个增强子并不限于促进某一特殊启动子的转录,它能刺激在它附近的任一启动子。
二、组蛋白修饰的CHIP-seq分析方法区分增强子和启动子
组蛋白修饰能预测染色质的类型(异染色质或常染色质)、区分基因组功能元件(启动子、增强子、基因主体)以及检测决定这些元件处于活性状态或是抑制状态。例如H3K4me2和H3K4me3修饰大多数富集在转录起始位点附近的启动子上激活基因表达,而H3K27me2和H3K27me3与基因抑制相关。
因此可通过CHIP-seq分析组蛋白修饰的分布寻找基因的启动子区和增强子区域及其是激活或抑制基因表达。
H3K4me1可作为增强子的标志,H3K4me3作为启动子标志。
研究表明,H3K4me1和H3K4me3与基因激活相关,H3K4me3主要富集在转录起始位点附近的启动子区域,而大多数H3K4me1修饰富集在增强子区域;H3K27ac与基因激活相关,主要富集在增强子和启动子区域,当增强子区只有H3K4me1修饰富集时,该增强子处于平衡状态,而当增强子区域同时富集H3K4me1和H3K27ac修饰时,该增强子就处于激活状态促进基因表达;H3K27的甲基化是可逆的过程,H3K27me1显示出对转录具有正向影响,启动子区域的H3K27me3甲基化修饰时抑制基因的转录,而H3K27me2广泛分布并且在沉默非细胞类型特异性增强子中起作用。
下表为常见的组蛋白修饰的主要分布及功能: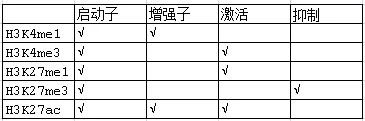
异染色质是染色质的浓缩,转录无活性状态,H3K9甲基化是异染色质的标志。H3K27me1和H3K9me3存在于着丝粒异染色质区域,而H3K27me3和H3K9me2共同存在于抑制的常染色质区域中。H3K9ac也与H3K14ac和H3K4me3高度共存共同作为活性基因启动子的标志。
三、应用案例
活性增强子鉴定 :
Histone H3K27ac separates active from poised enhancers and predicts developmental state. Creyghton, M.P. et al. Proc. Natl. Acad. Sci. USA 107,21931–21936 (2010)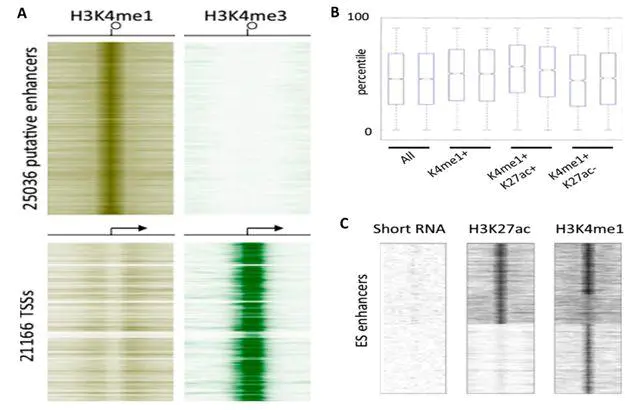
图注:A、使用ChIP-Seq鉴定的远端H3K4me1组蛋白标记鉴定了鼠ES细胞中的细胞类型特异性增强子:基于H3K4me1富集和非H3K4me3富集的的热图选出25,036个推定增强子。B、缺乏H3K27ac富集的增强子近端基因与平均增强子近端基因相比表现出较低的表达水平,表明H3K27ac是区分活性和平衡增强子状态的良好标志。C、选择富含H3K27ac的增强子使用先前发表的小RNA-Seq数据集检测这些短RNA表达与富含H3K27ac的增强子的关系,发现这些短RNA确实从H3K27ac阳性增强子转录。这再次支持H3K27ac是活性增强子元素的确定性因子。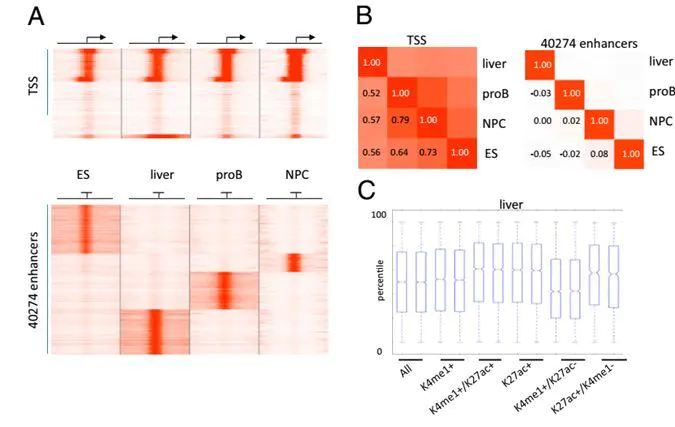
图注:使富含H3K4me1的远端区域的邻近基因活性增强是H3K27ac的功能。A、显示显示组织特异性分布的四种指定细胞/组织类型中增强子峰周围的四千碱基对H3K27ac富集的染色质状态(下图);B、A中所示的H3K27富集区域的相关性分析,近端基因的活性与所有成体组织中的远端H3K27ac富集正相关;C、成年肝脏的微阵列数据的基因表达,显示所有基因(全部)和发现与肝脏增强子特异性相关的基因的增强子富集(+)或未富集( - )的H3K4me1或H3K27ac的基因表达。
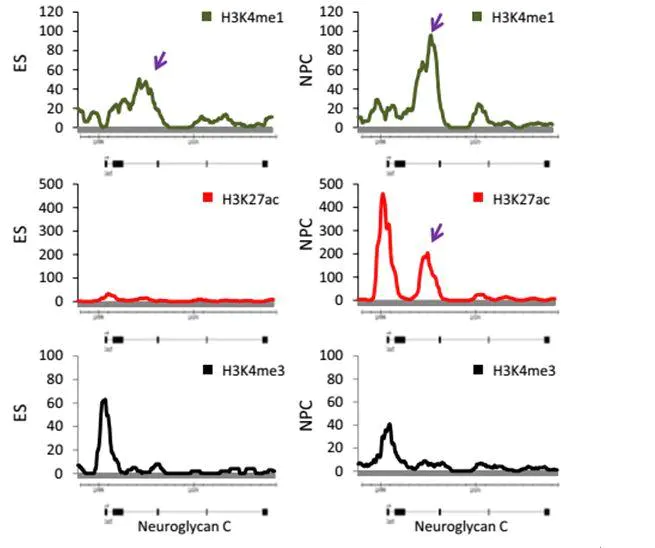
图注:神经糖蛋白是在ES细胞中具有平衡的增强子,其在神经祖细胞中变得活跃的基因。基因跟踪H3K4me1(绿色,顶部),H3K27ac(红色,中部)和H3K4me3(黑色,底部)在Neuroglycan C基因的20,792 bp大区域的富集。这些数据表明当细胞切换发育状态并且H3K27ac可用于区分两种增强子状态时,增强子通过激活平衡增强子在细胞分化中发挥确定性作用。
参考文献
1.Barski, A. et al. High-resolution profiling of histonemethylations in the human genome. Cell 129, 823–837 (2007) .doi:_10.1016/j.cell.2007.05.009
_2. Tian Y, Jia Z, Wang J, et al. (2011) Global Mapping ofH3K4me1 and H3K4me3 Reveals the Chromatin State-Based Cell Type-Specific GeneRegulation in Human Treg Cells. PLoS ONE 6(11): e27770.doi:10.1371/journal.pone.0027770
3. Musselman CA, Lalonde ME, Côté J, Kutateladz TG. Perceiving the epigenetic landscape through histone readers. Nat Struct Mol Biol 2012; 19: 1218–1227. doi:10.1038/nsmb.2436
4. Hong, S. et al. Identification of JmjC domain-containing UTX and JMJD3 ashistone H3 lysine 27 demethylases. Proc.Natl Acad. Sci. USA 104, 18439–18444 (2007). doi:10.1073pnas.0707292104
5.Creyghton, M.P. et al. Histone H3K27ac separates active frompoised enhancers and predicts developmental state. Proc. Natl.Acad. Sci. USA 107, 21931–21936 (2010).doi:10.1073/pnas.1016071107
作者:Ray钱
链接:https://www.jianshu.com/p/8aca72809c5c
来源:简书
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。

