
作者 陈河兵 陶 欢 伯晓晨 李 程
关键词:染色质三维结构 Hi-C 发育 疾病 ■
三维基因组研究主要关注染色质的三维结构与功能。染色质是遗传物质的载体,其复杂动态的三维结构参与调控DNA复制、基因表达等重要生物学过程,与疾病的发生发展密切相关。
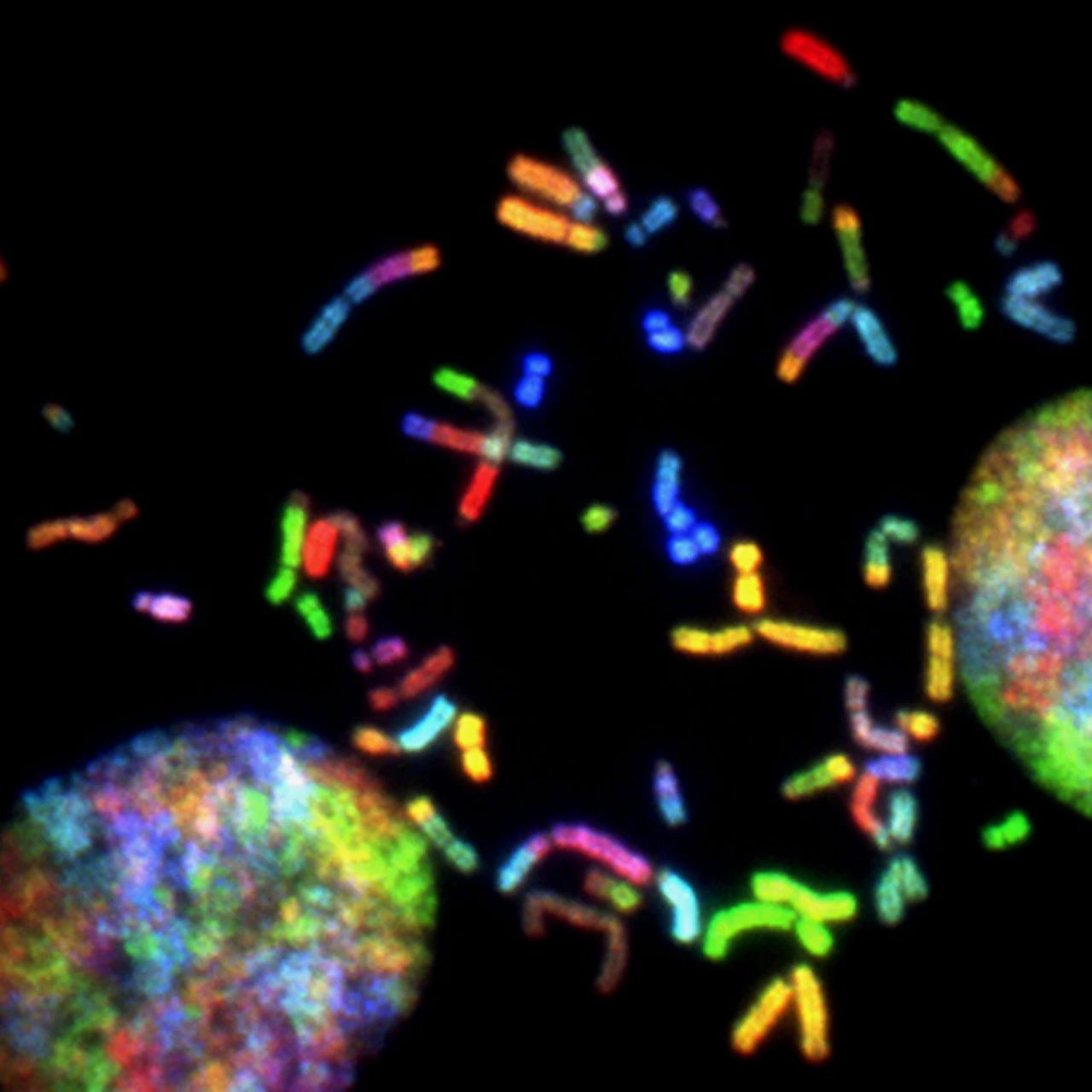
人类基因组分布在染色质上,包含约30亿个碱基对,如果我们每秒朗读一个碱基,每天读24小时,需要一个世纪的时间才能读完。当基因组DNA完全以线性排列方式展开时,其长度可达2米,而容纳基因组的细胞核只有几微米。这么长的DNA是如何被装进如此微小的细胞核中的呢?在这个过程中,DNA分子必定经历了复杂的折叠,并在折叠的同时调控基因表达、参与机体发育,并和疾病的发生发展密切关联。
奇妙的染色质结构
1953年,沃森和克里克提出了DNA分子双螺旋模型,并因此获得了诺贝尔奖。随着电子显微镜的发展,科学家们发现了染色质的基本结构单位——核小体。核小体由DNA缠绕组蛋白八聚体形成,并通过DNA串连起来形成直径为11纳米的“串珠”结构。核小体螺旋化形成30纳米的螺线管,进一步折叠形成300~700纳米的染色质,并在细胞分裂期浓缩形成1400纳米的染色体结构。整个过程中,基因组DNA被压缩了8000~10000倍,装进细胞的遗传信息储存中心——细胞核中。
经典的染色质多层级结构
巧妙的高通量染色体构象捕获技术
近年来,科学家发明了大量的方法来研究染色质的结构,主要包括基于电子显微镜的荧光原位杂交相关技术,以及基于测序的染色体构象捕获技术。而本文主要关注染色体构象捕获技术中的Hi-C技术及其衍生技术。
Hi-C技术及Hi-C衍生技术
2009年,科学家们研发了高通量染色体构象捕获技术(high-through chromosome conformation capture, Hi-C),为揭示染色质三维结构提供了有力的技术支撑 [1]。Hi-C技术将空间上相互作用的染色质通过甲醛固定,然后使用限制性内切酶切割基因组,并用生物素标记切割末端。再使用DNA连接酶连接带有生物素富集标记的邻近DNA片段,纯化和打断连接后的DNA分子,并筛选出带有生物素标记的DNA片段,最后对DNA文库进行高通量双端测序。Hi-C技术将细胞核中染色质空间互作的信息转换成测序信息,从而通过快捷的测序技术实现了全基因水平的染色质相互作用位点的识别。
在Hi-C技术的基础上,目前已有多种Hi-C衍生技术被开发,这些方法简化了Hi-C技术的实验流程,降低了实验噪声和成本,推动了染色质高级结构的研究。国内课题组也在相关技术上取得了大量突破性进展,如高分辨率BL-Hi-C技术以及简单高效的DLO Hi-C技术。笔者团队也开发了不依赖于探针序列及蛋白抗体,以高效富集全基因组活跃转录调控元件间相互作用的OCEAN-C技术 [2]。此外,笔者团队还与其他团队合作开发了基于Tn5转座酶扩增DNA片段的少量细胞Hi-C技术(tagHi-C)[3]。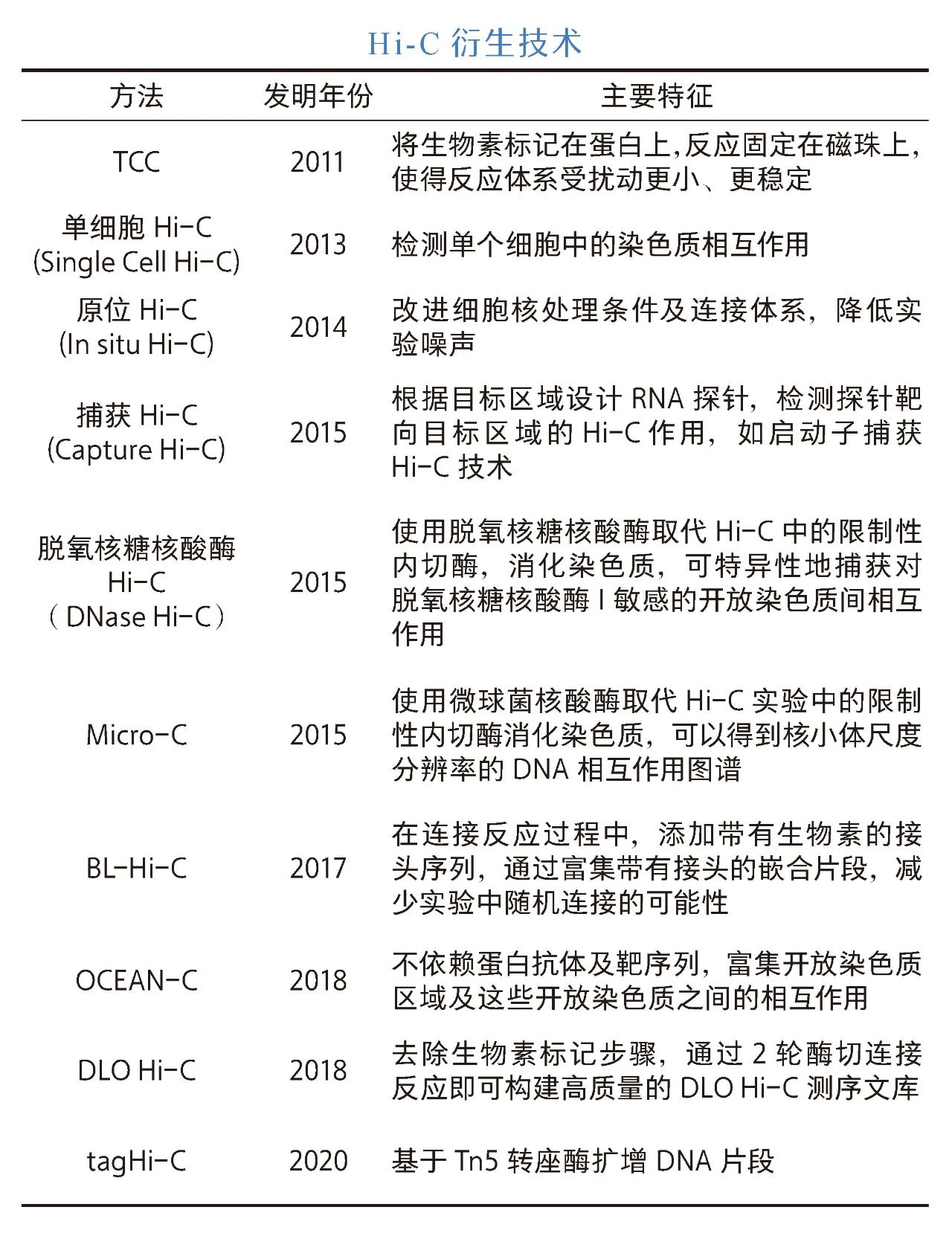
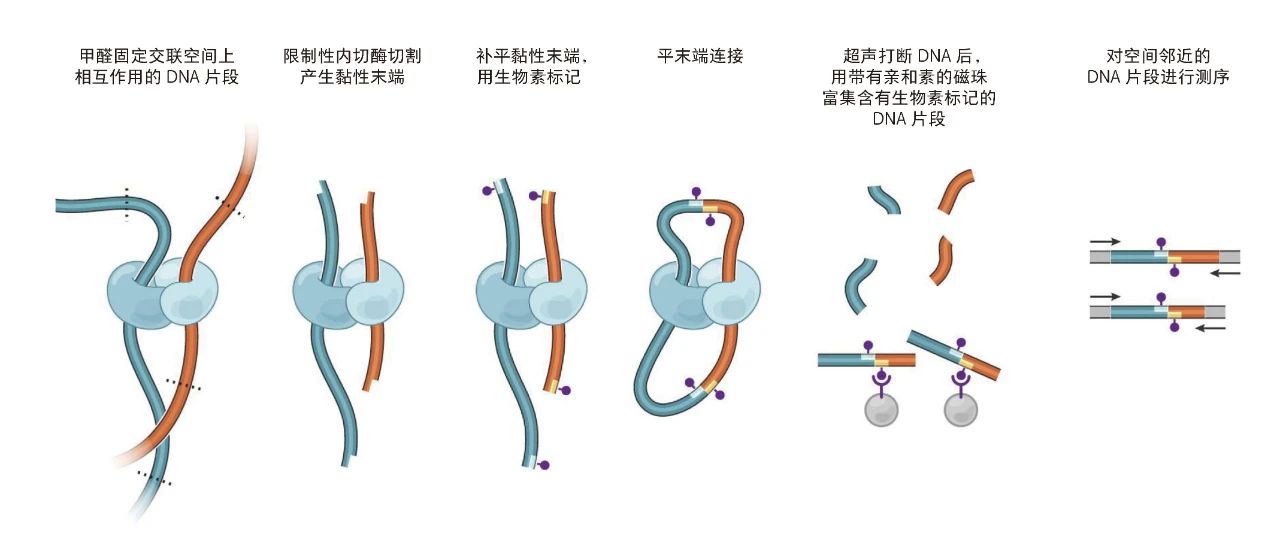
Hi-C技术实验原理
基于Hi-C识别的染色质多层级结构
Hi-C技术从一个新的角度展示了染色质的多层级结构。结合显微成像技术,研究者们发现细胞核中每条染色体倾向于占据独立不重叠的区域,这些区域被称为染色体疆域,疆域内的染色质会形成不同的区室结构,根据转录活性不同,这些区室可以被分为转录活跃的A区室及转录抑制的B区室,同类区室内染色质的相互作用较多,不同类区室间染色质的相互作用较少。随着测序精度的提升,研究人员发现区室内部1Mb左右的DNA组成了更小的空间结构,称为拓扑关联结构域 (topologically associating domain, TAD),TAD内一般包含8~10个基因,其内部的DNA元件之间形成了较为紧密的相互作用,而TAD间的染色质相互作用较少。相邻TAD边界上结合有染色质结构蛋白,如CTCF蛋白(CCCTC binding factor, CTCF)和黏连蛋白的蛋白复合体,这些蛋白起到组织染色质结构并隔离两个相邻的TAD之间互作的功能。TAD内部进一步包含染色质环,例如增强子—启动子环。
随着Hi-C测序技术逐步成熟,研究人员在染色质相互作用的图谱中,观察到更加精细的染色质相互作用模式,如染色质条带。条带反映了环挤出模型中染色质折叠过程中环形成时的动态情况。环挤出模型提出染色质环的形成是由染色质主动挤出机制介导的,其中CTCF蛋白附着在染色质上并随之在黏连蛋白复合体中滑动,当遇到另一个方向相对的CTCF蛋白时会将染色质固定为环状结构。染色质通过高度有序的多层级空间结构,限制顺式作用元件的相互作用,辅助基因表达调控。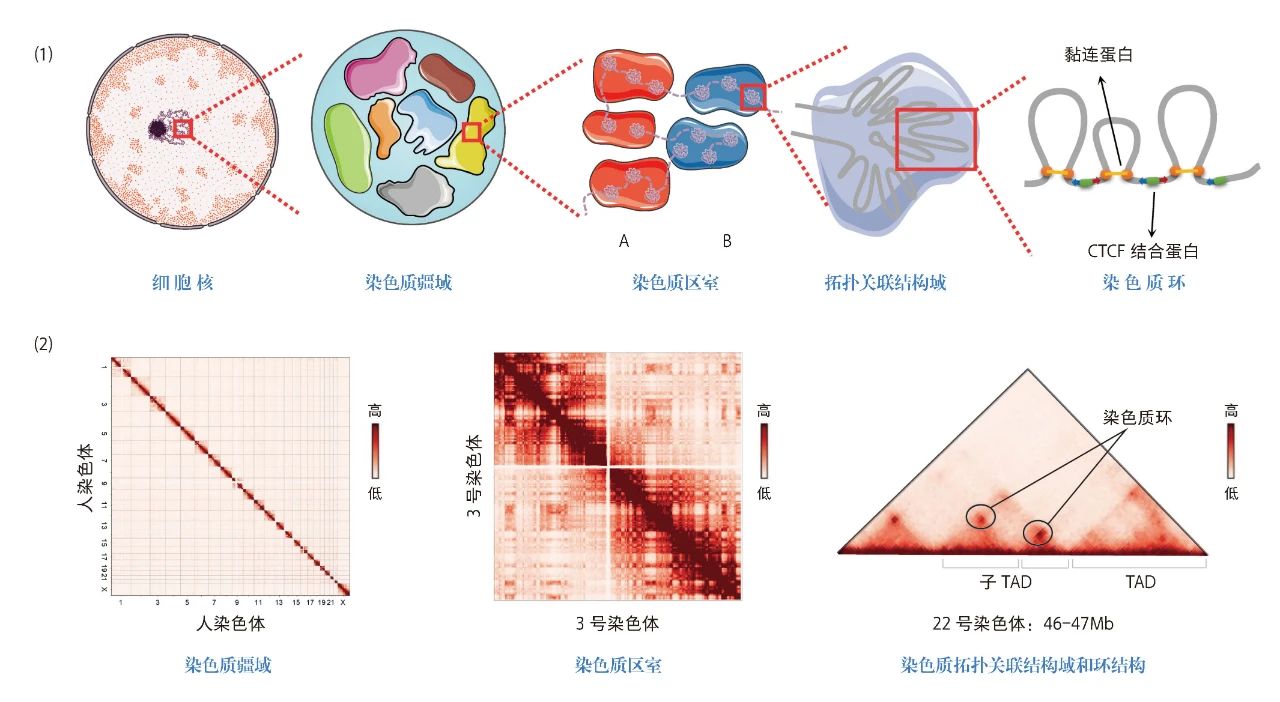
基于Hi-C识别的染色质高级结构 (1)基于Hi-C数据识别的染色质高级结构示意图(A代表转录活跃的染色质区室,B代表转录抑制的染色质区室);(2)不同分辨率尺度的Hi-C矩阵热图。 [4]
不可或缺的计算方法
采用Hi-C技术获取的数据量很大,一个样本可达到百GB级。为了通过Hi-C数据探究染色质的多层级结构,高效精确的计算方法不可或缺。另外,Hi-C技术仍存在分辨率不够、公共数据积累有限等局限性,相应计算工具的开发则对这些不足进行了弥补。
基于Hi-C数据的染色质多层级结构分析方法
Hi-C数据可通过HiC-Pro、HiCExplore等软件工具进行预处理,经过序列比对、数据过滤、图谱构建、数据质控等过程,预处理后的Hi-C数据可以用于染色质区室、拓扑关联结构域和染色质环等结构的识别。目前已有多种计算工具可基于Hi-C数据对染色质的各层级结构进行识别分析,例如识别区室结构的CscoreTools、SNIPER;识别拓扑关联结构域的Arrowhead、Insulation Score、Directionality index、TADbit、OnTAD、MrTADFinder;识别环结构的HiCCUPS、Juicer、Fit-Hi-C等。这些工具的开发使得研究人员可以探寻不同物种基因组的三维结构、机体不同发育阶段染色质高级结构的变化规律以及疾病状态下的染色质结构的变异。
基于序列及表观遗传数据的预测方法
DNA序列特征及表观遗传修饰与染色质结构的形成和变化密切相关,目前已有多个数据库提供了丰富的组织和细胞的序列及表观基因组信息,且有多种计算工具尝试通过序列和表观遗传数据预测染色质结构。笔者团队近期对48种基于DNA序列及表观遗传信息进行染色质相互作用及高级结构预测的计算方法进行了总结、分类及性能比较,并对这些方法在不同生物领域中的应用进行了总结 [4]。其中包括预测染色质环的CISD-loop、CTCF-MP、Lollipop和DeepMILO,预测拓扑关联结构域的CITD、PGSA、TAD-Lactuca、nTDP和BART,预测染色质多聚模型的MiChroM、HiP-HoP、Chromatin states-based model,以及预测Hi-C相互作用图谱的Rambutan、Akita和DeepC。这些工具可用于Hi-C数据缺乏时染色质高级结构的预测。
基于深度学习提升Hi-C数据的分辨率
Hi-C数据的分辨率直接影响染色质相互作用识别的有效性和准确性,目前大多数组织和细胞系仍缺乏高分辨率的Hi-C数据,通过大幅增加测序深度来获取它们成本仍然较高,且随着测序深度增加,指数增长的数据也给计算分析带来了新的挑战。笔者团队开发的DeepHiC通过生成对抗网络,提升Hi-C数据的分辨率,可以对染色质高级结构进行更加精准地预测 [5]。DeepHiC提供了网络运行界面(http://sysomics.com/deephic),用户可以通过网页提交数据,设定参数,计算后得到提升后的Hi-C数据 [5]。
染色质高级结构与发育
细胞分化不同阶段的基因组三维结构会发生动态变化,以此调控相关基因的表达,确保细胞可以正常发育并分化成特定的组织器官。研究表明,染色质高级结构在早期胚胎发育中具有重要作用,并与机体衰老密切相关。此外,在机体生长发育过程中,如果染色质高级结构出现异常,会影响细胞的正常分化,导致疾病的发生。
染色质高级结构与早期胚胎发育
哺乳动物的染色质多层级结构在早期胚胎发育过程中逐渐建立,并发生动态重排,从而调控DNA复制和修复、转录、X染色体失活等过程。染色质高级结构塑造了胚胎发育过程中基因表达的调控密码,调控细胞分化和命运决定。研究者们曾揭示了哺乳动物成熟精子和卵子的染色体3D结构及早期胚胎发育过程中染色体结构的重编程变化,为深入了解哺乳动物早期胚胎发育过程打下了重要基础。他们还揭示了人类早期胚胎中的染色体三维结构的动态变化,并发现CTCF蛋白对于早期胚胎发育中拓扑关联结构域的结构具有重要的调控功能,为进一步揭示人类胚胎发育机制提供了理论基础。此外,还有研究人员发现染色体的三维结构在受精后首先呈现出一种极其松散的状态,并在随后的胚胎早期发育过程中逐步地以亲本特异的方式建立和成熟。他们与其他团队合作通过体细胞核移植技术,发现核移植胚胎发育过程伴随着剧烈的染色体高级结构的重编程,黏连蛋白具有形成拓扑关联结构域和抑制minor ZGA基因的双重功能,为研究早期胚胎发育过程中独特的染色质高级结构的形成机制和功能提供了重要线索。
染色质高级结构与机体发育
染色质高级结构参与机体发育过程中的细胞谱系分化,维持机体的正常发育。染色质高级结构改变会影响基因调控,导致机体发育障碍。已有研究发现,拓扑关联结构域内IHH基因座增强子的复制,会导致该基因表达发生组织特异性失调,与多趾症的发生相关。而SOX9基因座的拓扑关联结构域边界复制异常,会导致新拓扑关联结构域的形成,与烹调综合征和短指等疾病相关。此外,先天性肢体畸形、自身免疫性疾病和性反转等发育相关疾病也被证实与染色质高级结构失调相关。近日,笔者与其他团队合作,开发了开放染色质致密度算法SDOC来表征染色质的结构变化,基于SDOC对T细胞分化过程的研究发现,在T细胞分化过程中,如果拓扑关联结构域的结构发生变化,会抑制其他谱系基因的表达,表明染色质结构参与细胞分化和命运的调控 [6]。
染色质高级结构与衰老
染色质高级结构在机体衰老进程中也发挥着重要的调控作用。研究人员结合多能干细胞定向分化技术、基因组靶向编辑技术,以及表观遗传组分析技术揭示了异染色质的高级结构失序是人类干细胞衰老的驱动力之一,为延缓和防治衰老相关疾病提供了新的潜在靶点和思路。
染色质高级结构与癌症
癌症是一种致死率极高的恶性疾病。在正常细胞转变为肿瘤细胞的多阶段转化过程中,基因组常发生各种突变,如点突变、小片段的插入和缺失、染色质拷贝数变异和重排等。这些突变可能会造成基因组三维结构变异,如染色质区室转换、拓扑关联结构域及染色质环结构改变等,这些变异可能会导致原癌基因或抑癌基因表达异常,使细胞表现出永生化等恶性特征 [7]。随着有关癌症与染色质空间结构变异关系研究的深入,以染色质高级结构为靶点的癌症治疗策略逐渐成为研究热点。
染色质高级结构变异与癌症
近日,有实验室整合分析了结肠癌和正常结肠的高通量染色体构象捕获技术数据、转录组测序(RNA sequencing,RNA-seq)及表观遗传数据,发现结肠癌中基因组区室发生了广泛重组,A区室和B 区室之间存在重组的中间区室。在正常细胞中,中间区室更趋近于A区室,而在癌细胞中,中间区室普遍处于低甲基化状态,更趋近于B区室 。通过结直肠肿瘤队列的数据分析发现,区室变化相关的转录特征可以用于预测患者的预后和转移。
染色质拓扑关联结构域作为染色质空间结构的基本单位,其结构变异会导致染色质相互作用的改变,从而促使新的调控元件和基因接近,影响基因表达,参与细胞恶化过程。笔者团队对骨髓瘤细胞与正常B细胞的高通量染色体构象捕获技术数据、全基因组测序数据及RNA-seq数据进行整合分析发现,与正常细胞相比,骨髓瘤细胞中拓扑关联结构域的数量增加,平均长度减小。基因组中的染色质拷贝数变异断点经常分布在拓扑关联结构域的边界,提示拓扑关联结构域边界的拷贝数变异位点更容易发生DNA链断裂或被癌细胞克隆选择 [8]。此外,还有研究团队通过T 细胞急性淋巴细胞白血病(T cell acute lymphoblastic leukemia, T-ALL)的研究发现,T-ALL中染色质拓扑关联结构域的结构变异,导致T-ALL重要致病基因MYC的异常表达。
此外,还有研究团队发现了脉络膜黑色素瘤特异性染色质环结构,使神经降压素基因的启动子区域与上游800 kb处的增强子相互作用,导致神经降压素的异常高表达,促进了脉络膜黑色素瘤细胞的增殖和迁移。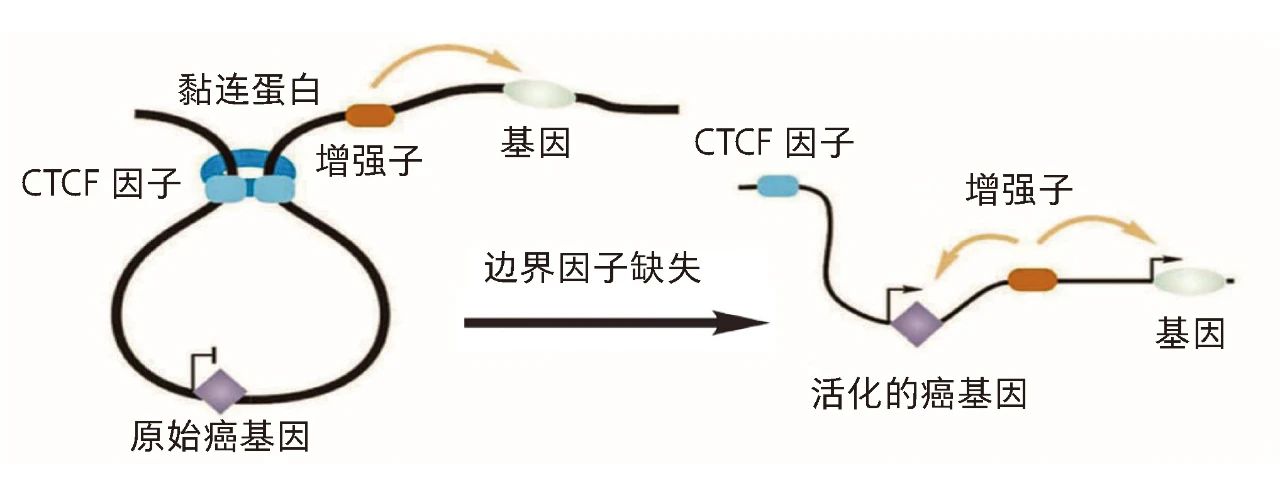
癌症中染色质三维结构的破坏导致基因表达异常 [7]
基于三维基因组的癌症治疗
随着Hi-C及其衍生技术的逐步成熟,染色质高级结构与疾病的关系逐渐被发现,三维基因组也成为了抗癌治疗的潜在靶标。笔者团队整合了公共Hi-C数据和染色质免疫共沉淀测序数据,构建了评价疾病相关染色体重排对基因组三维结构影响的统计学方法,筛选出潜在由于三维结构改变导致疾病发生的染色体重排数据,并开发了3Disease Browser网站(http://3dgb.cbi.pku.edu.cn/disease/)用于集成和可视化疾病相关染色体重排与邻近的染色体三维结构 [9]。另外,笔者团队还结合三维基因组、千人基因组项目、表观遗传修饰和基因组序列特征,建立非编码变异与其相互作用靶基因之间关联的数据库和网站3DSNP(http://www.cbportal.org/3dsnp/) [10]。
近年来,以染色质三维结构为靶标的抗癌疗法逐渐被开发。有实验室开发了一种癌症治疗策略——染色质保护疗法,从染色质整体构象出发,通过改变染色质的包装密度,阻止癌症的适应性。在使用具有改变染色质包装密度作用的塞来昔布和地高辛两种药物联合化学药物处理癌细胞后,研究人员发现癌细胞在两三天几乎被全部杀死,表明通过药物靶向染色质高级结构的方法改变细胞适应性,联合现有癌症疗法能够有效杀死癌细胞。另外,另一研究团队基于Hi-C、RNA-seq和CTCF的ChIP-seq数据,发现了人类急性白血病中的染色质三维结构变化,并证明靶向NOTCH1信号通路的γ-内分泌酶抑制剂可以改变白血病中发现的特定染色质3D相互作用。
染色质构象捕获技术结合相关计算工具的开发,极大地促进了对染色质三维空间结构及其在基因表达调控、细胞增殖分化、机体发育及疾病发生发展中作用机制的理解。染色质高级结构的研究将推动人们进一步探索染色质高级结构失调与疾病发生发展的关系,为癌症等疾病的诊疗提供潜在靶点。
参考文献:
- Lieberman-Aiden E, van Berkum N, Williams L, et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science, 2009, 326(5950): 289-293.
- Li T, Jia L, Cao Y, et al. OCEAN-C: mapping hubs of open chromatin interactions across the genome reveals gene regulatory networks. Genome Biology, 2018, 19(1): 54.
- Zhang C, Xu Z, Yang S, et al. tagHi-C reveals 3D chromatin architecture dynamics during mouse hematopoiesis. Cell Reports, 2020, 32(13): 108206.
- Huan Tao H L, Kang Xu, Hao Hong, et al. Computational methods for the prediction of chromatin interaction and organization using sequence and epigenomic profiles. Briefings in Bioinformatics, 2020.
- Hong H, Jiang S, Li H, et al. DeepHiC: A generative adversarial network for enhancing Hi-C data resolution. PLoS Computational Biology, 2020, 16(2): e1007287.
- Jiang S, Li H, Hong H, et al. Spatial density of open chromatin: an effective metric for the functional characterization of topologically associated domains. Briefings in Bioinformatics, 2020.
- Li R, Liu Y, Hou Y, et al. 3D genome and its disorganization in diseases. Cell Biology and Toxicology, 2018, 34(5): 351-365.
- Wu P, Li T, Li R, et al. 3D genome of multiple myeloma reveals spatial genome disorganization associated with copy number variations. Nature Communications, 2017, 8(1): 1937.
- Li R, Liu Y, Li T, et al. 3Disease Browser: A Web server for integrating 3D genome and disease-associated chromosome rearrangement data. Scientific Reports, 2016, 6: 34651.
- Lu Y, Quan C, Chen H, et al. 3DSNP: a database for linking human noncoding SNPs to their three-dimensional interacting genes. Nucleic Acids Research, 2017, 45(D1): D643-d649.

