摘自:白墨,知乎【https://zhuanlan.zhihu.com/p/166500744】
一、什么是染色质可及性?
染色质关闭:压缩DNA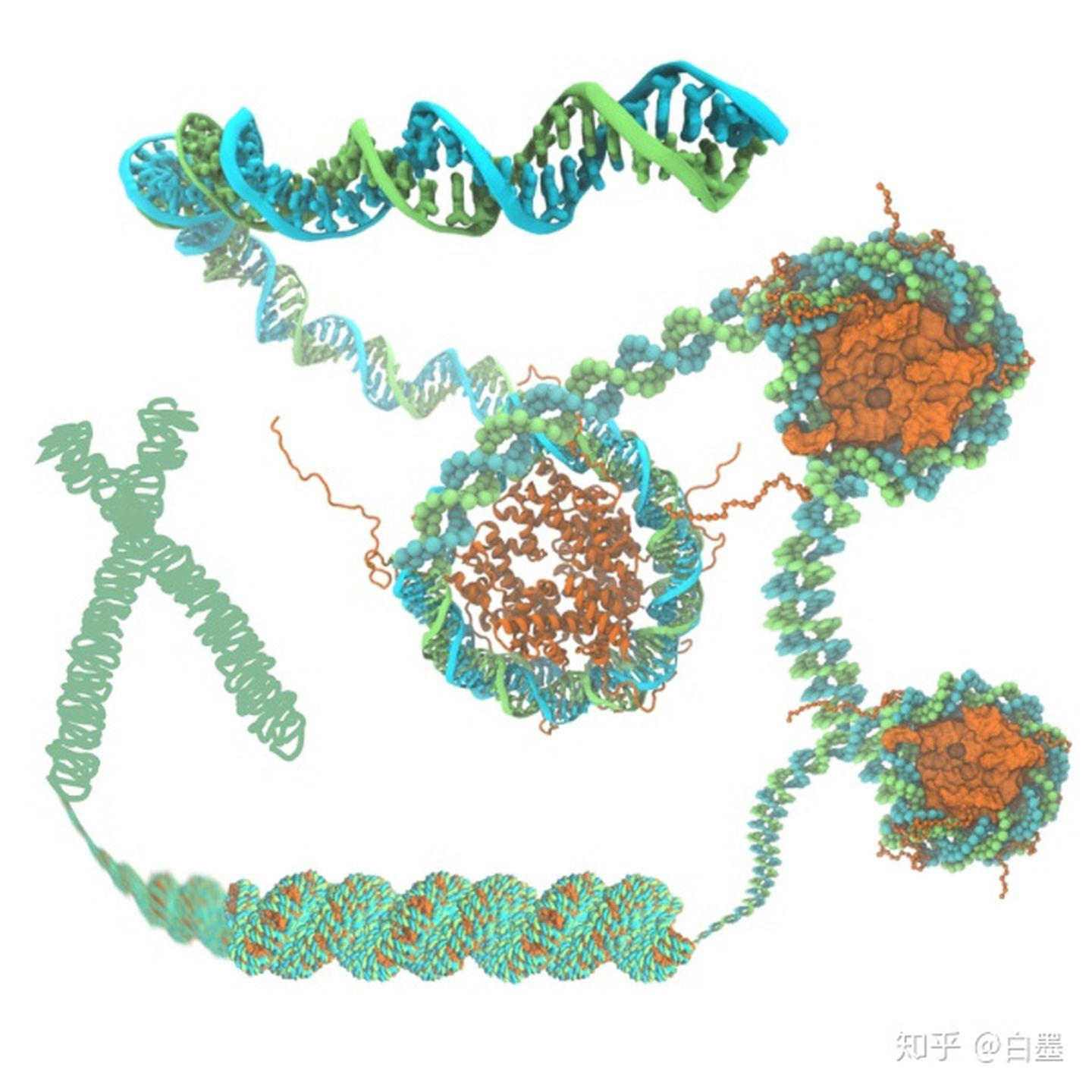
人的DNA链全部展开大约有2m,需要折叠为染色质结构才可以存储到放到细胞核中。染色质的基本结构单位是核小体(由组蛋白组成),核小体再折叠最终形成高度压缩的染色质结构。一般真核生物是这种方式来存储遗传信息。
这个过程像我们将文件压缩为zip或者rar的压缩包,减少它的占用空间。
染色质开放:解压DNA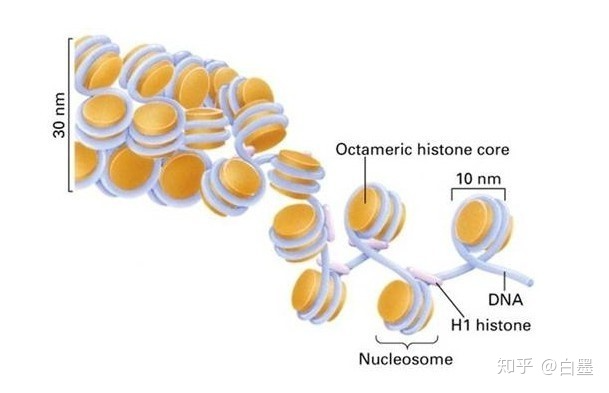
高度折叠的染色质结构在复制和转录时需要暴露出DNA序列,这段暴露的区域就是染色质开发区域,这个区域可以供转录因子和其他调控元件结合,所以它与转录调控是密切相关的。这种致密的核小体结构被破坏后,启动子、增强子、绝缘子、沉默子等顺式调控元件和反式作用因子可以接近的特性,叫染色质的可及性,也叫染色质开放性(chromatin accessibility ),这段区域叫开放染色质(open chromatin)。
这个过程类似于我们要查看刚刚压缩包里的文件,我们需要解压后才能查看到文件里的内容。
二、检测染色质可及性
为了研究染色质的这种特性,大家都先后尝试了好多测序来检测染色质可及性。但是目前最常用的是2013年由斯坦福大学William J. Greenleaf和Howard Y. Chang实验室开发的ATAC-seq(Assay for Transposase-Accessible Chromatin with high throughput sequencing),一种捕获染色质可及性(染色质开放性)的测序方法。
1. 原理
利用转座酶Tn5可结合开放染色质的性质,使用Tn5酶捕获DNA序列,进而上机测序。
转座酶会携带特定的已知序列,然后将这些序列插入到开放的染色质区域中,最后将带有转座酶标记过的序列上机测序,通过软件计算,就能获得基因组哪些地方是开放的。
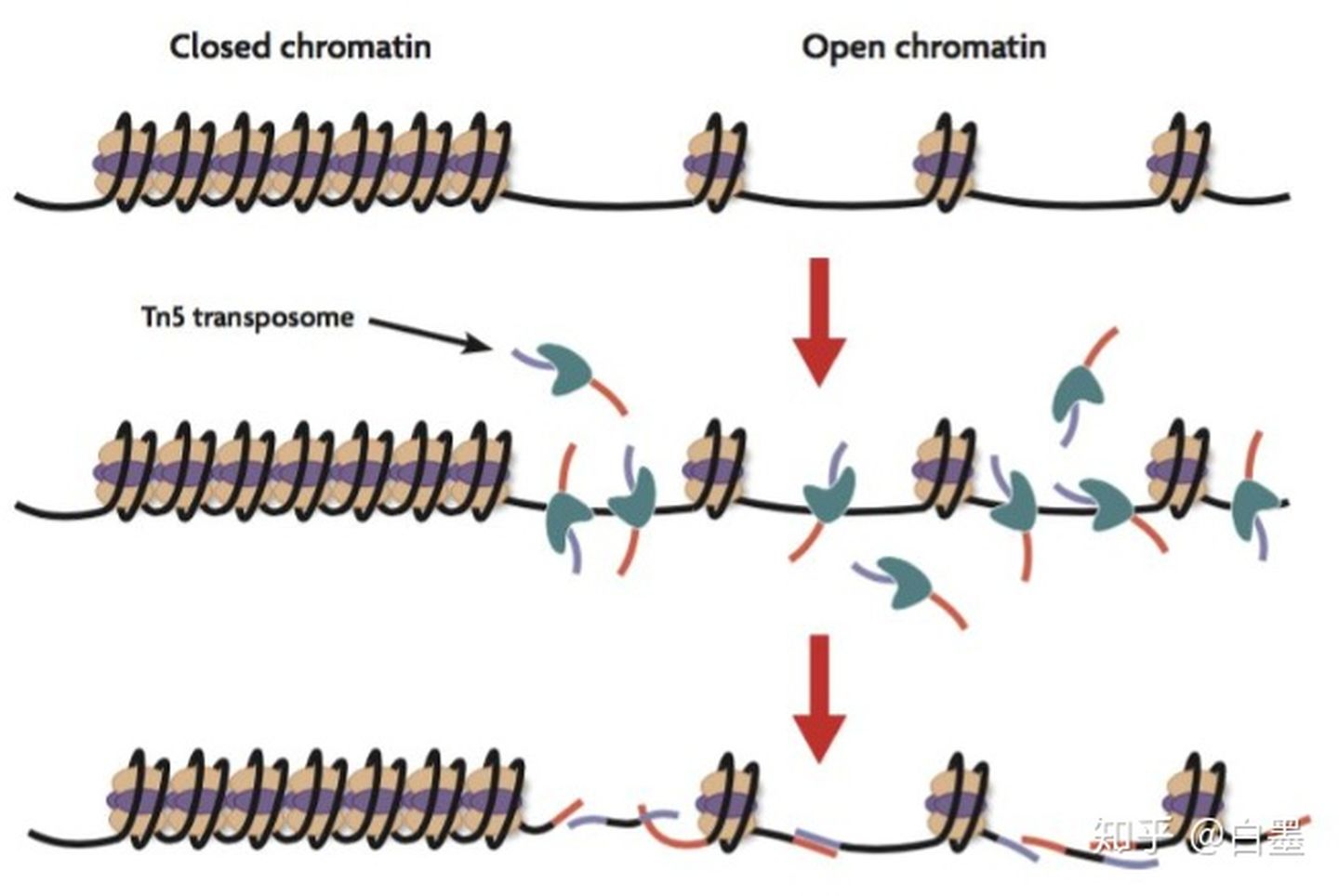
开放染色质的研究方法除了ATAC-seq,还有DNase-Seq,FAIRE-seq,MNase-seq 等。ATAC-Seq由于其所需样本少,建库快,重复性更高,是目前研究开发染色质的主流技术方法。
2. 应用
- 染色质开放性图谱绘制
- 找调控生物学过程的关键转录因子
- 找哪个转录因子调控了研究的基因
-
3. 技术限制
Tn5通过插入剪断DNA 并将测序接头连接到剪断的两个DNA 片段的末端,因此对于一个DNA 片段而言,其两端的接头连接是随机的,这便导致同一片段两端的接头有50%的概率是同一接头。而只有连接不同接头的片段才可用于富集扩增及测序,因此,有一半的片段无法利用;
- 大量剪断的DNA 由于片段过大,无法进行PCR富集;
- Tn5 的活性受反应溶液的组成及反应条件影响,仍然需要优化以便提高剪切效果;
ATAC-seq在植物细胞中存在以下难点:细胞壁的存在,叶绿体、线粒体等细胞器的污染,缺少稳定遗传的细胞系;
三、相似测序方法的异同
1. ATAC-Seq、ChIP-Seq、Dnase-Seq、MNase-Seq、FAIRE-Seq 比较
这些测序方法整体的分析思路一致,找到富集区域,对富集区域进行功能分析。
ChIP-Seq是揭示特定转录因子或蛋白复合物的结合区域,实际是研究DNA和蛋白质的相互作用,利用抗体将蛋白质和DNA一起富集,并对富集到的DNA进行测序。
DNase-Seq、ATAC-Seq、FAIRE-Seq都是用来研究开放染色质区域:
DNase-Seq是用的DNase I内切酶识别开放染色质区域,
ATAC-seq是用的Tn5转座酶,随后进行富集和扩增;
FAIRE-Seq是先进行超声裂解,然后用酚-氯仿富集;
MNase-Seq是用来鉴定核小体区域。
下图是不同测序方法获取的峰形: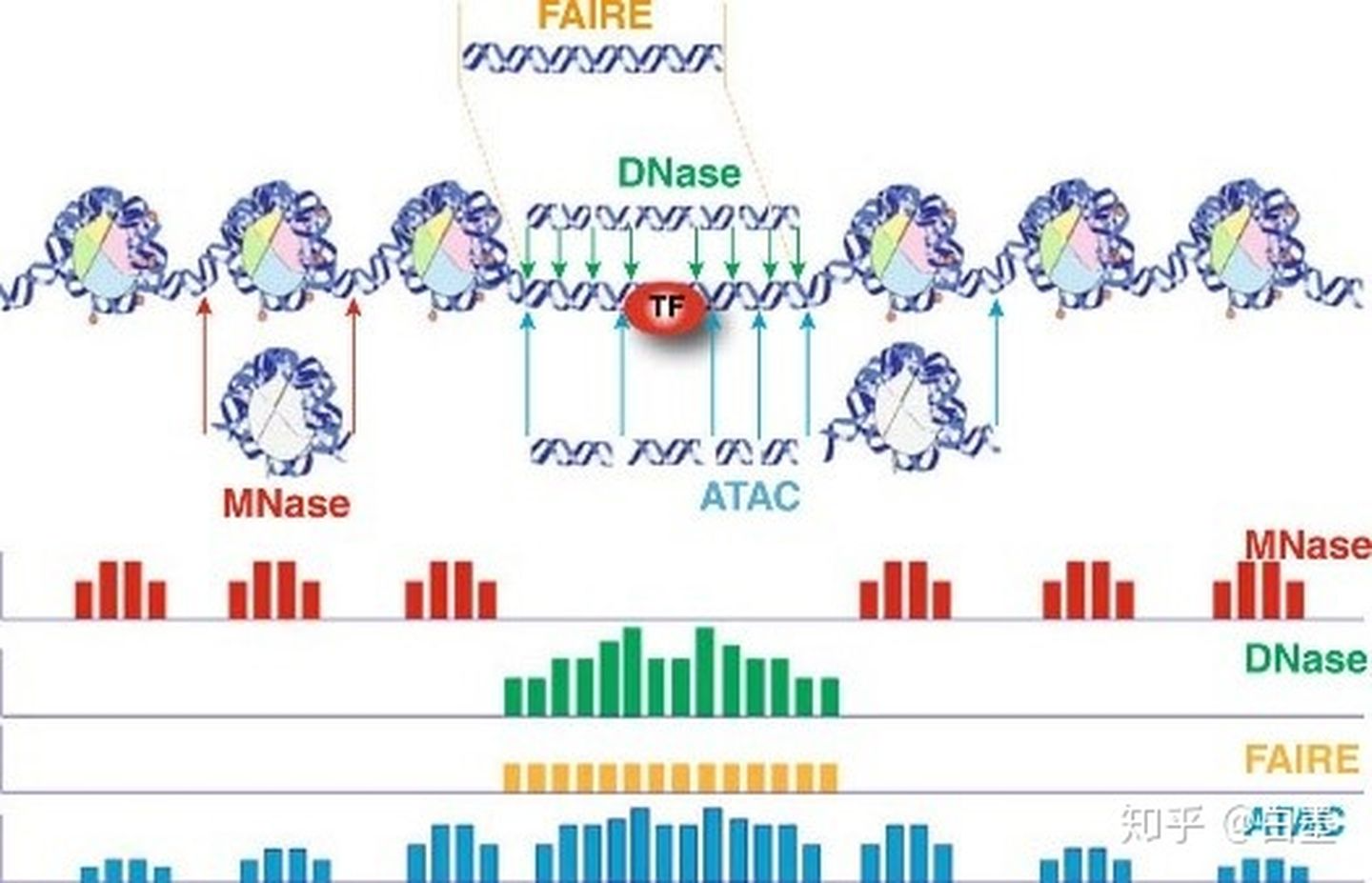
2. ChIP-seq 与 ATAC-seq 的比较
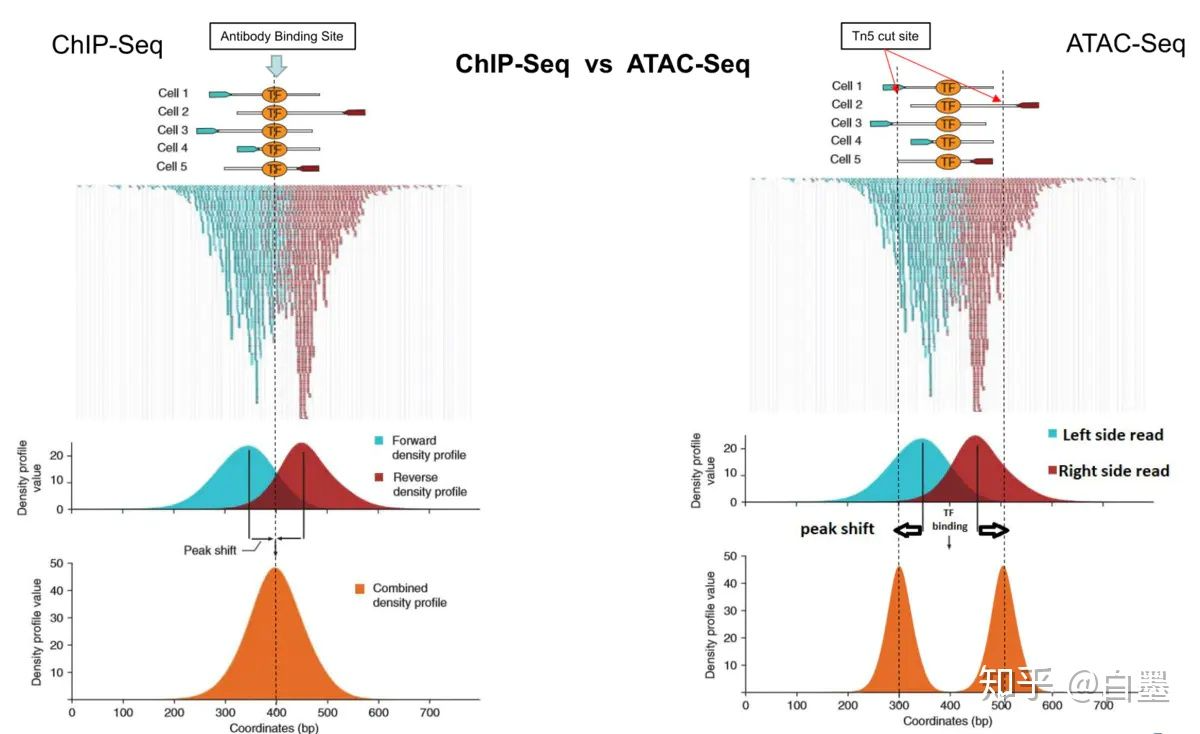
峰形状的区别
Chip-seq与ATAC-seq 的 peaks 有着明显的区别,前者peaks是代表抗体结合转录因子的位点,后者peaks是代表Tn5转座酶切开染色质开放区的两端,因此在一个位置,前者peaks有一个,后者有两个。
- 在应用上的区别
ATAC-Seq是可以检测到全基因组的DNA结合蛋白,转录结合位点 ,一般用于不知道特定的转录因子,用此方法与其他方法结合筛查感兴趣的特定调控因子;
ChIP-Seq是已知转录因子是什么,根据感兴趣的转录因子设计抗体去做ChIP实验富集它结合的DNA片段。在测定转录因子的 ChIP-seq 中独有的峰可能是先驱转录因子,其先结合到封闭染色质,然后招募染色质重塑因子或其他转录因子来起始转录。 这些转录因子 ATAC-seq是检测不到的。
- 整合分析
由于开放染色质是大多数TF结合的先决条件,因此ATAC-seq峰通常与TF ChIP-seq峰重叠,但通常更宽。因此,TF ChIP-seq和ATAC-seq可以在同一实验系统中相互验证彼此的质量和可靠性。
ATAC-seq与 histone marker ChIP-seq集成,发现与活跃染色质标 H3K4me3,H3K4me1,H3K27ac等正相关,与不活跃的染色质标记 H3K27me3 负相关。
参考:
- https://www.plob.org/article/13950.html
- https://www.zhihu.com/question/263776928
- Identifying and mitigating bias in next-generation sequencing methods for chromatin biology
- https://github.com/hbctraining/In-depth-NGS-Data-Analysis-Course/blob/master/sessionV/lessons/01_Intro_chipseq_and_setup.md
- https://pubmed.ncbi.nlm.nih.gov/24097267/#
- https://www.nature.com/articles/s41576-018-0089-8

