摘自:白墨,知乎【https://zhuanlan.zhihu.com/p/279354841】
一、介绍
- ChIP-seq,测序方法
- ChIP 指染色质免疫共沉淀技术(Chromatin Immunoprecipitation,ChIP),
- seq 指的是二代测序方法
- 作用:识别蛋白质与DNA互相作用情况
- 原理:染色质免疫共沉淀 + 二代测序
-
二、测序原理
1、使用甲醛将目标蛋白与染色质交联固定起来
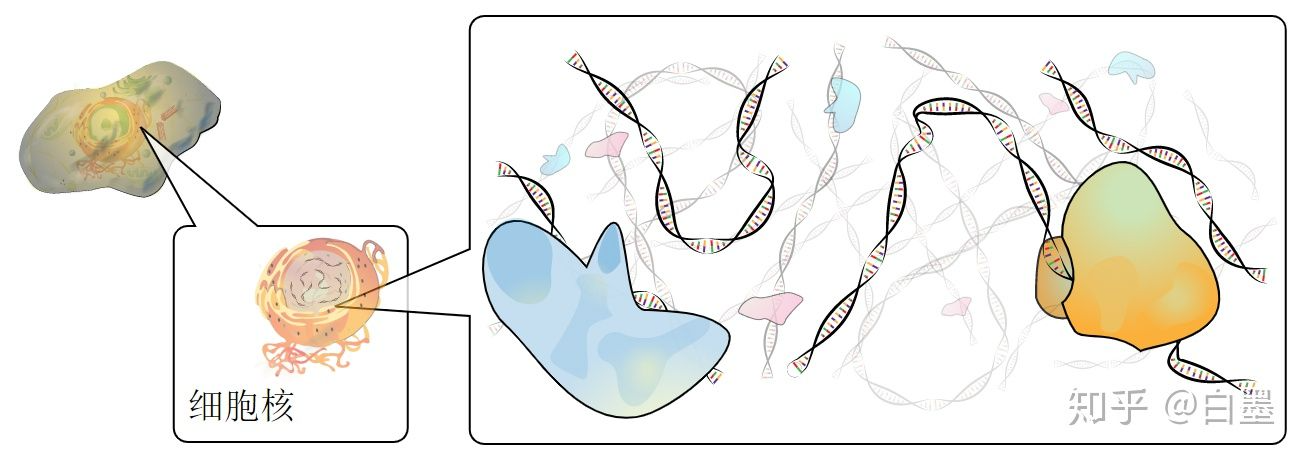
2、从细胞裂解液分离基因组DNA,通过超声打断DNA为一定长度的小片段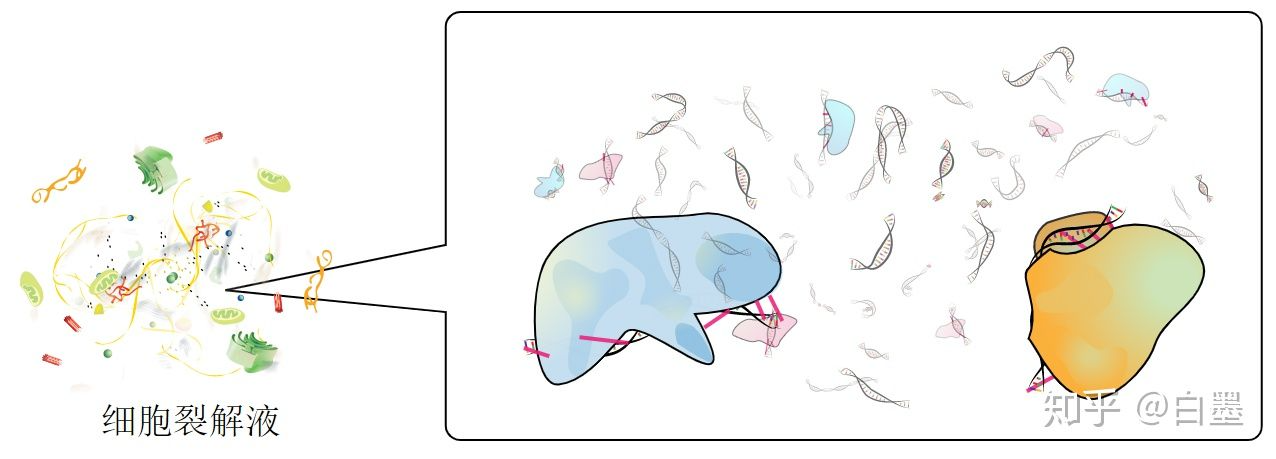
3、添加与目标蛋白质特异的抗体,该抗体与目标蛋白形成免疫沉淀免疫结合复合体
4、去交联,纯化DNA即得到染色质免疫沉淀的DNA样本,准备测序
5、建立好文库,用测序仪进行测序
详细测序过程可以参考:二代测序原理
三、检测蛋白质与DNA序列的结合峰
1、将测序得到的 DNA 片段(sequenced fragments)匹配到参考基因组
很显然,如果在基因组的某个位置蛋白质结合的概率越大,那么该位置检测到的 DNA 片段堆叠的就会越高。

2、使用空白对照(control)
为什么需要对照组?
一般检测出的峰值会有背景噪音,也就是会夹渣一些没有用抗体捕获的DNA片段也被测序了。
- 开放的染色质区域比封闭的区域更容易断裂
- 序列标签在基因组中分布不均
- 允许我们在比对的控件中与相同区域进行比较
- 消除 ENCODE 提供了 Black list的影响
所以会准备空白对照,排除假阳性,对照组有有两种:
- input DNA:不用任何抗体捕获的DNA;
- mock IP DNA:用不含有抗体的DNA
利用对照组去除实验组中的背景噪音,就会让我们检测到的峰更明显。
3、将覆盖到参考基因组的DNA片段堆叠用柱状图画出来,就会看到峰。
这里需要知道,ChIPseq是可以检测转录因子的结合,也可以检测组蛋白修饰的。而且二者有着截然不同的峰形:
转录因子结合的特征峰: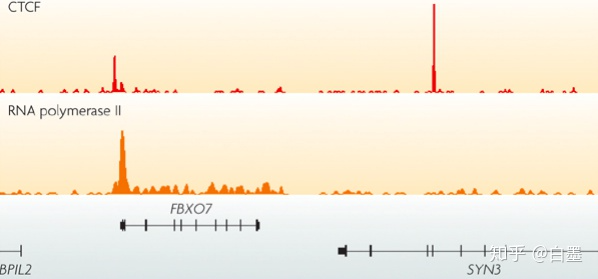
组蛋白修饰结合的特征峰: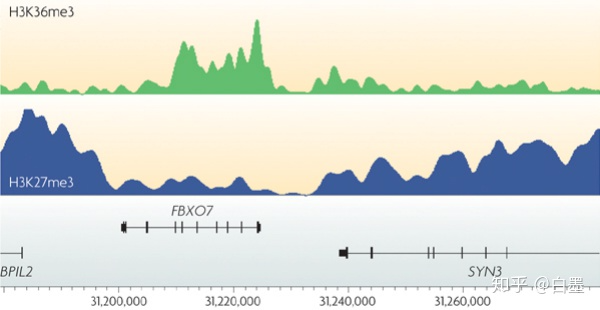
当然我们也可以使用,UCSC基因组浏览器显示。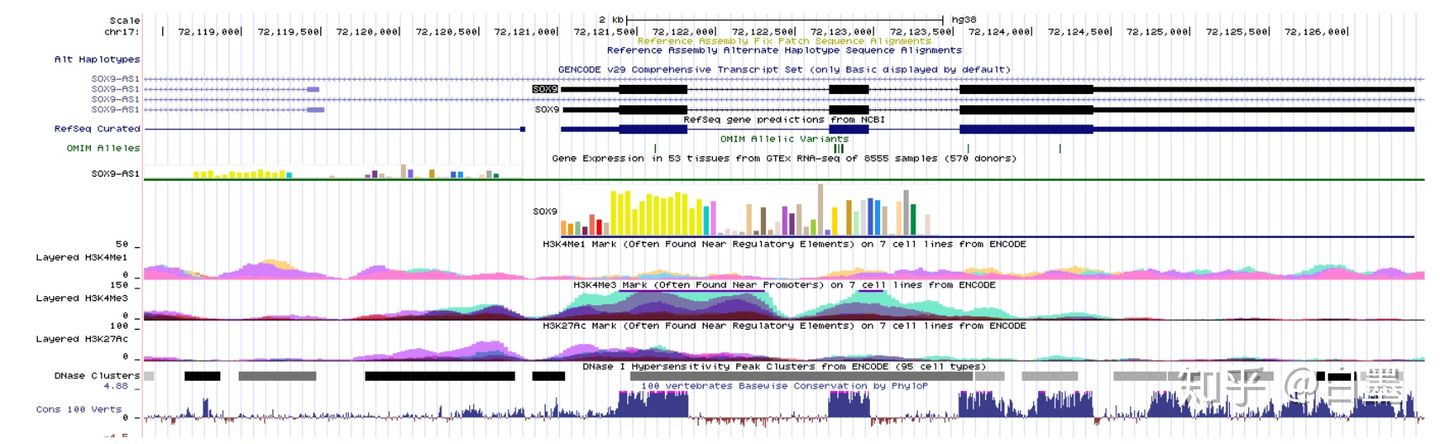
四、影响测序结果的因素
1、免疫共沉淀的影响
- 高效特异性抗体
- 起始物料量
- ChIP DNA产量取决于多种因素
- 细胞类型
- 标记或蛋白质丰富程度(组蛋白比TF具有更高的结合覆盖率)
抗体质量
对于组蛋白,使用来自T细胞的20ug染色质DNA作为起始材料,总共会得到15-50ng DNA。
对于TF,通常从2500万个细胞(200ug染色质)中得到5-25ng。
-Subhash Tripathi,ResearchGate染色质片段
- 片段大小:影响ChIP-seq中的信噪比
- 因细胞类型而异
- 偏向启动子区域的片段会在ChIP AND对照(输入)样品中的启动子上引起ChIP-seq富集
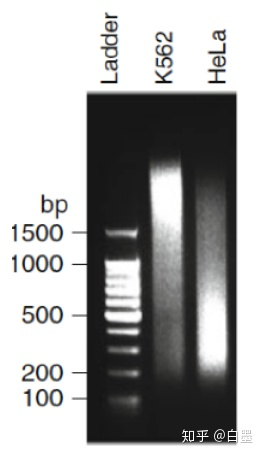
2、测序的影响
- Reads 长度
- 较长的 Reads 和双末端 Reads 可提高匹配率
- 仅对于等位基因特异性染色质事件,转座因子研究而言是必需的
- 避免分批次
- 测序深度(最小5-10M;对于转录因子,标准为20-40M;对于组蛋白修饰宽谱图则更高)
-
3、测序深度的影响
H3K4me3

H3K27me3
从每个子样本中H3K4me3,H3K36me3和H3K27me3回收的全部数据中获得的显著富集区域的百分比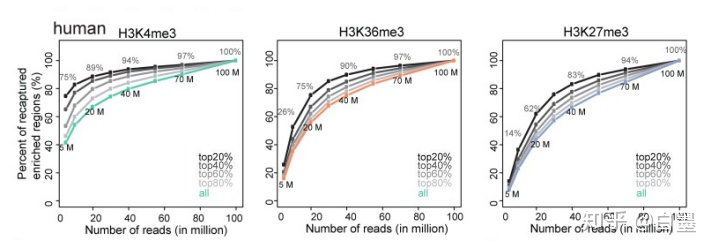
对另外100万条 Reads 进行测序时,捕获的富集区域增加的百分比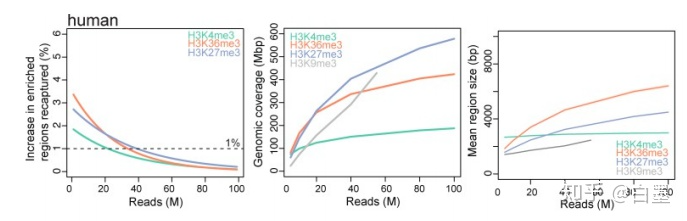
4、重复样和重现性
重复多次通常比更高的深度更有效
- 最好低深度测序高质量样本,而不是高深度低质量样本
参考:
https://academic.oup.com/nar/article/42/9/e74/1248114
https://en.wikipedia.org/wiki/ChIP_sequencing#/media/File:Chromatin_immunoprecipitation_sequencing.svg
https://www.abcam.com/epigenetics/studying-epigenetics-using-chip

