在准备NGS文库的时候,会有用到转座酶Tn5,Nextera DNA Library Preparation Kit ,比如ATAC-seq就有用到这个Tn5。转座酶携带有特定的序列称为转座子Transposon。
下面是ATAC-seq的工作原理,想必听说过ATAC-seq的,对这个图都会再熟悉不过了。
Tn5是kit里面带有的,没什么大不同。ATAC-seq 的barcoded primers (adaptors)会有一些不同,接下来,看下图中的每段序列对应的sequnce都是什么。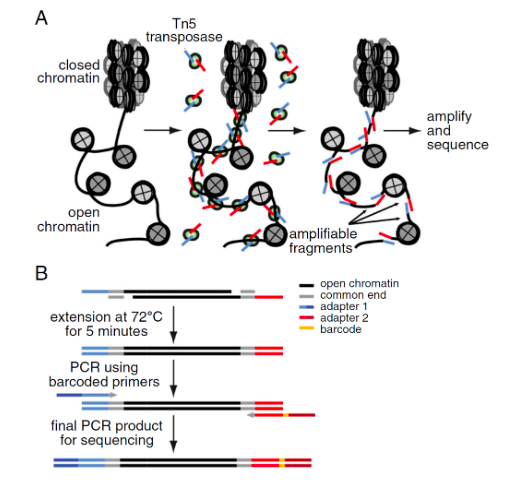
下面图例,Nextera tn5用来给基因组DNA加adaptors。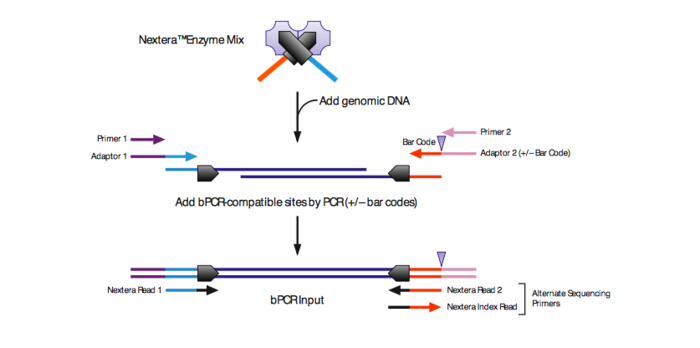
放大Tn5:
在tn5二聚体里面的双链的Oligo是Nextera Tn5 binding site,19个碱基,全部都是一样的。伸出来的部分是单链的序列,蓝色:Nextera tn5 read1 (5’-TCGTCGGCAGCGTC-3’);红色:Nextera tn5 read2(5’-GTCTCGTGGGCTCGG-3’)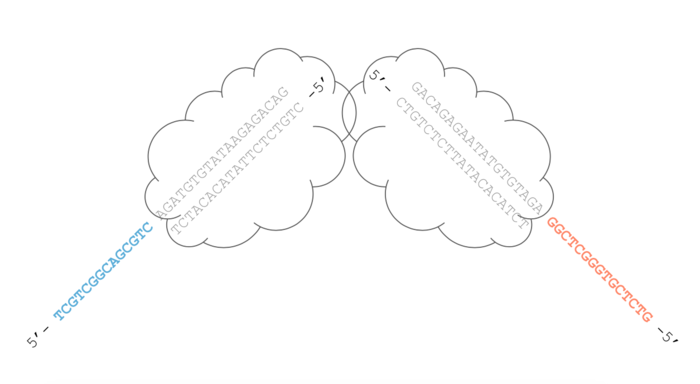
Nextera Tn5 binding site (19-bp Mosaic End (ME)): 5'- AGATGTGTATAAGAGACAG -3’Nextera N/S5xx primer entry point (s5)/Tn5 Read 1: 5'- TCGTCGGCAGCGTC -3'Nextera N7xx primer entry point (s7)/ Tn5 Read 2: 5'- GTCTCGTGGGCTCGG -3'Illumina P5 primer 1: 5'- AATGATACGGCGACCACCGAGATCTACAC -3'Illumina P7 primer 2: 5'- CAAGCAGAAGACGGCATACGAGAT -3’Tn5 Read 1 (包括ME)sequence: 5'- TCGTCGGCAGCGTC(AGATGTG,TATAAGAGACAG) -3’ (33bp)Tn5 Read 2 (包括ME)sequence: 5'- GTCTCGTGGGCTCGG(AGATGT,GTATAAGAGACAG )-3’ (34bp)
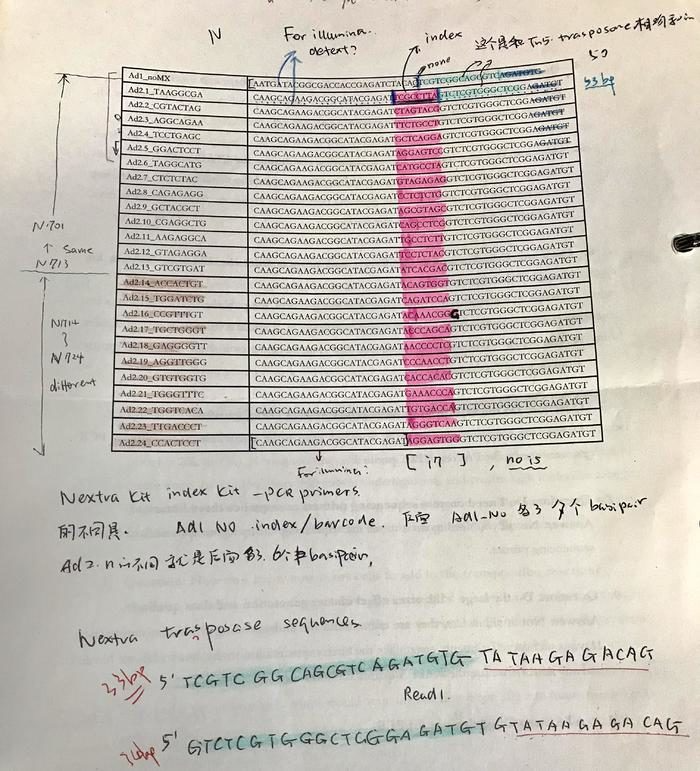
下面的图片就是准备ATAC-seq时候需要用到的index primer (adaptor),和Nextera DNA library prep kit里的有一点类似,但是也很不同。Ad1_noMX是Forward 引物,没有index (barcode),所以只有一个。Reversed引物有24个Ad2.(1-24),所以有24组不同的index (barcode)序列。这个index序列是和Nextera一样的。我涂红色部分就是index序列的反向反义序列,每个index序列是不同的8个碱基,这个和Nextera的i7(部分一样,请自己对照)是一样的。
i5 sequence: ATAC-seq的Adaptor1 里没有barcode
N/S502 : CTCTCTATN/S503 : TATCCTCTN/S505 : GTAAGGAGN/S506 : ACTGCATAN/S507 : AAGGAGTAN/S508 : CTAAGCCTN/S510 : CGTCTAATN/S511 : TCTCTCCGN/S513 : TCGACTAGN/S515 : TTCTAGCTN/S516 : CCTAGAGTN/S517 : GCGTAAGAN/S518 : CTATTAAGN/S520 : AAGGCTATN/S521 : GAGCCTTAN/S522 : TTATGCGA
i7 sequence:
N701 : TCGCCTTAN702 : CTAGTACGN703 : TTCTGCCTN704 : GCTCAGGAN705 : AGGAGTCCN706 : CATGCCTAN707 : GTAGAGAGN710 : CAGCCTCGN711 : TGCCTCTTN712 : TCCTCTACN714 : TCATGAGCN715 : CCTGAGATN716 : TAGCGAGTN718 : GTAGCTCCN719 : TACTACGCN720 : AGGCTCCGN721 : GCAGCGTAN722 : CTGCGCATN723 : GAGCGCTAN724 : CGCTCAGTN726 : GTCTTAGGN727 : ACTGATCGN728 : TAGCTGCAN729 : GACGTCGA
1. 所以,ATAC-seq的这个Oligo designs (Adaptors)可以大概总结:
有两种Adaptors,分别为Adaptor1 (50bp) 和Adaptor2 (53bp)。
其中Adaptor1(5’ illumina Primer1 sequence (29bp)+ Tn5 Read1 sequence(14bp)(再延伸到ME里面7bp)3’),Adaptor2(5’ illumina Primer2 sequence(24bp)+ barcode sequence (8bp)+ Tn5 Read2 sequence (15bp)(再延伸到ME里面6bp)3’)
这样算来,ATAC-seq library的长度=左面(Adaptor1 长度 +剩余部分Tn5的ME(12 bp_TATAAGAGACAG))+ open chromatin 的DNA长度+右面(剩余部分Tn5的ME(13bp_GTATAAGAGACAG)+ Adaptor2 长度)=open chromatin 的DNA长度 + 128bp。
所以,在ATAC-seq的libraries中 mono nucleosome的ATAC-seq library长度大概是(单核小体146bp+核小体free region)+两端的adaptor及Tn5最里面部分ME序列长度(50bp+12bp+13bp+53bp)=274bp+free region,也就是bioanalyzer里面看到的第二个peak。图中显示大概是340bp左右。所以free region应该是330bp-274bp=56bp 。
那么会问,对不对呢?看下bioanalyzer的第一个peak,显示是182bp,这个peak代表的是nucleosome free region的ATAC-seq library,所以是nucleosome free region序列长度 + 两端的adaptor及Tn5部分ME序列长度(50bp+12bp+13bp+53bp)=182, 所以,不难得出同样的结果,nucleosome free region序列长度是54bp。差不多哦
以此类推,第三个peak代表de-nucleosome , 500bp到550 bp之间,是不是等于两个核小体长度加上核小体空隙序列再加上两个adaptor, 2_146bp + 2_55bp + 128bp= 530bp (为什么55bp也要乘2?答:多出来一个核小体当然就多出来一份核小体空隙)
综上所述,ATAC-seq library长度比genome DNA实际长度一定多出128bp。
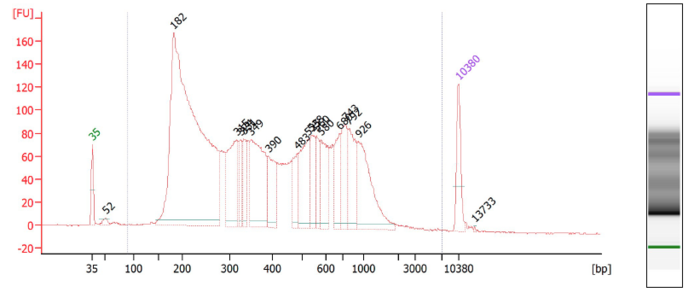
nucleosome free region, mono-nucleosome, de-nucleosome and try-nucleosome
2. 下面一个问题,tagmentation时候,会出现gap?Nextera tagmentation on amplified cDNA (will create 9-bp gap)?
“GAP不是服装品牌,而是个坑”。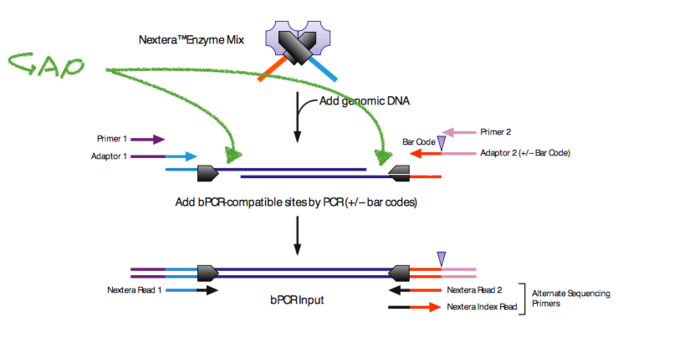
所以需要在PCR第一步,需要5min的72摄氏度来填坑。还要注意,就是在tagmentation的时候会出现三种产物,上面的图只是其中一种。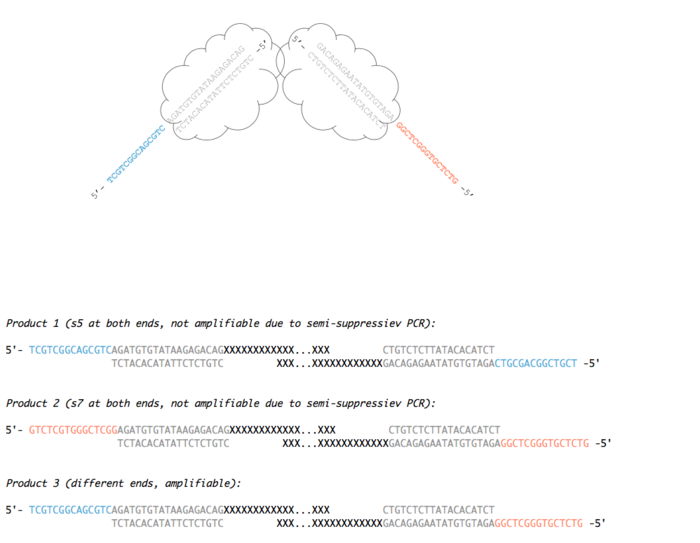
哪里说的不对欢迎纠正,请留言。
参考:
http://ecoliwiki.net/colipedia/index.php/Transposon_Tn5
http://nextgen.mgh.harvard.edu/attachments/Nextera%20Protocol.pdf
https://teichlab.github.io/scg_lib_structs/SMART-seq_family.html
http://www.epibio.com/docs/default-source/protocols/ez-tn5-transposase.pdf?sfvrsn=4- 转载请务必保留本文链接:https://www.plob.org/article/11443.html