概述
染色质免疫共沉淀技术( Chromatin Immunoprecipitation , ChIP )也称结合位点分析法,是研究体内蛋白质与 DNA 相互作用的有力工具,通常用于转录因子结合位点或组蛋白特异性修饰位点的研究。将 ChIP 与第二代测序技术相结合的 ChIP-Seq 技术,能够高效地在全基因组范围内检测与组蛋白、转录因子等互作的 DNA 区段。
ChIP-Seq 的原理是:首先通过染色质免疫共沉淀技术( ChIP )特异性地富集目的蛋白结合的 DNA 片段,并对其进行纯化与文库构建;然后对富集得到的 DNA 片段进行高通量测序。研究人员通过将获得的数百万条序列标签精确定位到基因组上,从而获得全基因组范围内与组蛋白、转录因子等互作的 DNA 区段信息。
技术路线
1 .实验流程( Solexa )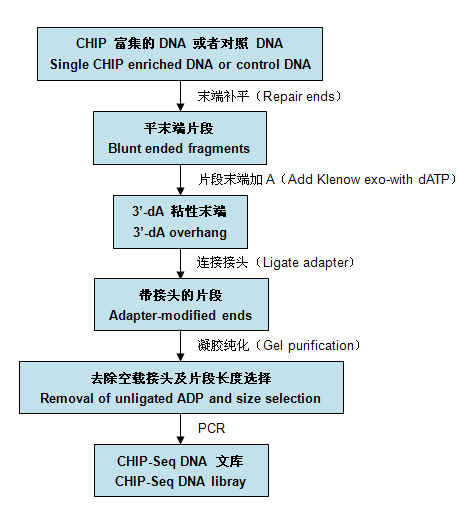
2. 生物信息分析流程示意图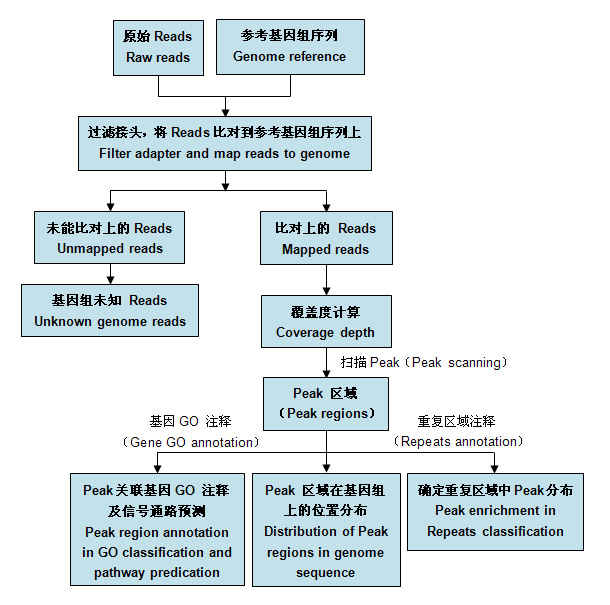
研究内容
1 .测序
对客户提供的 ChIP 样品(如果有阴阳参启动子区域或 DNA 序列的)进行定量检测,检测合格后进行测序文库构建、 DNA 成簇( Cluster generation )扩增、高通量测序。
2 .基本数据分析数据产出统计:对测序结果进行图像识别( Base calling ),去除污染及接头序列;统计结果包括:测定的序列( Reads )长度、 Reads 数量、数据产量。
3. 高级数据分析
标准高级数据分析内容包括:
( 1 ) ChIP-Seq 序列与参考序列比对;
( 2 ) Peak calling :统计样品 Peak 信息(峰检测及计数、平均峰长度、峰长中位数);
( 3 )统计样品 Uniquely mapped reads 在基因上、基因间区的分布情况及覆盖深度;
( 4 )给出每个样品 Peak 关联基因列表及 GO 功能注释;
( 5 )在多个样品间,对与 Peak 关联基因做差异分析。
技术特点
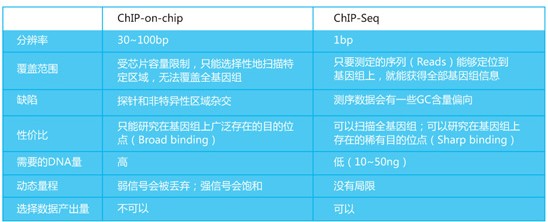
应用领域
由于 ChIP-Seq 的数据是 DNA 测序的结果,为研究者提供了进一步深度挖掘生物信息的资源,研究者可以在以下几方面展开研究:
(1)判断 DNA 链的某一特定位置会出现何种组蛋白修饰;
(2)检测 RNA polymerase II 及其它反式因子在基因组上结合位点的精确定位;
(3)研究组蛋白共价修饰与基因表达的关系;
(4) CTCF 转录因子研究。
- 本文由 ybzhao
- 转载请务必保留本文链接:https://www.plob.org/article/1538.html

