一、染色质的拓扑结构
染色质是DNA和蛋白质的复合物,遗传物质被包装在生物细胞内核。染色质结构是动态的并且精确调控基因表达及基本的细胞进程。其结构的变化可以被下一代遗传。
1.染色质的折叠
染色质的基本组成单位是核小体,核小体由165bp的DNA螺旋缠绕核心组蛋白(核心组蛋白由两个H2A、H2B、H3、H4组成的组蛋白八聚体构成)两圈构成。这个结构使DNA浓缩了5-10倍。DNA缠绕在组蛋白八聚体的表面,可能被一些调节蛋白调控。组蛋白的尾端也可以被一些蛋白修饰(如甲基化,乙酰化,磷酸化,泛素化等),促使核小体运动或者解开,进而对染色体复合物产生影响。
核小体之间是通过一段10-80bp的DNA连接起来。这些核小体进一步螺旋折叠形成30nm的核纤维,这个过程约将染色质压缩了50倍,这个30nm的核纤维是非常稳定的,这是因为H1组蛋白结合核小体之间,起到固定作用。关于这一结构仍有多的争论,染色质纤维内核小体包装的细微点,和关于这些纤维进一步发展的方式知之甚少。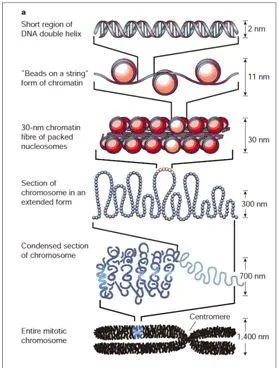
Felsenfeld G and Groudine M. Nature (2003)
核纤维经过进一步的折叠形成约300nm的染色质结构,这就是细胞正常状态下,染色质的结构。我们可以看到染色质是由许多环形的核纤维组成。下图蝾螈卵母细胞的灯刷染色体可以清楚地看到染色质的环形结构。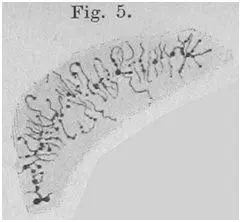
W. Flemming 1882; Paulson & Laemmli1978
当细胞进行有丝分裂时,染色质进一步折叠压缩形成染色体。
二、3D基因组的结构与组成
生命体的遗传功能元件,包括编码基因、非编码基因、顺式调控元件等,在空间结构上,并不是在染色体上呈线性地一字依次排开,而是随着DNA形成复杂高级结构的同时,具备了三维组织形式。
如下图所示,DNA双链像纠缠在一起的电话线一样,一圈圈地螺旋缠绕,压缩,最终形成染色质。我们可以看到30nm的核纤维经过环形折叠形成染色质。这种结构使得一维层面上相隔比较远的DNA区域反而靠的更近。比如下图中的ABCD四个点,若以A为参照物,C比B远,但由于基因组形成了高级结构,反而把A和C拉得更近。这个示意图还提示了另外一个问题,即同一条染色体上的某些区域,可能很难互相接触,比如B和D之间就,被环状结构给隔开了。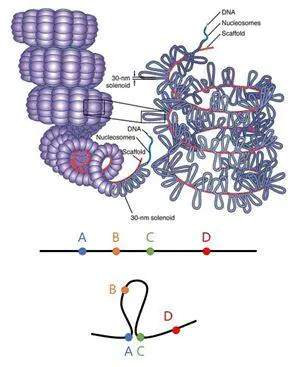
这种相对稳定的高级结构,是由蛋白质来维持的。这些蛋白大多是一些DNA转录因子、辅助因子等调控元件,其中CTCF是一个重要的成环蛋白。除了这些调控元件外,还有需要一类重要的蛋白去稳固这种高级结构,这类重要的蛋白就是黏着蛋白,它由SMC1、SMC3、RAD21和STAG1/2组成,其中SMC1和SMC3头尾相连组成一个环形,两蛋白的尾部是由相互作用的,紧密靠在一起,其头部游离,RAD21与两个蛋白的头部相结合,STAG1/2蛋白进一步加固结合。黏着蛋白是一个蛋白复合物,它像戒指一样固定在环的开口处,维持环的稳定性,标志性的黏着蛋白为RAD21蛋白。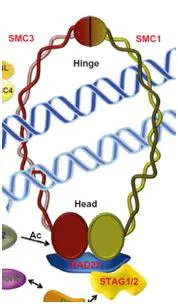
这种环形结构在体内并不是松散排布的。这些环形的染色质折叠让一些DNA片段彼此靠近并发生互作,这样的区域称为拓扑相关结构域TAD,TAD是非常长的DNA片段,在其中DNA形成了包含组蛋白、调节蛋白和转录因子的大型三维结构。每个TAD包含一个或多个基因连同其所有的调控元件。它们的结构在进化上是保守的,可能存在于不同的细胞类型以及各种物种中。一段TAD内的调控元件只能在“它们”的TAD中起作用;相反,在邻近TAD中的基因不受它们的影响。TADs通过许多边界元件隔开的,其中CTCF就是一个重要的TAD边界元件。
三、3D基因组的调控模型
我们已经知道,染色质是由独立功能单元TADs(拓扑相关结构域)组成的,TAD是由多个相互靠近的功能环组成。因此。我们研究3D基因组,实际上是研究DNA的功能环机构,前面我们已经了解到染色质的环形结构包括DNA,调节蛋白及黏着蛋白。下面我们来了解一下染色质功能环的几种基本模型。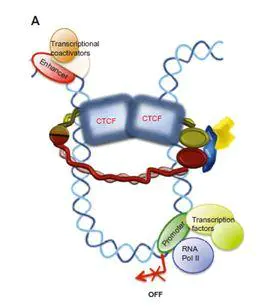
A图为典型的绝缘子调控模型,其中CTCF是一种重要的绝缘子蛋白。当CTCF蛋白成环时,CTCF在空间上阻隔了增强子与基因启动子之间的联系,RNA聚合酶II无法招募足够的转录因子,以致于基因无法正常转录。当CTCF环打开后,增强子与基因启动子之间的阻隔消失,增强子招募激活型转录因子,与RNA聚合酶II互作,开启基因转录。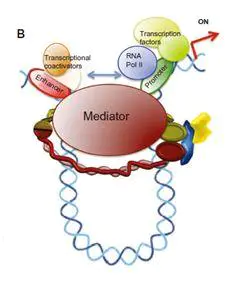
B图为经典的远程增强子调控模型。这类模型的调控模式与A图相对,它是成环后增强基因的表达,而不是抑制。其中的调节蛋白不再是CTCF蛋白,而是一些转录因子等其他调控元件。在DNA的一维结构上,增强子与基因启动子相隔较远,在没有成环的条件,增强子无法启动基因表达。但当环形成后,远程增强子与基因启动子的距离被拉进,增强子与RNA聚合酶II互作,启动基因转录。
四、3D基因组模型构建实例
基因组的三维构型是复杂的,动态的,且对基因调控至关重要。在过去的几年中,染色体构象捕获方法和显微技术的技术进步揭示了基因组如何与核结构相互联系,也部分解释了细胞类型之间以及细胞分化和发育过程中基因组发生的变化。
1.癌症模型中CTCF环模型
①CTCF位点超甲基化模型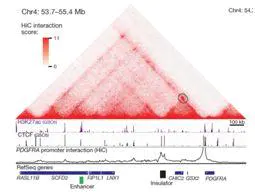
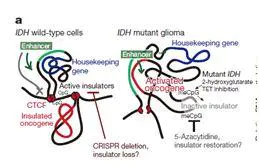
这是一个远程增强子互作模型,我们可以看到左侧是Hi-C与ChIP-seq联合分析的结果,我们从中明确了增强子,绝缘子与基因的位置线性关系和3维结构关系。从而分析出右图的模型。从模型中我们得知,在正常细胞中,CTCF稳定成环,使得原癌基因无法表达。IDH突变的神经胶质瘤中,CTCF位点也就是绝缘子被甲基化,CTCF成环消失,染色质变松散,原癌基因受到远程增强子的调控,表达增强。
②CTCF位点SNP突变模型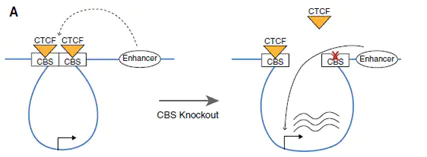
这是一个CTCF绝缘子上的SNP位点突变,导致原癌基因表达的模型,在正常条件下,CTCF结合在绝缘子处,阻碍了增强子与原癌基因的互作,使原癌基因沉默。当绝缘子上的SNP位点突变后,CTCF无法结合,增强子促进原癌基因表达。
2.CTCF环内部调控模型
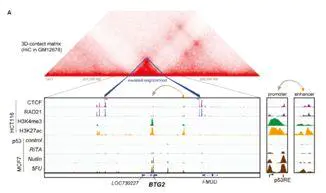
这是一个CTCF环内部的调控模型,从左图的Hi-C与ChIP-seq联合分析结果得P53成环在CTCF成环的内部,且成环对P53有依赖性。当P53低表达时,环会消失,当P53高表达时环形成,当环形成时,增强子与基因距离拉进,基因转录。
作者:Ray钱
链接:https://www.jianshu.com/p/e1ed65a65dab
来源:简书
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。

