写在前面
以下内容均来自我在菲沙基因(Frasergen)暑期生信培训班上记录的课堂笔记
1.Hi-C原理简介
1.1 Hi-C技术
高通量染色体构象捕获技术(High-throughput chromosome conformation capture)研究全基因组三维构象及分析染色质片段相互作用的实验技术- 1.2 Hi-C目的
了解核内染色质的三维构象、获得细胞核内空间位置非常接近或存在相互作用的染色质测序片段更好地研究染色质内或染色质间的互作、基因调控元件在全基因组范围内调控的情况 - 1.3 Hi-C应用方向
辅助基因组组装、揭示空间调控、揭示物种进化、疾病研究、三维结构差异分析、还原染色体三维结构、构建染色体跨度单体型 - 1.4 互作本质
- 1.5 Hi-C实验原理
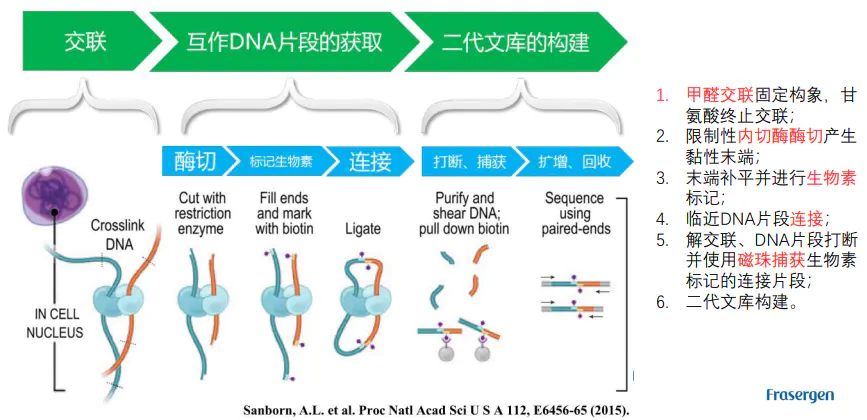
- 1.6 二代文库构建及测序
二代文库进行片段筛选400-600bp的片段,实际插入片段长度为300-500bp
一般测序读长:PE150
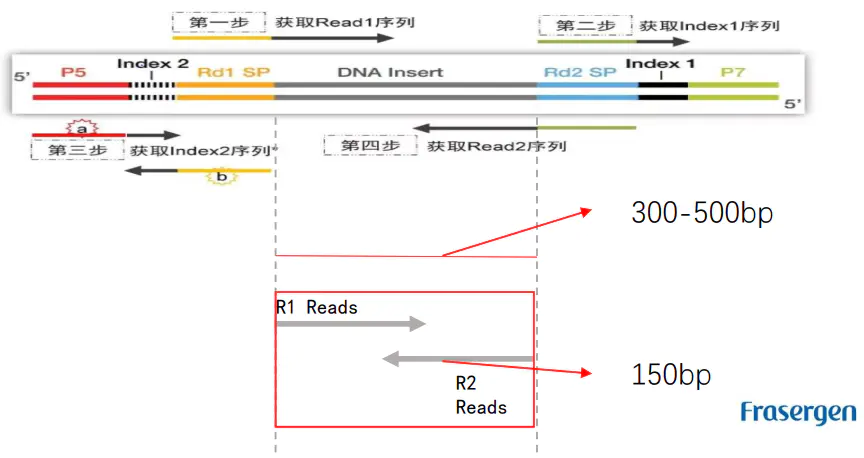
- 1.7 Hi-C实际文库类型
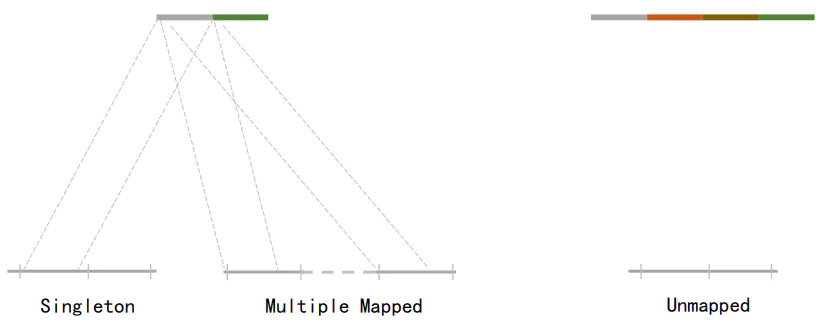
将HIC数据进行比对是会出现不同的比对情况,我们需要的是。对单端匹配、多处比对、未比对的reads进行过滤。
对Hi-C文库构建中产生的多种分子类型,包括 re-ligation、Dangling ends、self circle 、dump reads 及valid pairs reads等类型。 在 Hi-C 分析中,仅valid pair可以反映基因组上位点与位点间的互作信息。因此,非重复的valid pair所占的比例是评估Hi-C文库质量的 重要指标。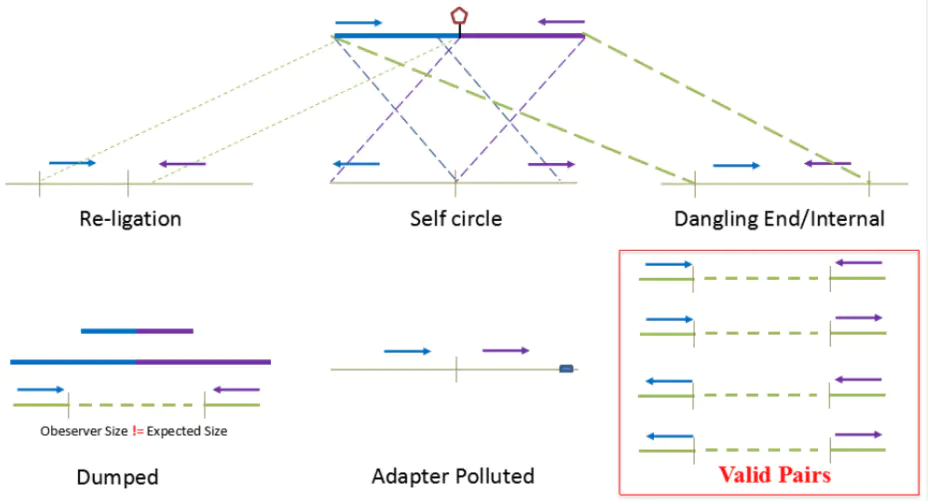
- 互作矩阵的生成
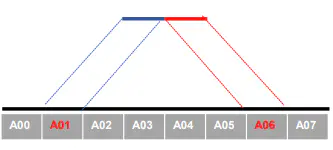
由于计算资源,数据量等因素,我们往往认为确定一个互作单位:bin。将基因组按照一定大小分成bin。将过滤后的有效序列分配到这些bin中。
互作矩阵的矫正
Hi-C数据中由于内切酶的偏好性、基因组本身质量、基因组序列特异性会导致其在基因组不同位置间存在偏差。因此,我们会对互作矩阵进行校正,使其数据在基因组上每个位点的覆盖度一致。
常用的矫正方式有等

2.比对软件介绍
常用短序列比对软件 | | Bowtie2 | BWA | | —- | —- | —- | | 算法原理 | FM-Index(基于BWT) | BWT construction algorithm | | 常用比对模式 | End-to-End | Mem(pair-end) | | 输出 | SAM、TSV | SAM | | 特点 | 支持单端、双端reads比对;支持插入、缺失错误比对 | 支持单端、双端reads比对;支持插入、缺失、嵌合reads比对 | | 区别 | MAPQ值打分算法不同于BWA | 处理嵌合reads时会分段输出比对结果;基因组mapping率略高于Bowtie2 |
SAM格式详解
SAM分为两部分,注释信息(header section)和比对结果部分(alignment section)
注释信息:可有可无,以@开头,用不同的tag代表不同的信息
比对结果: | 列 | 字段名 | 中文解释 | 举例 | | —- | —- | —- | —- | | 1 | QNAME | 比对片段的编号,read name | V300059328L4C001R0010000044 | | 2 | FLAG | 位标符,reads mapping情况的数字表示 | 16 | | 3 | RNAME | 比对上参考序列的编号 | chr10 | | 4 | POS | 比对上参考序列的位置,1-based | 321541 | | 5 | MAPQ | 比对的质量分数MAPQ=-10 * log10(mapping出错的概率) | 60 | | 6 | CIGAR | 简要比对表达式 | 150M | | 7 | MRNM | mate比对上的参考序列 | chr10 | | 8 | MPOS | mate比对参考序列的位置 | 322000 | | 9 | ISIZE | reads比对长度 | 470 | | 10 | SEQ | reads的序列 | | | 11 | QUAL | ASCII 码格式的序列质量 | | | 12 | 可选区域 | AS:i 匹配的得分;XS:i 第二好的匹配的得分;YS:i mate 序列匹配的得分 | |
3.HiC常规软件比较
| 软件名 | hiclib | HiC-Pro | HICUP | Juice |
|---|---|---|---|---|
| 比对软件 | Bowtie2 | Bowtie2 | Bowtie2 | BWA-mem |
| 比对策略 | 迭代比对 | 全局、局部比对 | 先截短后比对 | Pair-end,嵌合reads过滤 |
| 嵌合reads处理 | √ | √ | √ | √ |
| 构建矩阵 | √ | √ | × | √ |
| 标准化 | ICE | ICE | × | KR |
| 结果文件 | hdf5、hm、bychr(HDF5) | SAM、validpair | SAM | SAM、MND、.hic |
| 特点 | 比对结果可靠,存储消耗小 | 简单易用,输出结果可读 | 过滤非常严格 | 后续分析接口多,juicebox可视化 |
4.HiC-Pro代码实操
4.1 软件安装
-
git clone https://github.com/nservant/HiC-Pro.gitcd ./HiC-Provi config-install.txt
修改HiC-Pro目录下的config-install.txt
########################################################################### Paths and Settings - Start editing here !#########################################################################PREFIX = 文件安装位置BOWTIE2_PATH = bowtie2安装目录SAMTOOLS_PATH = samtools安装目录R_PATH = R的安装目录PYTHON_PATH = python安装目录CLUSTER_SYS = 用于集群提交的调度器,必须为TORQUE,SGE,SLURM,LSF四个中的一种
修改保存后
make CONFIG_SYS=config-install.txt install
4.2 bowtie2索引构建
bowtie2-build [options] <reference> <bt2_index_base>
reference : 下载的参考基因组,genome.fa
- bt2_index_base: 构建索引前缀
4.3 使用digest_genome.py生成酶切片段文件
-r:常用限制性内切酶:python HiC-Pro/bin/utils/digest_genome.py -r [常用限制性内切酶序列] [-o OUT] fastafile
| 限制性内切酶 | 酶切位点,^为切割位点 |
|---|---|
| MboI | ^GATC |
| DpnII | ^GATC |
| BglII | A^GATCT |
| HindIII | A^AGCTT |
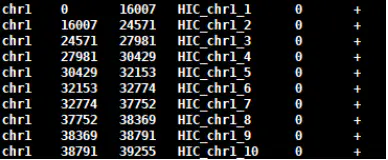
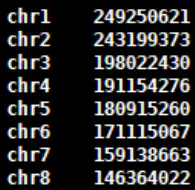
4.3 生成基因组sizes文件,获得基因组每条染色体bases数bed文件
samtools faidx genome.faawk ‘{print $1 "\t" $2}‘ genome.fa.fai > genome_sizes.bed
4.4 Hi-C数据准备
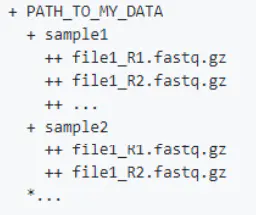
- 创建sample文件夹,一个文件夹放入一个样品的fastq文件(生物学重复可以放入)
4.5 配置Config文件
vi ./config-install.txt
需要修改的参数有:
N_CPU:给定的CPU内存数,给的越多,运行的越快(根据服务器配置);
LOGFILE:日志文件的名称;
JOB_MEM:内存的大小
PAIR1_EXT= _R1 :R1测序数据名称中有_R1
PAIR2_EXT = _R2:R2测序数据名称中有_R2
MIN_MAPQ: 最低的质量分数,用于筛选,表示低于该MAPQ值会被过滤
BOWTIE2_IDX_PATH: 基因组bowtie2索引路径,eg:/path/hg19
BOWTIE2_GLOBAL_OPTIONS: 默认GLOBAL比对设置
BOWTIE2_LOCAL_OPTIONS: 默认LOCAL比对设置
REFERENCE_GENOME: Bowtie2索引前缀
GENOME_SIZE: 基因组sizes bed文件
GENOME_FRAGMENT: 基因组酶切文件,eg. /path/hg19_HindIII.bed
LIGATION_SITE: 酶切位点末端补平再次连接后形成的嵌合序列,eg. AAGCTAGCTT
MIN_FRAG_SIZE: 最小的理论酶切片段大小,eg. 100
MAX_FRAG_SIZE: 最大的理论酶切片段大小,eg. 100000
MIN_INSERT_SIZE: 最小的文库片段大小,eg.100
MAX_INSERT_SIZE: 最大的文库片段大小,eg.1000
BIN_SIZE:需要生成的矩阵分辨率(bp)
MATRIX_FORMAT:矩阵的形式,upper表示保留上半部分4.6 HiC-Pro运行
HiC-Pro -i INPUT -o OUTPUT -c CONFIG [-s ANALYSIS_STEP] [options]
-c: config文件路径
-o: 结果生成路径
-i: 原始数据路径
-p: 集群运行5.结果解读
总目录
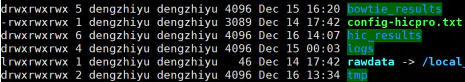
bowtie_results:比对结果目录
hic_results:hic矩阵及分析结果目录
logs:存放分析日志
rawdata:链接了原始数据
tmp:存放中间文件Bowtie_result目录
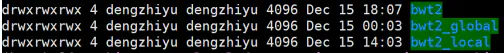
bwt2:存放合并后的bam文件和统计结果
bwt2_global:存放全局比对结果
bwt2_local:存放局部比对结果hic_result目录
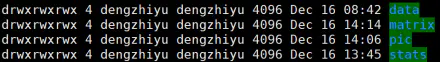
data:存放validpair及其他无效数据文件
matrix:存放不同分辨率矩阵文件
pic:存放统计分析图片
stats:存放统计表Data文件
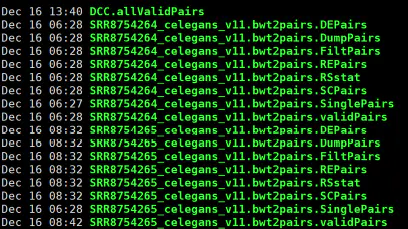
- Matrix文件
raw:原始矩阵
iced:ice标准化后的矩阵
- Pic文件,出图
HiC文库片段分布文件 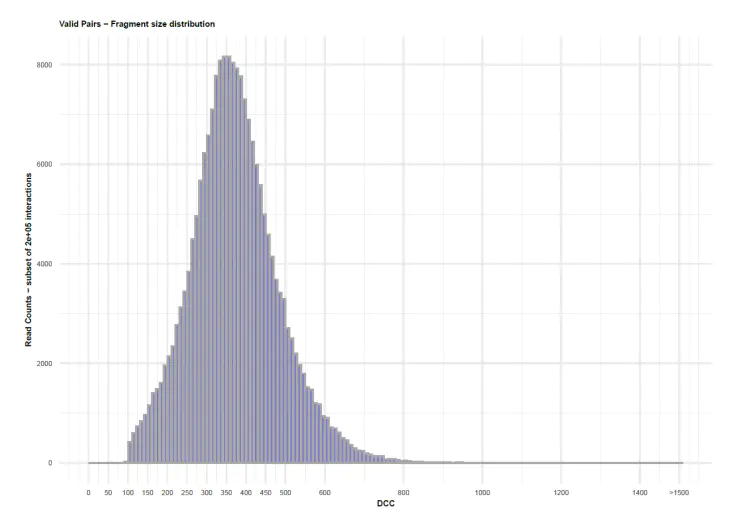
双端比对过滤质控图 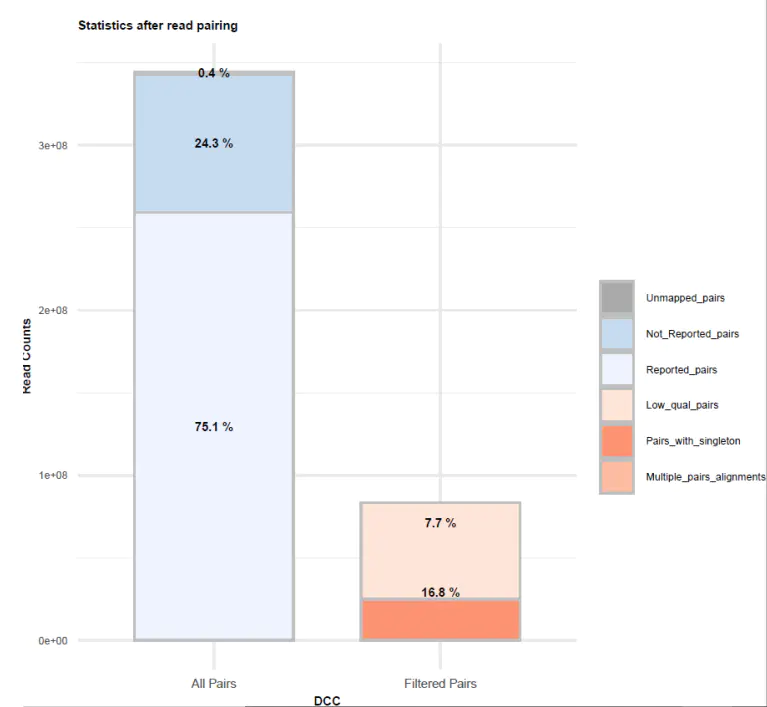
有效数据过滤质控图 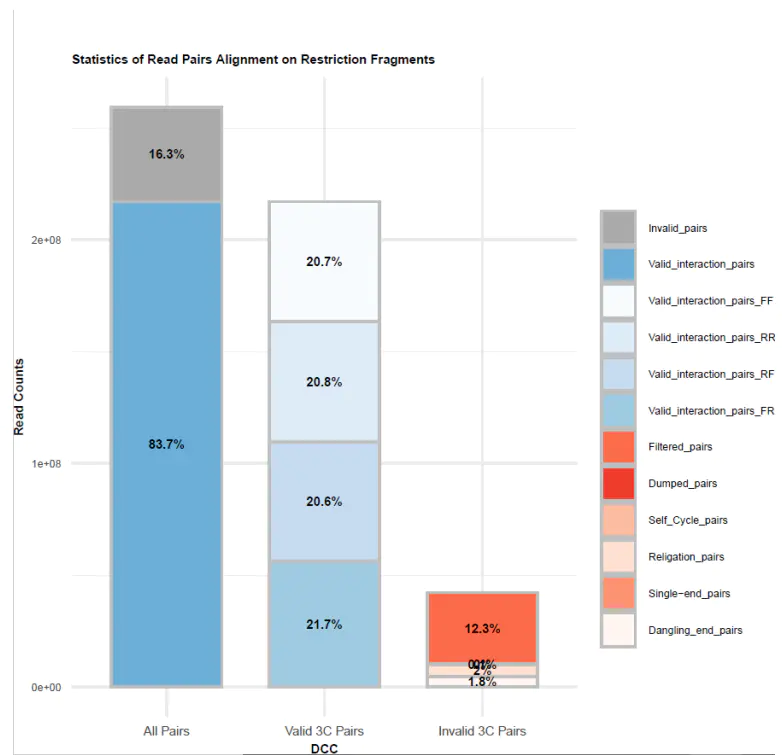
配对数据不同类型数据比例展示图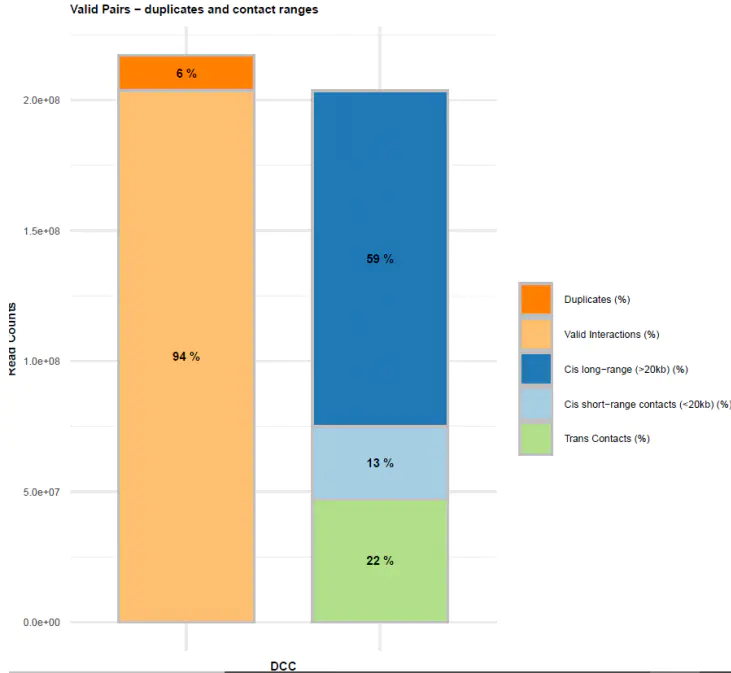
作者:Bioinfo鱼
链接:https://www.jianshu.com/p/facec96ee6ac
来源:简书
著作权归作者所有。商业转载请联系作者获得授权,非商业转载请注明出处。