ATAC-Seq分析教程系列
ATAC-Seq分析教程:ATAC-seq的背景介绍以及与ChIP-Seq的异同
ATAC-Seq分析教程:原始数据的质控、比对和过滤
ATAC-Seq分析教程:用MACS2软件call peaks
ATAC-Seq分析教程:对ATAC-Seq/ChIP-seq的质量评估(一)phantompeakqualtools
ATAC-Seq分析教程:对ATAC-Seq/ChIP-seq的质量评估(二)ChIPQC
ATAC-Seq分析教程:重复样本的处理-IDR
ATAC-Seq分析教程:用ChIPseeker对peaks进行注释和可视化
ATAC-Seq分析教程:用网页版工具做功能分析和motif分析
ATAC-Seq分析教程:差异peaks分析——DiffBind
ATAC-Seq分析教程:ATAC-Seq、ChIP-Seq、RNA-Seq整合分析
由于基因表达调控机制的复杂性,多种组学数据的整合分析,从不同的层面探究生物问题越来越重要。从RNA-Seq层面,我们可以探究哪些基因具有显著差异,上调或下调;从ChIP-Seq层面,我们可以研究某个特定转录因子的调控作用;从ATAC-Seq我们可以了解到染色质可及性的动态变化,由于染色质的可及性与调控元件或转录因子的结合密切相关,在转录调控中发挥着重要的作用。因此,整合分析可以进一步探究调控某一生物学过程的关键因子(包括顺式调控元件和转录因子),以及哪个转录因子调控了感兴趣的基因,以及感兴趣的转录因子的靶基因等。
目前,好像并没有标准化的方法用来整合比较这三种数据。不过在文献中可以看到有很多相同的或不同的思路和方法做整合分析,大家可以在学习交流群中推荐文献,一起解读学习。
下面的内容主要介绍这一节课程中RNA-Seq和ChIP-Seq的整合分析中提到的两种方法:
一是直接比较,即首先得到差异基因与ChIP-Seq靶基因的overlap,然后选择一些关键基因比较一下谱图。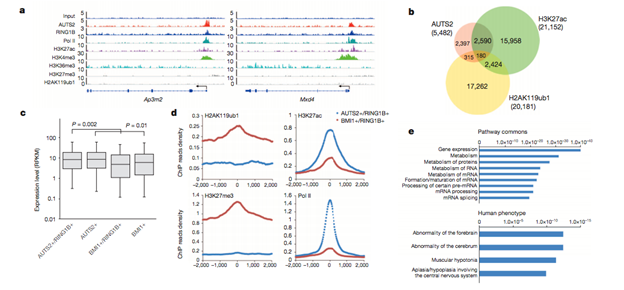
课程中提到的另一种方法是使用BETA工具:
BETA (Binding and Expression Target Analysis)是 Shirley Liu实验室开发的工具,通过整合转录因子或染色质调控因子的ChIP-Seq与差异基因的表达直接预测靶基因,而且有可能发现增强子区的蛋白质的靶基因。
BETA有三个功能:
- 预测转录因子的功能是激活还是抑制
- 预测转录因子的靶基因
- 鉴定转录因子的motif以及调控转录因子激活或抑制的其他因子
BETA包括三个命令取决于输入数据格式和想要的输出数据
- BETA basic: 预测转录因子的功能是激活还是抑制,直接检测靶标
- BETA plus:BETA basic 靶标区域的motif分析
- BETA minus:只基于结合数据的调控潜能的值预测TF靶基因
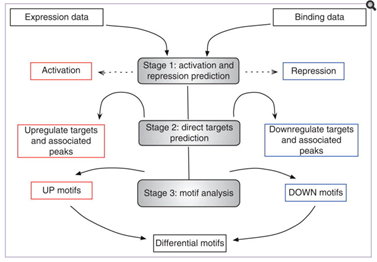
工作原理
- 每个基因的调控潜能值是通过计算基因TSS的指定范围内的所有结合位点来计算的
- 调控潜能是基因受因子调节的可能性,取决于TSS范围的结合位点数以及与结合位点与TSS之间的距离
- BETA minus按照调控潜能值对基因排序来鉴定靶标
BETA basic需要特定格式的差异分析结果和显著性的结果。它使用CDF来判断上调基因和下调基因是否与NON-DE不同,这是用来识别激活和抑制功能。使用调控潜能和差异统计量计算每一个基因的得分排序,该得分排序可以用于识别靶标。
- 转载请务必保留本文链接:https://www.plob.org/article/24710.html

