文章来源:BMC中国
_北京大学生命科学学院贾璐萌,李亭亭(李程研究组)
_
上回说到Hi-C更适用于研究大尺度上的染色质结构,而不适用于研究转录调控元件之间的相互作用。
转录调控元件主要包括启动子、增强子、绝缘子及抑制子。在转录的起始阶段,基因的启动子与其他远距离调控元件(增强子、绝缘子及抑制子)空间靠近(图 4)。远距离调控元件通过帮助启动子招募具有抑制或者激活功能的转录相关蛋白,调控基因的转录 (Zhang et al., 2013)。为了结合转录相关蛋白,这些转录调控元件通常缺少核小体的结合,这使得活跃调控区域的染色质相对开放(开放染色质)。针对转录调控元件的这些特点,可通过捕获转录相关蛋白结合区域 [ChIP-seq (Johnson et al., 2007)] 及捕获染色质开放区域 [DNase-seq (Boyle et al., 2008)、FAIRE-seq (Giresi et al., 2007)、MNase-seq (Schlesinger et al., 2013)、ATAC-seq (Buenrostro et al., 2013)、NicE-seq(Ponnaluri et al., 2017)] 的技术来检测转录调控元件。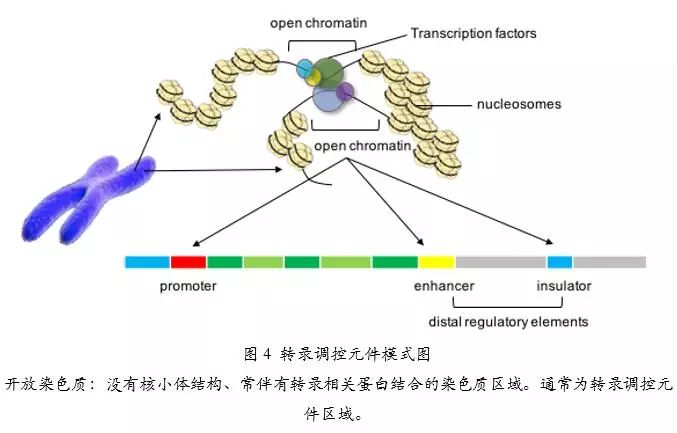
为了更好的捕获基因组转录调控元件之间的远距离相互作用,结合转录调控元件检测技术的其他染色质构象捕获方法被提出。这些方法主要包括三类。
第一类,是转录相关蛋白介导的染色质相互作用的捕获技术(ChIA-PET (Fullwood et al., 2009)、HiChIP (Mumbach et al., 2016)、PLAC-seq (Fang et al., 2016))。在这类技术中,最早被提出的是阮一骏等开发的ChIA-PET技术(图5)。在ChIA-PET技术中,交联好的染色质首先被超声打碎成独立的相互作用复合体;结合有目的蛋白的复合体通过免疫沉淀技术被富集;目的蛋白结合的复合体DNA末端被连接包含MmeI位点的生物素化寡核苷酸linker;通过linker间的连接,目的蛋白介导的存在相互作用的DNA被连接至同一片段; MmeI片段化DNA后,DNA被纯化;随后PET片段被建库测序。ChIA-PET作为一种可以有效富集蛋白介导的染色质间相互作用的技术,自开发以来,得到了广泛的应用。RNAPII ChIA-PET及CTCF ChIA-PET技术不仅帮助解析了转录相关的染色质拓扑结构 (Li et al., 2012; Tang et al., 2015),还成功帮助解释了非编码区SNP位点对远距离调控基因表达的影响(Tang et al., 2015)。2017年,同样是用于捕获基因组转录调控元件之间的远距离相互作用的技术Hi-ChIP (Mumbach et al., 2016)及PLAC-seq(Fang et al., 2016)被开发。这两种技术都在Hi-C技术的基础上结合了免疫沉淀技术,从实验目的上与ChIA-PET一致,目前来看,与ChIA-PET技术相比,Hi-ChIP与PLAC-seq能得到更高比例的有效reads。相信Hi-ChiP与PLAC-seq也将被广泛应用于蛋白介导的染色质相互作用的研究。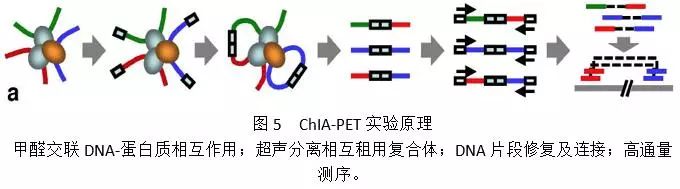
第二类,是目的探针所在区域的染色质相互作用的捕获(Capture Hi-C)。这类技术以promoter capture Hi-C(Mifsud et al., 2015)技术为代表,根据promoter区域设计RNA探针,在Hi-C实验的基础上加入了一步探针的富集,检测promoter探针所在区域的染色质相互作用。
第三类,是基于开放染色质间相互作用的捕获[DNase Hi-C (Ma et al., 2015)、Micro-C (Hsieh et al., 2015)、BL-Hi-C (Liang et al., 2017)]。DNase Hi-C技术使用DNase酶取代Hi-C实验中的限制性内切酶,消化染色质,可特异性地捕获DNase I敏感的开放染色质间相互作用 (Ma et al., 2015)。Micro-C技术使用MNase取代Hi-C实验中的限制性内切酶消化染色质,可以得到核小体尺度分辨率的DNA相互作用图谱 (Hsieh et al., 2015)。BL-Hi-C 技术使用识别序列为“GGCC”的限制性内切酶HaeIII进行染色质消化。由于结合有转录相关蛋白及结构蛋白的活跃染色质区域DNA的GC碱基组成比例更高,故BL-Hi-C可富集高GC区域的活跃染色质间的相互作用。
这些方法各有优劣及侧重,从不同层面上解析染色质空间结构、回答重要生物学问题。ChIA-PET (Fullwood et al., 2009)、HiChIP (Mumbach et al., 2016)、PLAC-seq (Fang et al., 2016)仅能捕获某一种蛋白介导的染色质间的相互作用;Capture Hi-C技术通过特异性探针,捕获探针所在区域(如启动子区)染色质间相互作用 (Mifsud et al., 2015);DNase Hi-C虽然可以富集开放染色质之间的相互作用,但是由于DNase I 整合效率非常高,高度片段化的染色质之间自连或随机连接,减少了真实相互作用的捕获比例 (Li et al., 2018)。BL-Hi-C捕获了10000多个高GC区域的染色质相互作用,这些相互作用大多是CTCF HiChIP及RNAPII HiChIP的子集。这些技术虽然各有优势,然而大都依赖目的蛋白、探针序列、酶切偏好。综上所述,一种不依赖于探针序列及蛋白抗体、用染色质开放程度为富集条件、直接高效富集全基因组活跃转录调控元件间相互作用的技术需要被开发。
参考文献:
- Boyle, A.P., Davis, S., Shulha, H.P., Meltzer, P., Margulies, E.H., Weng, Z., Furey, T.S., and Crawford, G.E. (2008). High-resolution mapping and characterization of open chromatin across the genome. Cell 132, 311-322.
- Buenrostro, J.D., Giresi, P.G., Zaba, L.C., Chang, H.Y., and Greenleaf, W.J. (2013). Transposition of native chromatin for fast and sensitive epigenomic profiling of open chromatin, DNA-binding proteins and nucleosome position. Nature methods 10, 1213-1218.
- Fang, R., Yu, M., Li, G., Chee, S., Liu, T., Schmitt, A.D., and Ren, B. (2016). Mapping of long-range chromatin interactions by proximity ligation-assisted ChIP-seq. Cell research 26, 1345-1348.
- Fullwood, M.J., Liu, M.H., Pan, Y.F., Liu, J., Xu, H., Mohamed, Y.B., Orlov, Y.L., Velkov, S., Ho, A., Mei, P.H., et al. (2009). An oestrogen-receptor-alpha-bound human chromatin interactome. Nature 462, 58-64.
- Giresi, P.G., Kim, J., McDaniell, R.M., Iyer, V.R., and Lieb, J.D. (2007). FAIRE (Formaldehyde-Assisted Isolation of Regulatory Elements) isolates active regulatory elements from human chromatin. Genome research 17, 877-885.
- Hsieh, T.H., Weiner, A., Lajoie, B., Dekker, J., Friedman, N., and Rando, O.J. (2015). Mapping Nucleosome Resolution Chromosome Folding in Yeast by Micro-C. Cell 162, 108-119.
- Johnson, D.S., Mortazavi, A., Myers, R.M., and Wold, B. (2007). Genome-wide mapping of in vivo protein-DNA interactions. Science 316, 1497-1502.
- Li, G., Ruan, X., Auerbach, R.K., Sandhu, K.S., Zheng, M., Wang, P., Poh, H.M., Goh, Y., Lim, J., Zhang, J., et al. (2012). Extensive promoter-centered chromatin interactions provide a topological basis for transcription regulation. Cell 148, 84-98.
- Li, T., Jia, L., Cao, Y., Chen, Q., and Li, C. (2018). OCEAN-C: mapping hubs of open chromatin interactions across the genome reveals gene regulatory networks. Genome biology 19, 54.
- Liang, Z., Li, G., Wang, Z., Djekidel, M.N., Li, Y., Qian, M.P., Zhang, M.Q., and Chen, Y. (2017). BL-Hi-C is an efficient and sensitive approach for capturing structural and regulatory chromatin interactions. Nat Commun 8, 1622.
- Ma, W., Ay, F., Lee, C., Gulsoy, G., Deng, X., Cook, S., Hesson, J., Cavanaugh, C., Ware, C.B., Krumm, A., et al. (2015). Fine-scale chromatin interaction maps reveal the cis-regulatory landscape of human lincRNA genes. Nature methods 12, 71-78.
- Mifsud, B., Tavares-Cadete, F., Young, A.N., Sugar, R., Schoenfelder, S., Ferreira, L., Wingett, S.W., Andrews, S., Grey, W., Ewels, P.A., et al. (2015). Mapping long-range promoter contacts in human cells with high-resolution capture Hi-C. Nature genetics 47, 598-606.
- Mumbach, M.R., Rubin, A.J., Flynn, R.A., Dai, C., Khavari, P.A., Greenleaf, W.J., and Chang, H.Y. (2016). HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature methods 13, 919-922.
- Ponnaluri, V.K.C., Zhang, G., Esteve, P.O., Spracklin, G., Sian, S., Xu, S.Y., Benoukraf, T., and Pradhan, S. (2017). NicE-seq: high resolution open chromatin profiling. Genome Biol 18, 122.
- Schlesinger, F., Smith, A.D., Gingeras, T.R., Hannon, G.J., and Hodges, E. (2013). De novo DNA demethylation and noncoding transcription define active intergenic regulatory elements. Genome research 23, 1601-1614.
- Tang, Z., Luo, O.J., Li, X., Zheng, M., Zhu, J.J., Szalaj, P., Trzaskoma, P., Magalska, A., Wlodarczyk, J., Ruszczycki, B., et al. (2015). CTCF-Mediated Human 3D Genome Architecture Reveals Chromatin Topology for Transcription. Cell 163, 1611-1627.
- Zhang, Y., Wong, C.H., Birnbaum, R.Y., Li, G., Favaro, R., Ngan, C.Y., Lim, J., Tai, E., Poh, H.M., Wong, E., et al. (2013). Chromatin connectivity maps reveal dynamic promoter-enhancer long-range associations. Nature 504, 306-310.

