文章来源:BMC中国
北京大学生命科学学院 贾璐萌、李亭亭(李程研究组)
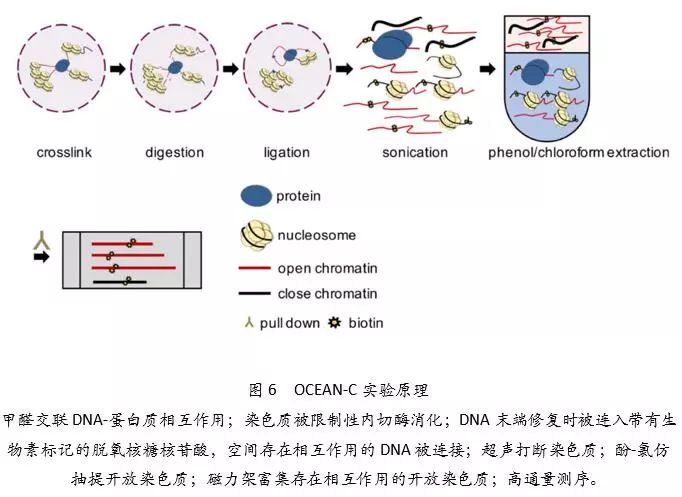
OCEAN-C(Open Chromatin Enrichment And Network Hi-C)技术(图 6)(Li et al., 2018)是结合了 FAIRE-seq 技术及 Hi-C 技术的关键步骤而开发的一种可以不依赖蛋白抗体及靶序列的新技术。在OCEAN-C技术中,染色质首先被甲醛交联;随后,染色质被限制性内切酶(MboI)消化;DNA末端修复时引入带有生物素标记的脱氧核糖核苷酸;随后空间存在相互作用的DNA被连接;超声打断染色质后,开放染色质被酚-氯仿抽提;最后,存在相互作用的开放染色质因为有生物素信号,可以被富集用于后续文库构建。 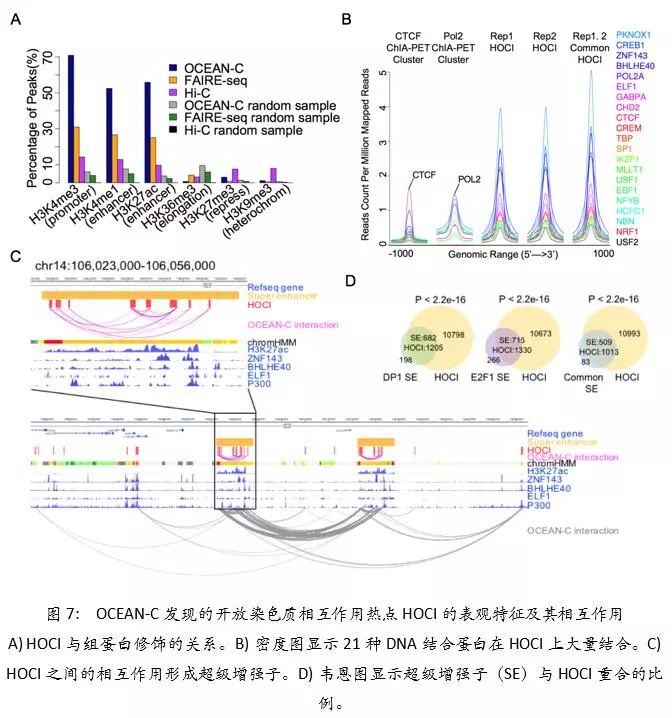
与其他技术相比,第一:OCEAN- C 技术不仅能检测到TAD、compartments等三维基因组结构,而且同时富集开放染色质区域及这些开放染色质之间的相互作用。第二:我们在三种 B 细胞相关细胞(U266,RPMI8226,GM12878)中应用了 OCEAN-C 技术,发现三种细胞中均有近 10000 个开放染色质相互作用中心(HOCI)。HOCI长度在 1 kb 左右、以活跃启动子及增强子为主、并结合有大量转录相关蛋白。许多HOCI也与Super-enhancer、broad H3K4me3区域有重合,帮助理解这些新发现的重要调控区域内部和之间的互作(图7)。第三:HOCI作为相互作用网络的重要节点,与其他开放染色质互作,形成全基因组开放染色质相互作用网络,调控基因表达(图8)。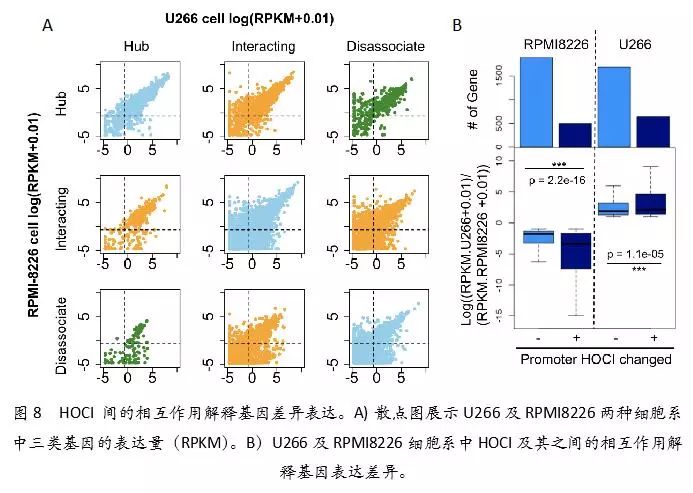
随着二代测序技术的发展,越来越多的染色质构象捕获技术被开发。三维基因组实验技术的应用越来越广泛,在细胞周期、细胞分化、疾病、指导基因组组装等各方面的研究中都有应用。例如,Hi-C技术用于研究细胞有丝分裂过程中的染色质结构变化:在有丝分裂中期,染色质会失去分区和 TAD结构,坍缩为一个密集的染色质状态 (Naumova et al., 2013);5C技术用于研究干细胞分化过程中核染色质的变化:在分化过程中,由于染色质三维结构的变化,OCT4的启动子和远程增强子的相互作用减弱,导致OCT4表达量下降,促进细胞分化 (Beagan et al., 2016)。Hi-C技术同样成功揭示了TAD边界的变化对疾病产生的影响:TAD边界遭到破坏造成原本被隔离的增强子与启动子形成异常的相互作用,导致基因时空表达的紊乱而产生疾病,如手指畸形疾病 (Lupianez et al., 2015)和大脑胶质瘤 (Flavahan et al., 2016);
如今,随着三维基因组研究方法的更新和研究范围的扩展,越来越多的新的染色质构象捕获技术被开发。最近,曹罡课题组和李国亮课题组又合作开发了更加高效经济的DLO Hi-C技术(Lin et al., 2018)。不仅如此,单细胞Hi-C技术也逐渐被开发 (Flyamer et al., 2017; Nagano et al., 2013; Ramani et al., 2017)。2014年美国国立卫生研究院(NIH)为此专门制定了4DN计划(4D Nucleome)(Dekker et al., 2017),斥资1.2亿美元资助了24个研究项目。4DN计划结合三维基因组学、单细胞测序、单细胞实时成像等前沿技术,开展标准化、高通量、单细胞层面、可动态观测的三维基因组学技术和应用研究。这些技术和项目融合了多学科的技术,将极大地促进后基因组学时代从基因组的一维列研究向四维的基因组空间结构和随时间变化的研究转变。我们将继续开发和应用OCEAN-C技术,研究以富集超级增强子、broad H3K4me3等结合有大量转录因子的基因调控元件间的相互作用,并与基因表达变化关联,帮助进一步阐明基因组结构、功能与疾病的关系。
参考文献:
- Beagan, J.A., Gilgenast, T.G., Kim, J., Plona, Z., Norton, H.K., Hu, G., Hsu, S.C., Shields, E.J., Lyu, X., Apostolou, E., et al. (2016). Local Genome Topology Can Exhibit an Incompletely Rewired 3D-Folding State during Somatic Cell Reprogramming. Cell Stem Cell 18, 611-624.
- Dekker, J., Belmont, A.S., Guttman, M., Leshyk, V.O., Lis, J.T., Lomvardas, S., Mirny, L.A., O’Shea, C.C., Park, P.J., Ren, B., et al. (2017). The 4D nucleome project. Nature 549, 219-226.
- Flavahan, W.A., Drier, Y., Liau, B.B., Gillespie, S.M., Venteicher, A.S., Stemmer-Rachamimov, A.O., Suva, M.L., and Bernstein, B.E. (2016). Insulator dysfunction and oncogene activation in IDH mutant gliomas. Nature 529, 110-114.
- Flyamer, I.M., Gassler, J., Imakaev, M., Brandao, H.B., Ulianov, S.V., Abdennur, N., Razin, S.V., Mirny, L.A., and Tachibana-Konwalski, K. (2017). Single-nucleus Hi-C reveals unique chromatin reorganization at oocyte-to-zygote transition. Nature 544, 110-114.
- Li, T., Jia, L., Cao, Y., Chen, Q., and Li, C. (2018). OCEAN-C: mapping hubs of open chromatin interactions across the genome reveals gene regulatory networks. Genome biology 19, 54.
- Lin, D., Hong, P., Zhang, S., Xu, W., Jamal, M., Yan, K., Lei, Y., Li, L., Ruan, Y., Fu, Z.F., et al. (2018). Digestion-ligation-only Hi-C is an efficient and cost-effective method for chromosome conformation capture. Nat Genet.
- Lupianez, D.G., Kraft, K., Heinrich, V., Krawitz, P., Brancati, F., Klopocki, E., Horn, D., Kayserili, H., Opitz, J.M., Laxova, R., et al. (2015). Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell 161, 1012-1025.
- Nagano, T., Lubling, Y., Stevens, T.J., Schoenfelder, S., Yaffe, E., Dean, W., Laue, E.D., Tanay, A., and Fraser, P. (2013). Single-cell Hi-C reveals cell-to-cell variability in chromosome structure. Nature 502, 59-64.
- Naumova, N., Imakaev, M., Fudenberg, G., Zhan, Y., Lajoie, B.R., Mirny, L.A., and Dekker, J. (2013). Organization of the mitotic chromosome. Science 342, 948-953.
- Ramani, V., Deng, X., Qiu, R., Gunderson, K.L., Steemers, F.J., Disteche, C.M., Noble, W.S., Duan, Z., and Shendure, J. (2017). Massively multiplex single-cell Hi-C. Nat Methods 14, 263-266.

