有时候,我们希望知道某些序列模式在给定的序列集合,如基因组中,出现的具体位置和频次。可以使用这一功能,快速定位,支持正则表达式等。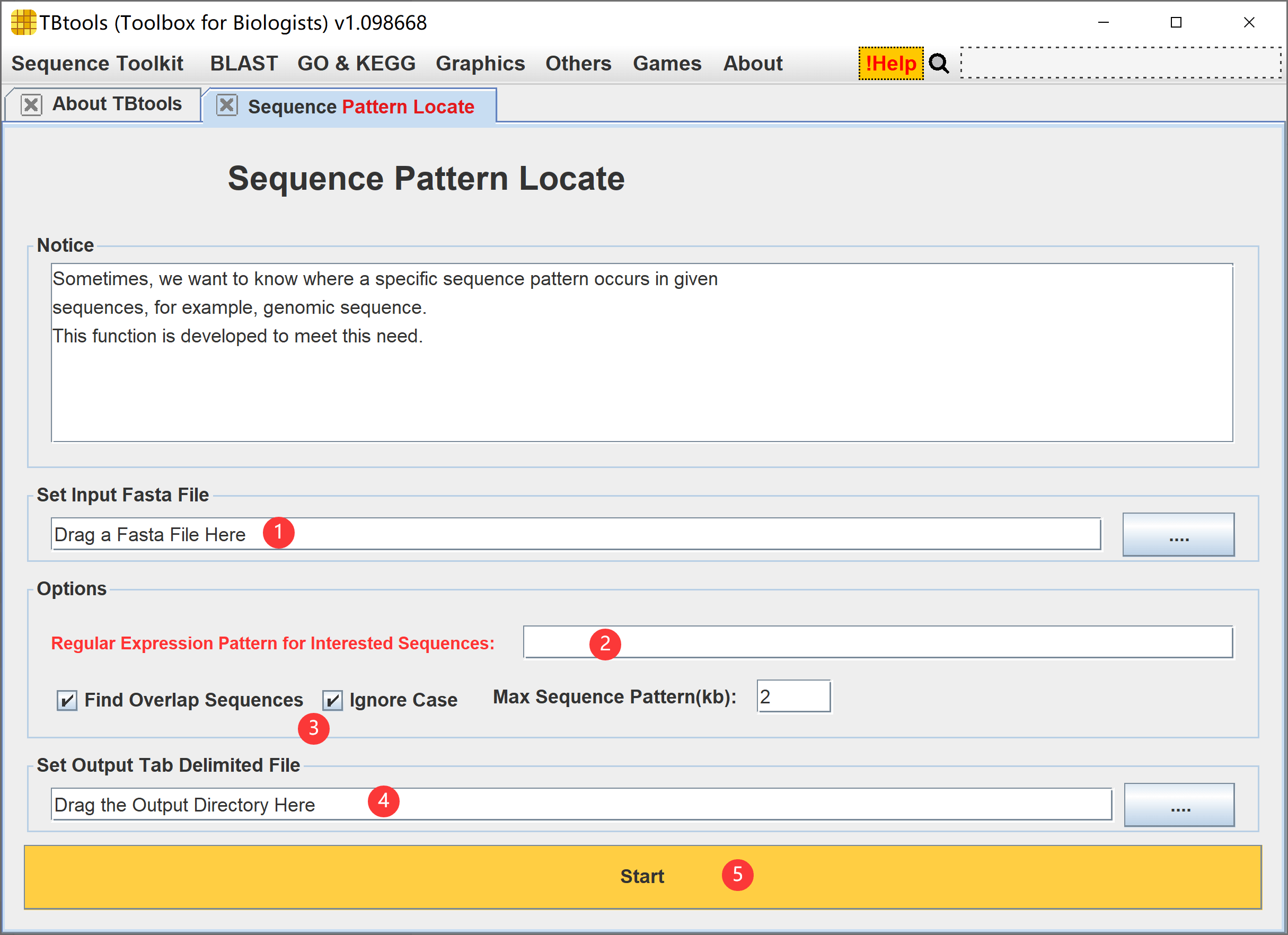
参数详解:
- 设置输入的 Fasta 序列文件
- 设置待搜索的序列模式,支持正则表达式,如使用“(AT){10,}”可检索出“AT”串联重复超过10次以上的位置
- 调整检索参数:
- Find Overlap Sequences,即是否允许搜索出来的模式存在重叠,如“ATG(\w{3})+TGA”,那么可能搜索出“ATGATGCATCGATCGACGTAGCTGA”,事实上,其中还覆盖另一个模式“ATGCATCGATCGACGTAGCTGA”
- Ignore Case,大小写不敏感
- Max Sequence Pattern(kb),注意到,一个序列模式匹配的字符串跨越长度受这个参数限制
- 设置输出的文件路径
具体使用示例,建议参考大范围查找序列模式所在位置,用TBtools啊~

