TBtools 的早期版本即开始 提供 GO 注释功能,主要原理,即 ID Mapping。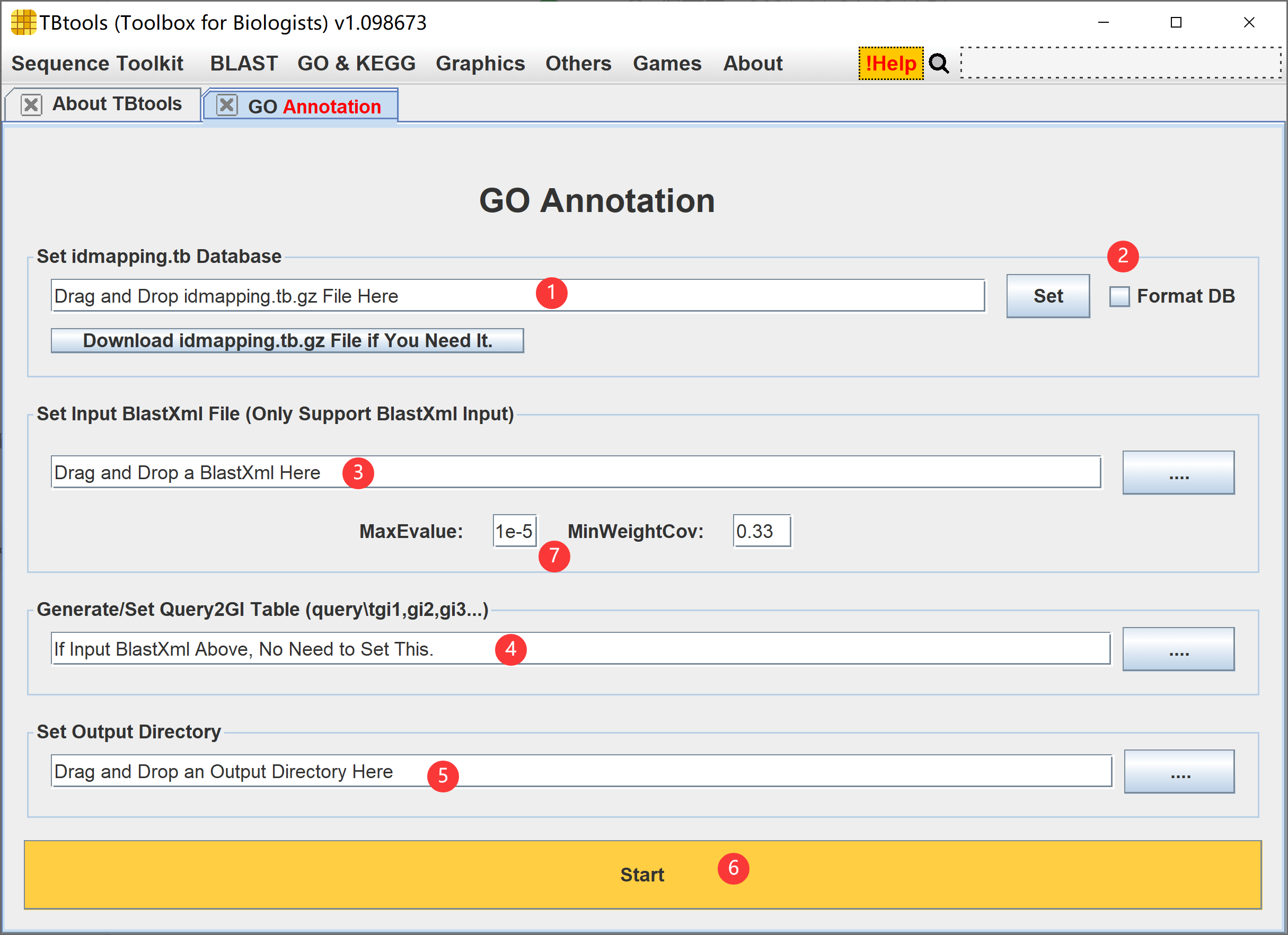
这一功能的使用简单:
设置输入的 ID mapping 数据库文件,一般用户可以从下述链接下载
ftp://ftp.proteininformationresource.org/databases/idmapping/idmapping.tb.gz
也可以直接下载我放在网盘的文件 其他文件下载如大型插件/数据库链接。注意文件交大
Format DB,意思是“是否格式化数据库”,如果用户输入的是 idmapping.tb.gz,那么需要勾选这个选项,从而使 TBtools 格式化对应数据库并生成“idmapping.tb.gz.DB”文件。后续 .DB 文件可直接用做 ID mapping 输入(若输入.DB 文件,那么不勾选 Format DB 选项)。简单来说,一旦有了 .DB 文件,原始的 idmapping.tb.gz 文件可以删除,至少节省 90% 的硬盘空间。
- 设置输入的 BLAST 比对结果,要求 XML 格式,一般建议直接全部蛋白比对到 Swissprot 蛋白数据库(本地就可以了)或者 NR 数据库。注意,3. 和 4. 只需要设置一个就可以了。
- 如果不是使用 BLAST 比对,那么可能无法得到 XML 比对结果,那么可以输入制表符分隔文件,两列,格式为“基因ID\tGI号或Accession号或uniprotID”,比较简单。
- 设置输出文件路径,注意给到输出文件路径,拖拽输入的,应补齐输出文件名。
- 点击 Start 即可开始注释,一般需要一点点时间。
- 如果输入的是 XML 比对结果,可以调整两个参数,筛选比对结果,一般不建议调整。
当然,现在一般建议直接使用 eggNOG-mapper 在线注释功能,速度快,而且也全面,感兴趣的应该了解零基础快速完成基因功能注释 / GO / KEGG / PFAM…。

