写在前面
目前基因组测序和组装成本几乎已经到任何一个课题组都可以单独负担的价码,大量物种的基因组序列被测定和释放。与此同时,对应的基因结构注释信息文件,如GTF或GFF3文件等,也可公开下载。
对于绝大多数要使用这些公共资源的研究人员而言,有了这两个文件就足够了。但想象总是美好,现实却常常骨感。物种基因组很多,基因组序列质量不高的同时,基因结构注释信息文件更加不规范,往往直接影响下游数据分析。我最近在看一些数据,也就遇到类似的问题。如果规范化一个GFF3文件,补充对应的信息,使得该文件能够尽可能适应各类下游数据分析,更或者方便进一步分析使用。
做了大概检索,发现几乎没有同类功能的工具,除了一个perl脚本集合(github搜索 aget )或许可以做类似工作外,确实没发现相关功能的软件实现,尤其是…又还有界面。(Emmm,我还是会乐于承认功能已有实现的软件存在,当然可能是就算啥也没有,我还有 JIGplot 绘图引擎。不会像某两三个团队,似乎有意回避 TBtools,无视他人科研贡献。 :D - PS: 好彩 TBtools 文稿,尤其是预印本、github和一些早期视频讲演早早放到网上去了,不然就要被“李鬼打李逵”了)
回到主题,花了点时间,写了 GXF Fix,然后放到 TBtools 主程序,主要用于修复基因结构注释文件中的缺失部分(基于文件中已有信息)。
常见基因结构注释信息文件问题
基因结构注释信息文件,
- GENE:有不少注释信息缺少 gene feature,比如一些很粗糙的,直接转录组有参考组装结果 gtf 文件
- mRNA:存在一些基因结构注释信息文件,缺少了mRNA feature,只有exon和cds,或者只有cds。这类文件对于一些转录组分析流程,如STAR align - stringtie这个流程来说,可能直接无法动。
- UTR:只有极少数物种会提供 UTR区间,一般是只给出exon和cds,甚至只有mRNA和cds,剩下的 UTR 信息其实是很可能存在的,但是得用户自己去做坐标计算。有 UTR 区间的好处就是…知道一些位点的绝对可靠序列特征信息,如 miRNA 靶位点等。
- 排序:存在不少基因结构注释信息文件的排序混乱
- ….:当然,还有很多比较坑爹的情况
所有这些,都可能影响下游分析。如果有一个方便快捷的功能就好了。
GXF Fix 修复基因结构注释信息文件 - GTF/GFF3
我已经很久没有写出这么符合 TBtools 开发理念的功能了。我们的口号是 -IOS~:
- Input
- Output
- Start
于是,功能界面如下,通过菜单跳转。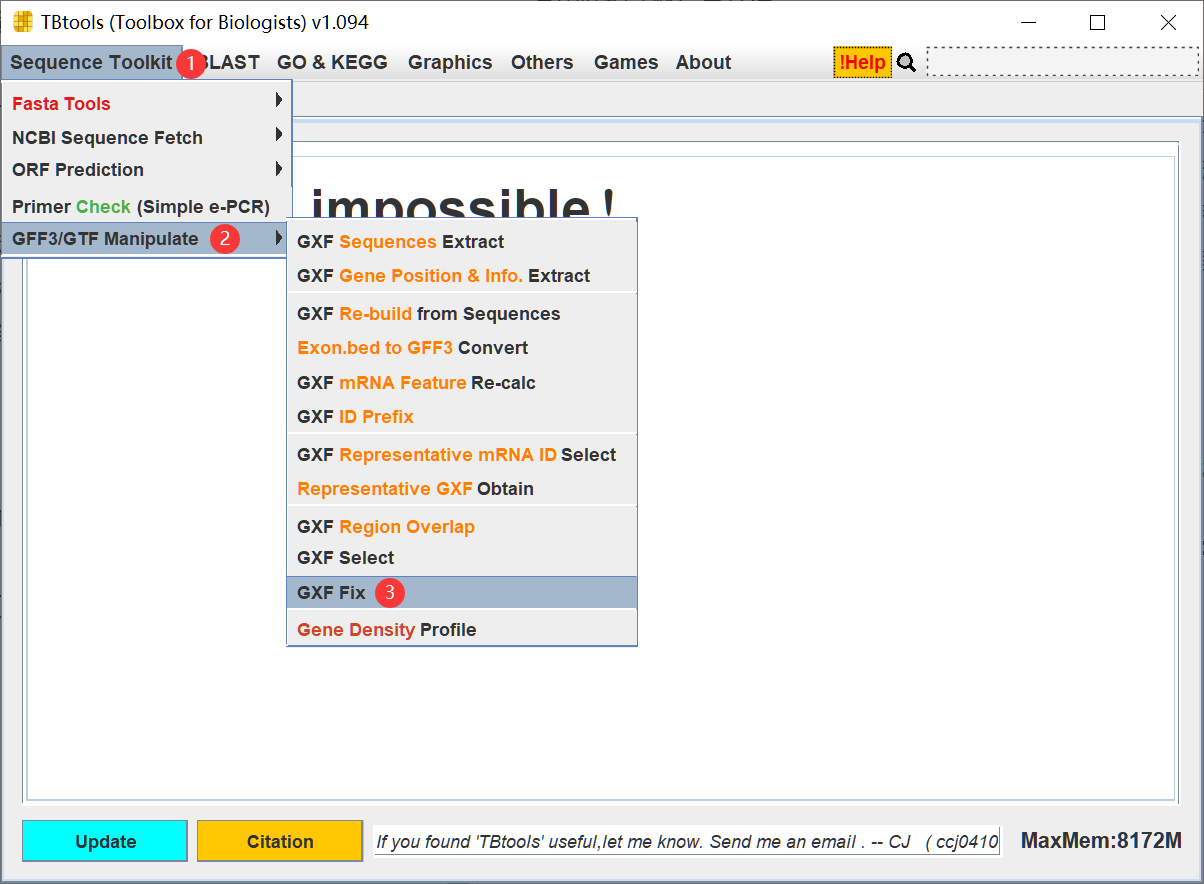
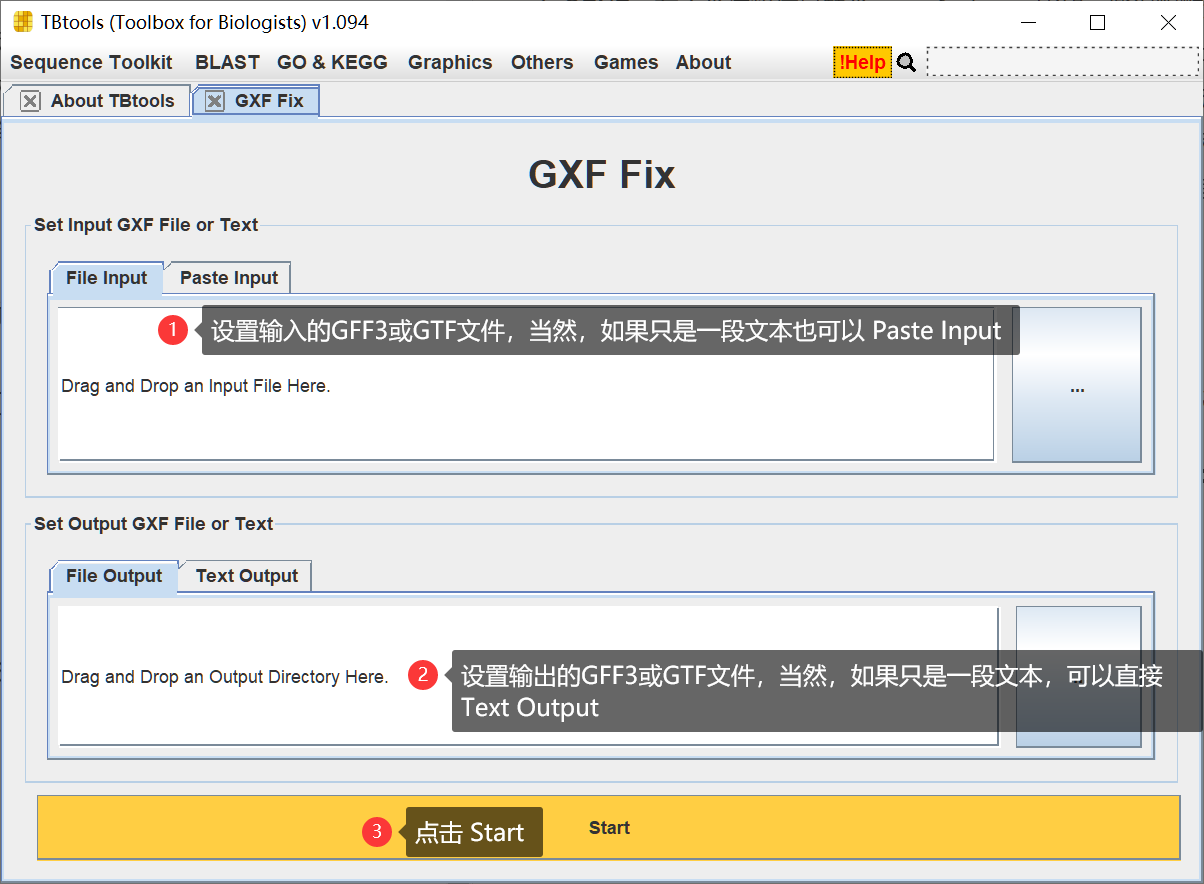
使用实例之一 - 拟南芥
Emmm,拟南芥的基因结构注释信息文件,可以说是非常全面….我们可以跑跑看。
内容对比如下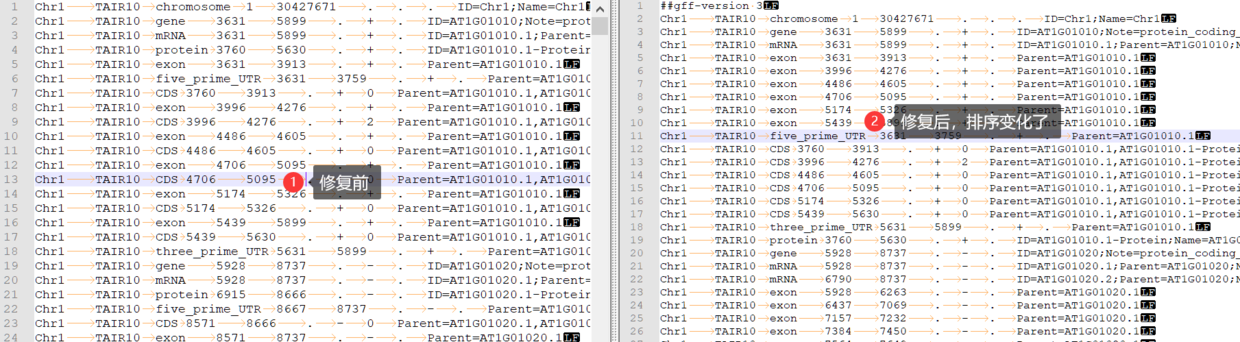
整体行数,完全没变化…
因为其实就没啥好修复的,这个注释信息很好。
使用示例之三 - 香蕉
香蕉的基因结构注释质量有待提升,不过具体内容上都还可以。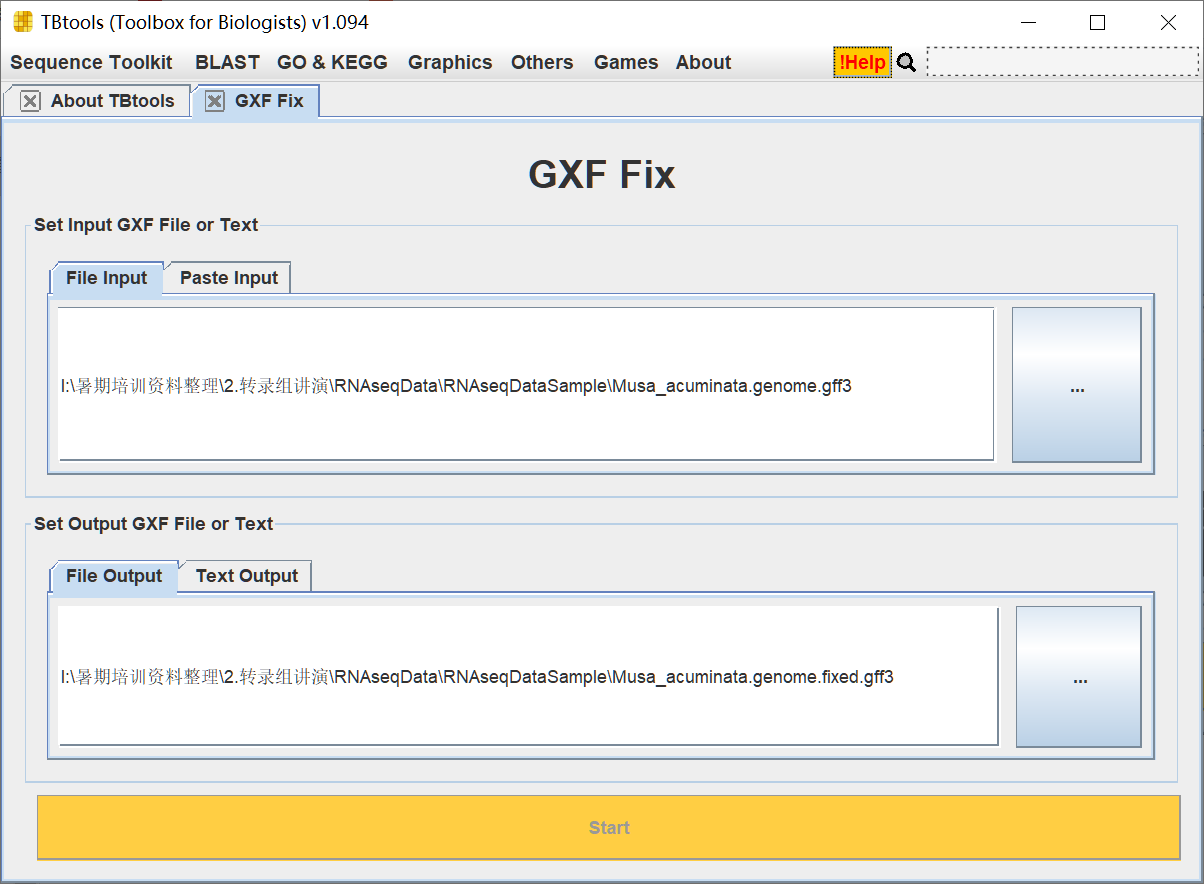
内容对比如下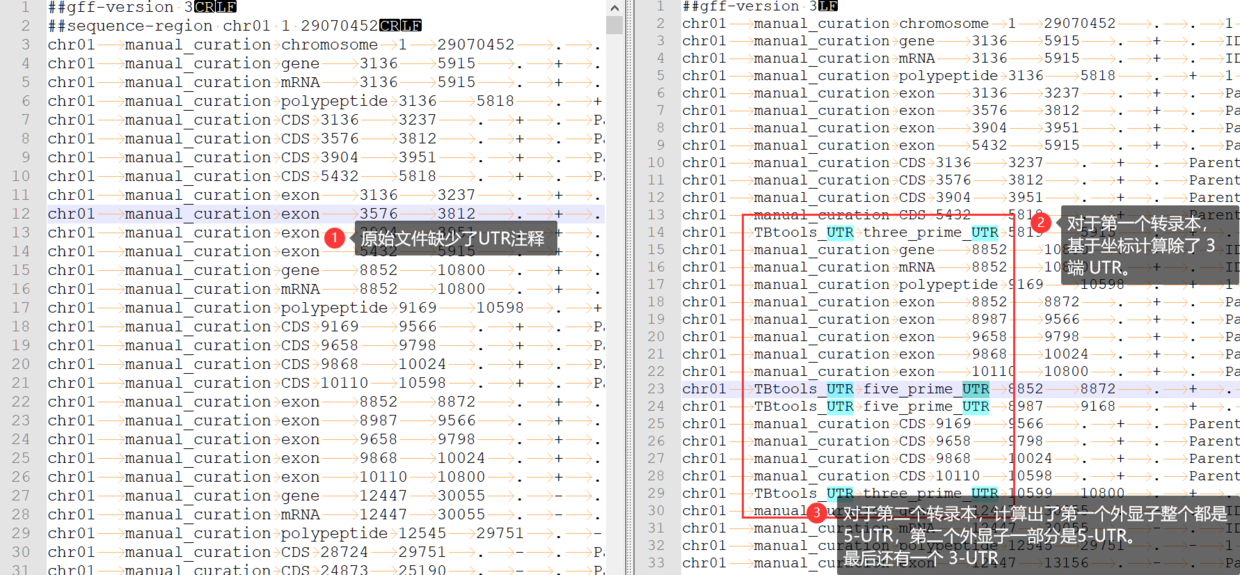
整体行数变化较多,毕竟 UTR 还是有不少。
使用示例之三 - 粗糙注释
有一些注释信息文件,还是比较粗糙的。比如缺少 gene,甚至 mRNA feature都没有。
# stringtie --merge -G F:\RNA-seq\algnment\sme.gff3 -o merged.stringtie.gtf WT.cd1_1.fq_clean.fq.pos.sorted.assembly.gtf WT.cd2_1.fq_clean.fq.pos.sorted.assembly.gtf WT.unc1_1.fq_clean.fq.pos.sorted.assembly.gtf WT.unc2_1.fq_clean.fq.pos.sorted.assembly.gtf# StringTie version 2.1.4SMEL3Ch00.00004 StringTie transcript 42 1079 1000 - . gene_id "MSTRG.1"; transcript_id "MSTRG.1.1";SMEL3Ch00.00004 StringTie exon 42 717 1000 - . gene_id "MSTRG.1"; transcript_id "MSTRG.1.1"; exon_number "1";SMEL3Ch00.00004 StringTie exon 836 1079 1000 - . gene_id "MSTRG.1"; transcript_id "MSTRG.1.1"; exon_number "2";SMEL3Ch00.00004 StringTie exon 97 210 1000 - . gene_id "MSTRG.1"; transcript_id "SMEL_000g000120.1.01"; exon_number "1";SMEL3Ch00.00004 StringTie exon 484 717 1000 - . gene_id "MSTRG.1"; transcript_id "SMEL_000g000120.1.01"; exon_number "2";SMEL3Ch00.00004 StringTie exon 836 1079 1000 - . gene_id "MSTRG.1"; transcript_id "SMEL_000g000120.1.01"; exon_number "3";
直接进行修复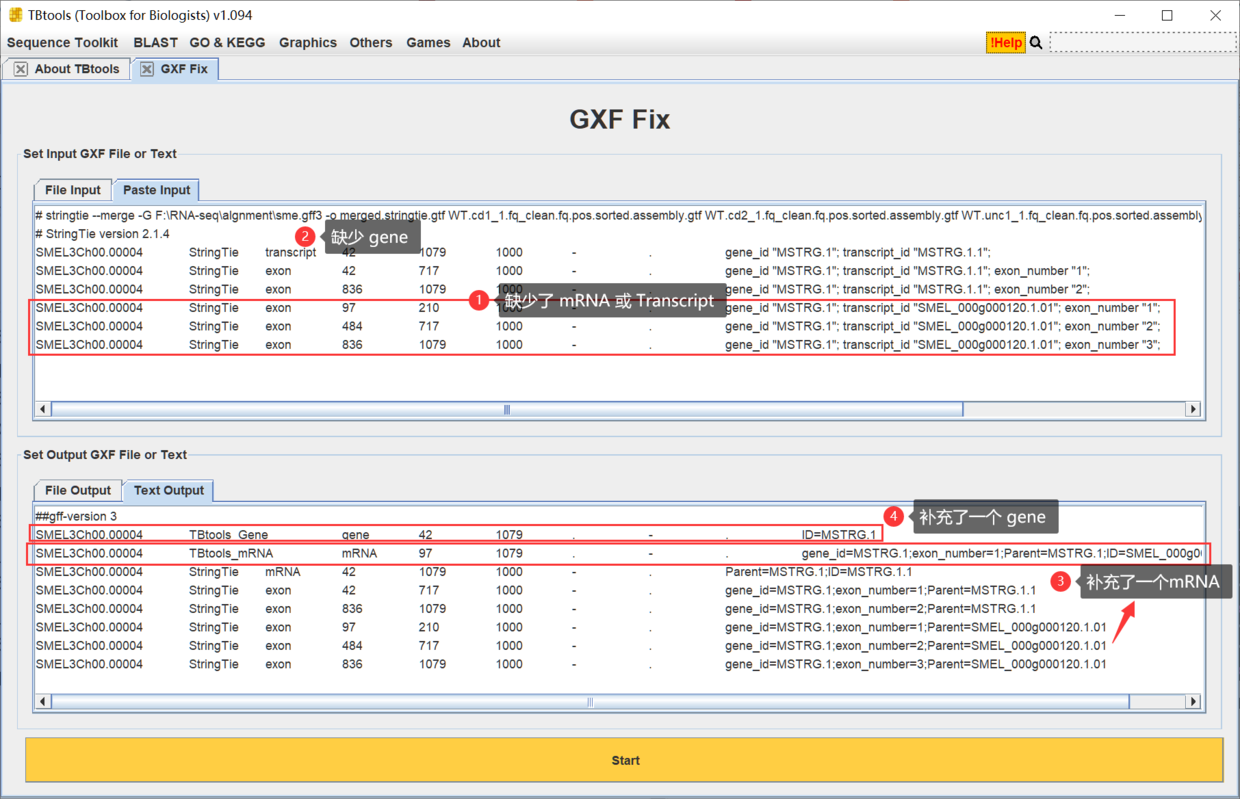
写在最后
Emmm,功能还是很不错的。我觉得这个应该会成为一个常用功能吧。尤其是,越来越多不够规范的 GTF 或者 GFF3 文件出现之后。

