写在前面
近日在鼓捣课题的过程中,遇到了一些数据整理需求。需要快速统计物种的序列特征情况,比如基因,转录本,外显子,内含子,CDS,UTR等。但我们其实都清楚,很多物种的基因结构注释信息比较粗糙,所以前面我写了一个功能GXF Fix,详细见《GXF Fix 修复 / 优化基因结构注释信息文件 - GTF/GFF3》。说实话,我觉得这个功能还是比较有用的。
既然Fix有了,那么就可以搞“Stat”,于是今天主要介绍GXF Stat。
看看结果文件先
Emmm,这个功能说来也简单,就是做一个信息整理,结果文件如下。
看个拟南芥的,我们知道,拟南芥的注释很全面,完全不需要Fix,统计结果如下。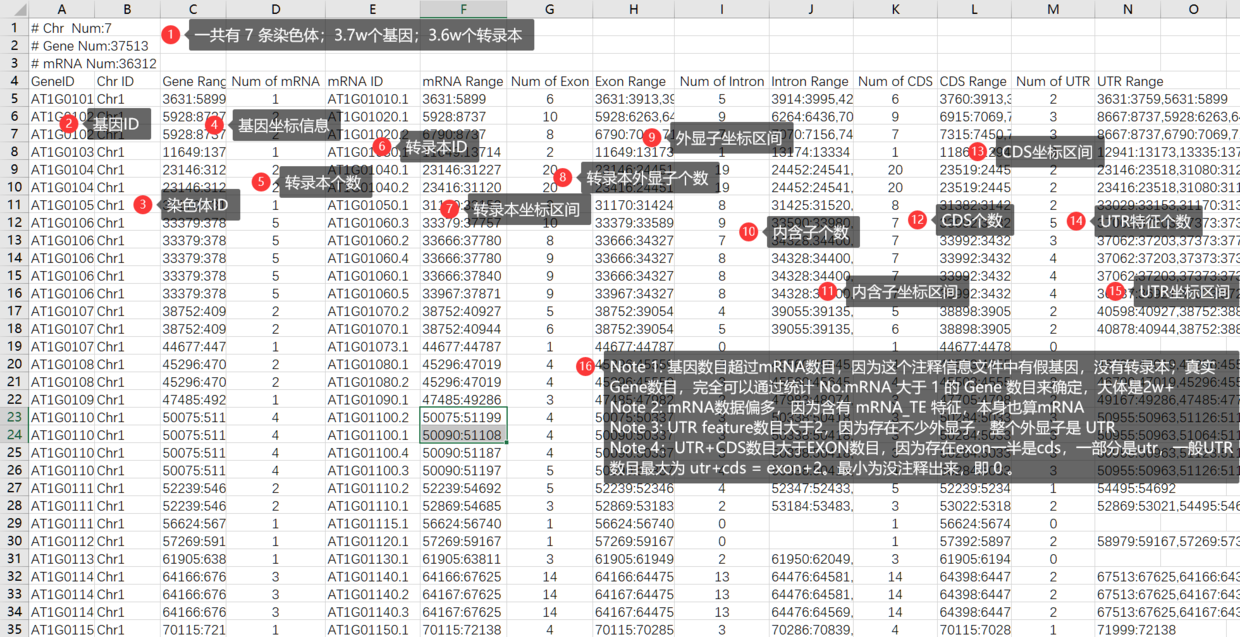
看个香蕉的统计结果,就相对简单,因为不存在 TE 的注释,也没有直接的假基因的特征标识。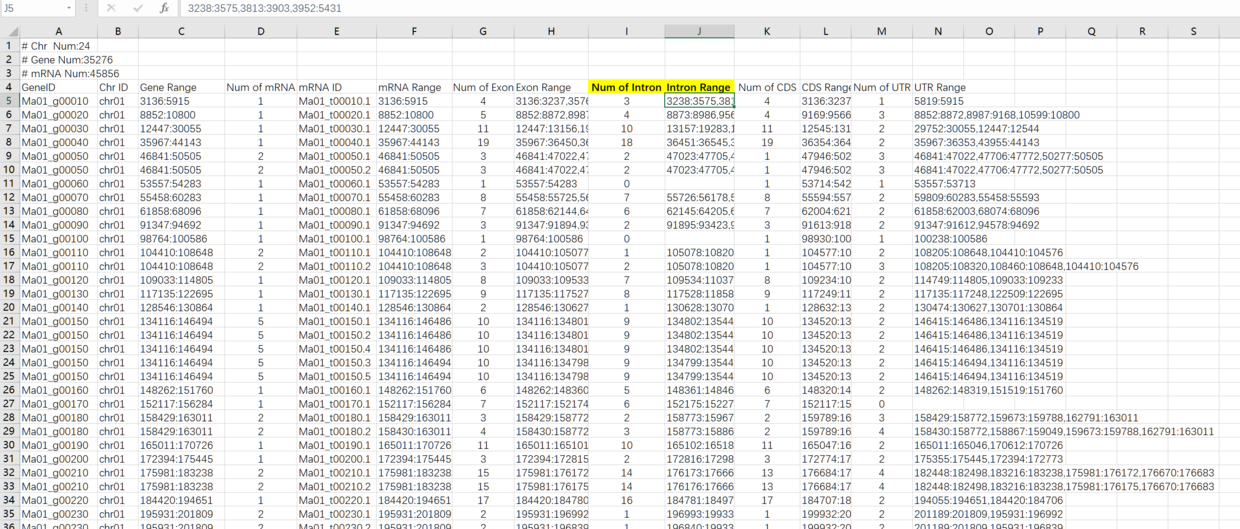
整体上,我们可以看到,GXF Stat的统计结果,包括:
- 染色体数目
- 基因数目
- 转录本数目
- 基因ID
- 染色体ID
- 基因在染色体上的坐标跨度
- 每个基因的转录本个数
- 每个转录本的ID
- 每个转录本在染色体上的坐标跨度
- 每个转录本的外显子数目
- 每个外显子在染色体上的坐标跨度
- 每个转录本的内含子个数
- 每个内含子在染色体上的坐标跨度
- 每个转录本的CDS 特征个数
- 每个转录本的CDS 特征在染色体上的坐标跨度
- 每个转录本的UTR 特征个数
- 每个UTR 特征在染色体上的坐标跨度
写在最后
多少还是有点失落。尽管我知道现在公众号订阅的人数是 3w+。过去几天分别推了一些推文,其中有一些是不少人会点开看的,也有一些阅读量很低。往往,阅读量很低的,反而是我个人更为喜欢的推文。
想来想去,这应该就是推文的局限。
每个人都很忙,要么就是没时间看推文,要么就是只会看标题新奇的推文。或许,这就是不少流量号存在的根本。优质的内容是被需要的,但真正能受到广泛关注的,还是新奇程度。
一个好的推文标题,应该符合推文内容,
而一个获取流量的推文,需要符合大众的猎奇心理。

