这两天在群里发生的一件小事,我觉得整个过程挺适合分享。可以说是一个 老铁用户 和 开发者 以及 软件 三者之间的短篇故事。供大伙了解,或许这是某种程度上,TBtools相关的文化?— CJ - 陈程杰
最近参与了一个基因组项目,顺利的拿到了基因组序列fasta文件和gtf格式的注释文件。当我看到注释文件是gtf格式的时候,我暗自窃喜,因为我知道gtf相比gff3,格式会更规范一些。可我万万没有想到,就是这个我以为规范的gtf文件困扰了我好几天。
先看看我的gtf文件长什么样子(图1B),对比一个规范的文件(图1A),我的gtf文件有两个明显的问题:
1.缺少信息。例如没有gene这一行;
2.基因的命名方式有些随意。
作为TBtools的忠实用户,第1个问题自然好解决,那就是用GXF Fix修复。可第2个问题怎么办呢?
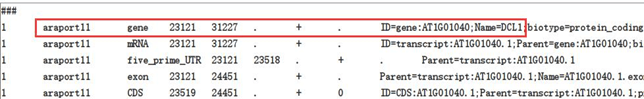
图1A 拟南芥gff3文件
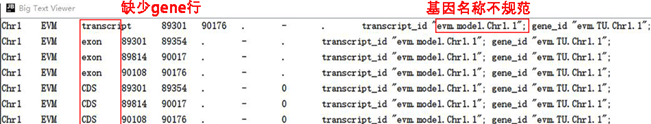
图1B 我的gtf文件
这时刚好TBtools交流群里有个同学问相关问题,然后我顺藤摸瓜,在Batch String Replace找到了替换功能,用法也是符合TBtools的一贯容易操作的特性。首先输入需要替换的文件;然后输入需要替换的名称,因为需要用制表符分隔,我一般在excel里填好,然后另存为txt格式,直接拖进Replace Map里;最后设置输出文件,Start开始即可。在经历了1个多小时的等待后,大功告成。
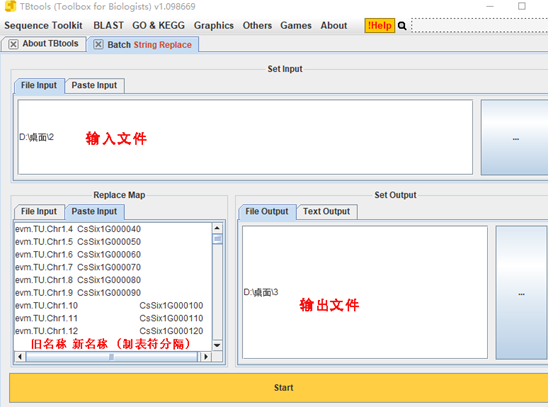
图2 Batch String Replace批量替换
可是,当我打开替换后文件后,发现从第10个基因名起便开始出现错误,例如我想要“00110”,替换后变成了“00101”。于是我向CJ求助,他回答是因为重复替换的问题,接着他在Batch String Replace增加了一个“Full Word Match Mode”新功能(默认勾选)。有了这个功能,就可以完美替换了。同时,为了解决Batch String Replace速度较慢的问题,他又写了GXF Rename(图3),用法与Batch String Replace一样,但不同之处在于速度,2万个基因名称的批量替换,GXF Rename一秒钟搞定,String Replace得1个多小时。
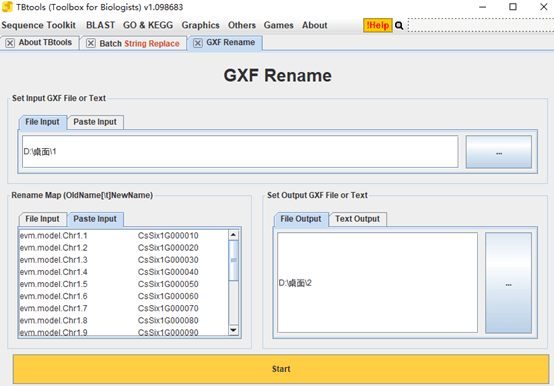
图3 GXF Rename批量重命名
但是,事情还没有结束。因为我的gtf文件没有Name,导致GXF Fix修复后的文件gene行也没有Name(图4),这样的文件导入到可视化软件里不显示基因名字,所以我就想添加上Name列。我能想到的方法也很简单,就是将ID=CsSix1G000010替换成ID=CsSix1G000010;Name=CsSix1G000010。于是我又打开了GXF Rename,但是,它没有完成替换,而String Replace却可以。虽然我的问题解决了,但我还是想知道这一问题的答案,于是再次求助CJ。CJ测试后的结论是:GXF Rename针对的是标签内部的值,是单元,是重命名;而String Replace针对的是String,任何字符串都在考虑范围内。对于我这种生信小白,这句话并不容易理解。但就我使用的经验来说,GXF Rename速度快,2万个基因名称的替换也是毫秒级完成,但一些复杂格式的替换无法完成;而Batch String Replace,速度相对慢一些,但包容性更强。
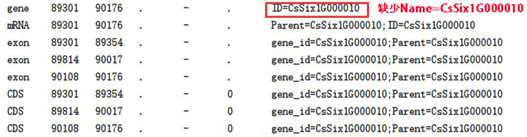
图4 GXF Fix修复后的文件gene行只有ID
最后做个总结,如果你需要批量替换GFF3/GTF的基因名,首先试试GXF Rename,反正速度快。如果不行的话,就使用Batch String Replace,速度会有点慢,但毕竟可以解决问题,这点等待可以接受。当然了,如果不放心的话,可以先少量替换,看看结果。
最后,我想分享个故事。一个人点了份小龙虾外卖,收到后发现包装盒上有张纸,上面写着:亲爱的主人,龙虾用啤酒炒一下味道更鲜美哦!他觉得很诧异,小龙虾还有这种做法,好在也不麻烦,于是他倒了点啤酒,炒了一下。结果他发现,味道还真不错。那么,到底是啤酒炒后的小龙虾好吃了,还是因为他自己参与了这个过程,觉得自己做的好吃呢?或许很大原因是他自己参与了做饭的过程。同理,经过这次的经历,TBtools对我而言又有了不一样的意义,也希望TBtools能够带给大家不同的意义!

