我原本是想给这个工具取个名字叫做Amazing TreeViewer,但我还是无法下定决心,所以他现在叫做TreeTreeTree。翻译成中文,树蜀黍。
写在前面
在很久很久之前,我提到了,在下一篇文章投稿之前,停更。当然,这段时间,我确实还是推送了两三个推文。但没有专门给TBtools的新功能写过一篇推文。于是,今天,推送已经在TBtools中沉睡了有一段时间,却少有人知道或者用上的功能 —- **`TreeTreeTree`**。 提到进化树可视化/注释工具,以我个人的认知,那么现在可能只有两个优秀的工具。一个是ITOLs,一个是ggtree。前者便不过多说明,是一个网页工具。而后者则是你Y叔我大湿兄(公众号:biobabble的运营者)。应是有部分朋友知道我从来就喊你Y叔叫大湿兄,也基本只有我这么喊(我的意思是,不是每个人都能这么喊)。原因有三:
- 大湿兄是潮汕人,所以在广州,我俩还说得上是老乡。
- 我与大湿兄本科是一个学校的,从某种意义上来说,确实存在迷之师兄弟的关系。
- 我不是随便喊喊,而是一直在追赶。
大湿兄的ggtreex目测已经万能,同时甚至他还专门为这个R包整理了一本书,详细见github。ggtree能做的事情太多,这或许是大湿兄的一个追求,那么就是自由。而我往往是另外一个追求,那么是简单。我写代码的开始写的是perl,而我现在依然每天会写一些perl码,虽然都只是单行,不过我依然觉得是不错的操作,因为简单的事情,应该轻而易举地被完成。于是,虽然已经有ITOL和ggtree两个过分优秀的工具,我仍然会有想法自己写一个,主要原因是:
- 我并不喜欢用网页工具,网络延时喜闻乐见
- 继续学习R语言,呃….
到底,我还是简单地写一个,也使用在我们近期投稿的一个文章中。希望一切顺利,早日见刊。
稍微看看
回到主题,先摆两三个小图,大体说明下目前可做成的样子
先来一个直的,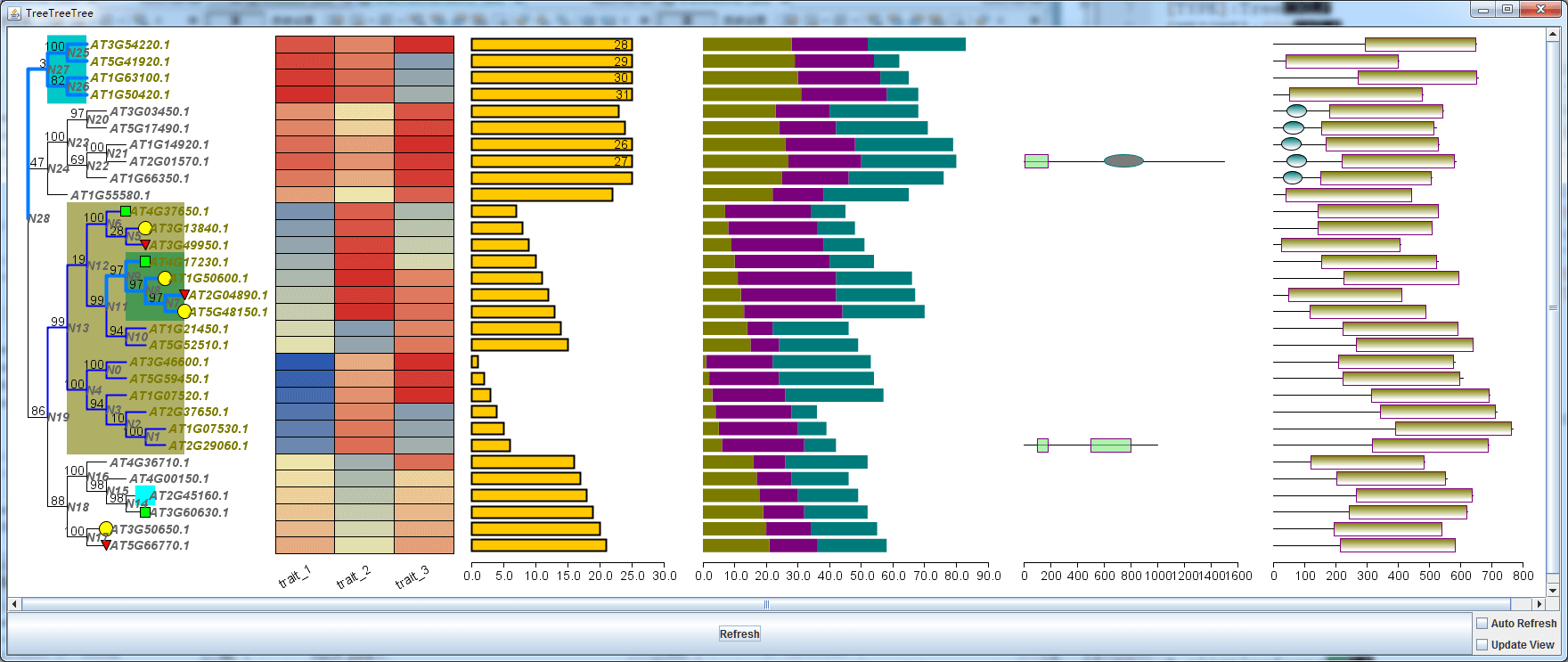
从上面的图片,我们可以发现,事实上,这个功能目前支持:
- 进化树的可视化
- 叶节点图形注释
- 分支背景高亮
- 分支线条风格调整
- 显示不出来的,可以做一定的分支实时调整。
- 热图注释
- 柱形图注释
- 堆叠柱形图注释
- 手动事实自定义的结构域特征或基因结构
- 结构域信息
然后,当然可以掰弯。
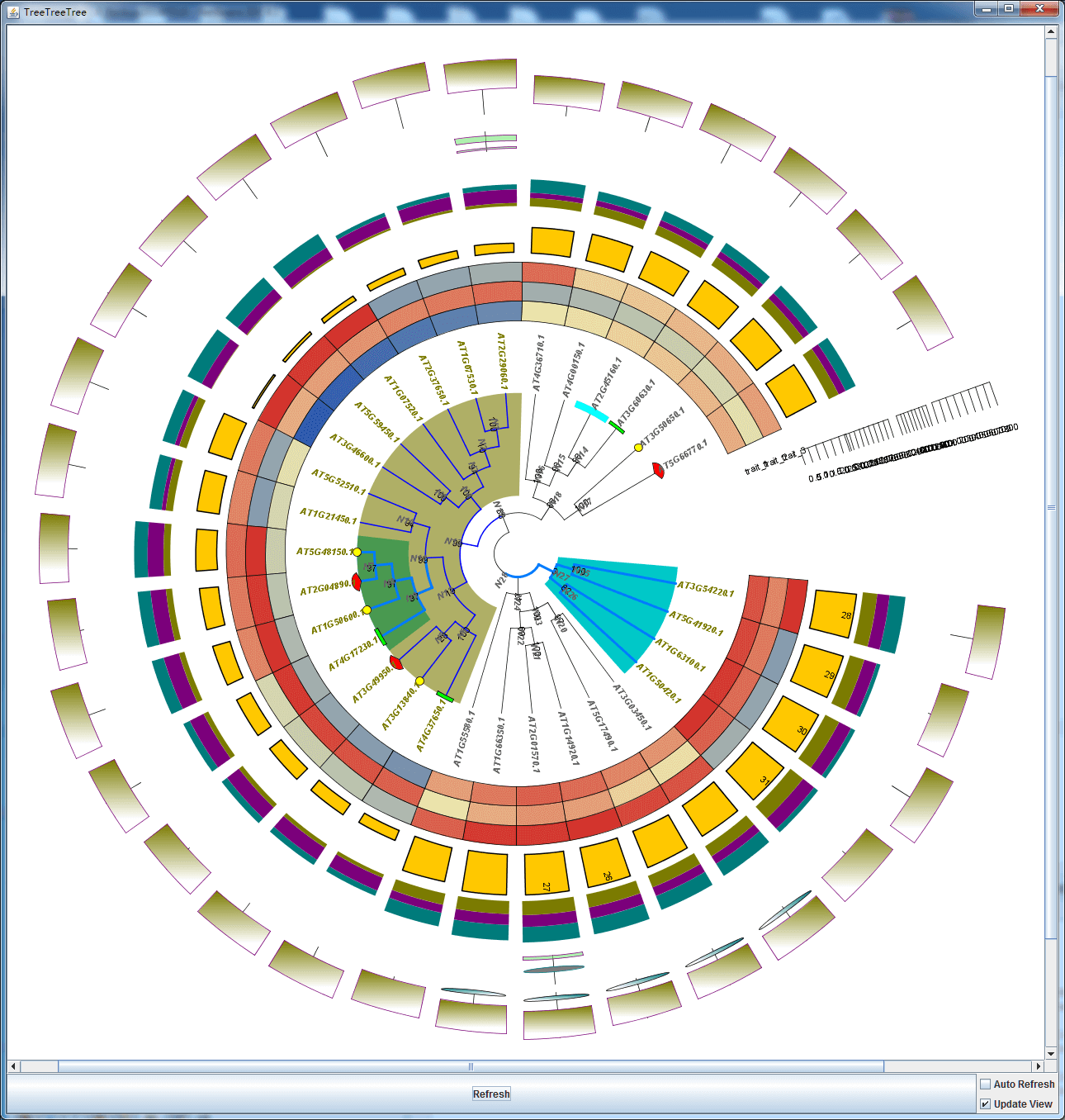
打开工具
当然,这个功能已然在TBtools中。但是要使用这个功能必须,准备一个配置文件。是的,我后来才明白,其实复杂的图形,最好是使用配置文件,而不是纯粹的交互式操作,比如Circos。所以,第一步,新建一个文件(事实上,你需要找个地方下载到一个配置文件模板,然后….按照这个模板去替换或修改参数,从而实现可视化):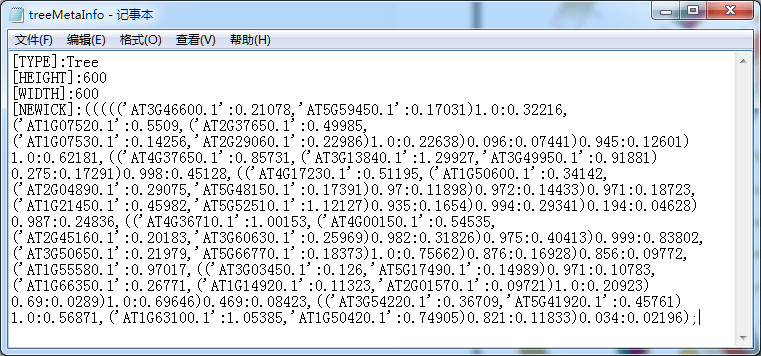
从图上可以看出,这个工具的基本要求就是,一个Newick字符串。随后,保存,并使用TBtools对应的功能进行导入并可视化。
在弹出的对话框中,选择刚才保存的文件即可看到可视化的结果。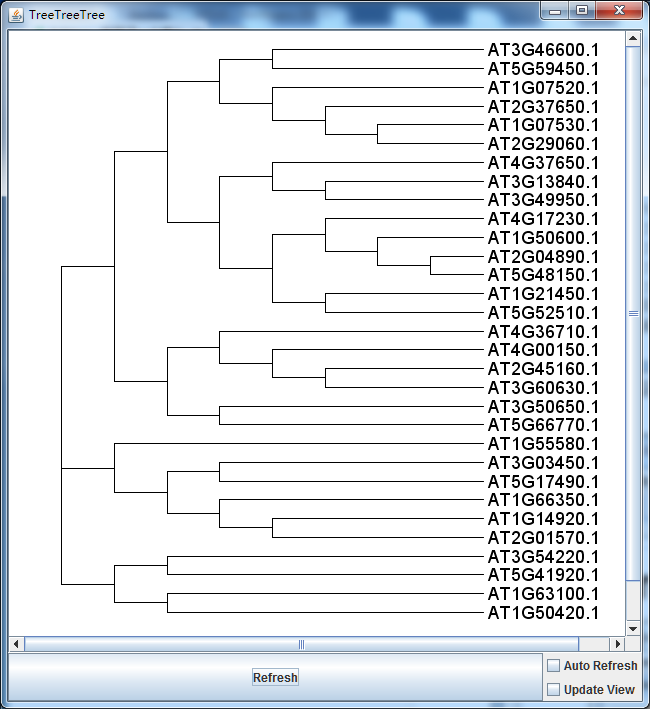
此时,勾选Auto Refresh选项,以方便后续操作。
进行简单的进化树美化
推荐,使用一个具有代码高亮功能的文本编辑器,如果你是在windows下,那么使用Notepad++。在文本编辑器中打开刚才保存的配置文件treeMetaInfo.txt,并使用Bash语法高亮。可看到
[TYPE]:Tree[HEIGHT]:600[WIDTH]:600[NEWICK]:((((('AT3G46600.1':0.21078,'AT5G59450.1':0.17031)1.0:0.32216,('AT1G07520.1':0.5509,('AT2G37650.1':0.49985,('AT1G07530.1':0.14256,'AT2G29060.1':0.22986)1.0:0.22638)0.096:0.07441)0.945:0.12601)1.0:0.62181,(('AT4G37650.1':0.85731,('AT3G13840.1':1.29927,'AT3G49950.1':0.91881)0.275:0.17291)0.998:0.45128,(('AT4G17230.1':0.51195,('AT1G50600.1':0.34142,('AT2G04890.1':0.29075,'AT5G48150.1':0.17391)0.97:0.11898)0.972:0.14433)0.971:0.18723,('AT1G21450.1':0.45982,'AT5G52510.1':1.12127)0.935:0.1654)0.994:0.29341)0.194:0.04628)0.987:0.24836,(('AT4G36710.1':1.00153,('AT4G00150.1':0.54535,('AT2G45160.1':0.20183,'AT3G60630.1':0.25969)0.982:0.31826)0.975:0.40413)0.999:0.83802,('AT3G50650.1':0.21979,'AT5G66770.1':0.18373)1.0:0.75662)0.876:0.16928)0.856:0.09772,('AT1G55580.1':0.97017,(('AT3G03450.1':0.126,'AT5G17490.1':0.14989)0.971:0.10783,('AT1G66350.1':0.26771,('AT1G14920.1':0.11323,'AT2G01570.1':0.09721)1.0:0.20923)0.69:0.0289)1.0:0.69646)0.469:0.08423,(('AT3G54220.1':0.36709,'AT5G41920.1':0.45761)1.0:0.56871,('AT1G63100.1':1.05385,'AT1G50420.1':0.74905)0.821:0.11833)0.034:0.02196);
在文本下方添加,随后直接保存文本。TBtools会定时读取配置文件,并实时刷新可视化结果。当你保存文件时,图片也同步刷新。
[setting]#options:Origin|Equal|Cladogram|Proportionallayout:Equal
其中有几个layout可以选择,比如Proportional。保存文件,即可看到图片刷新了。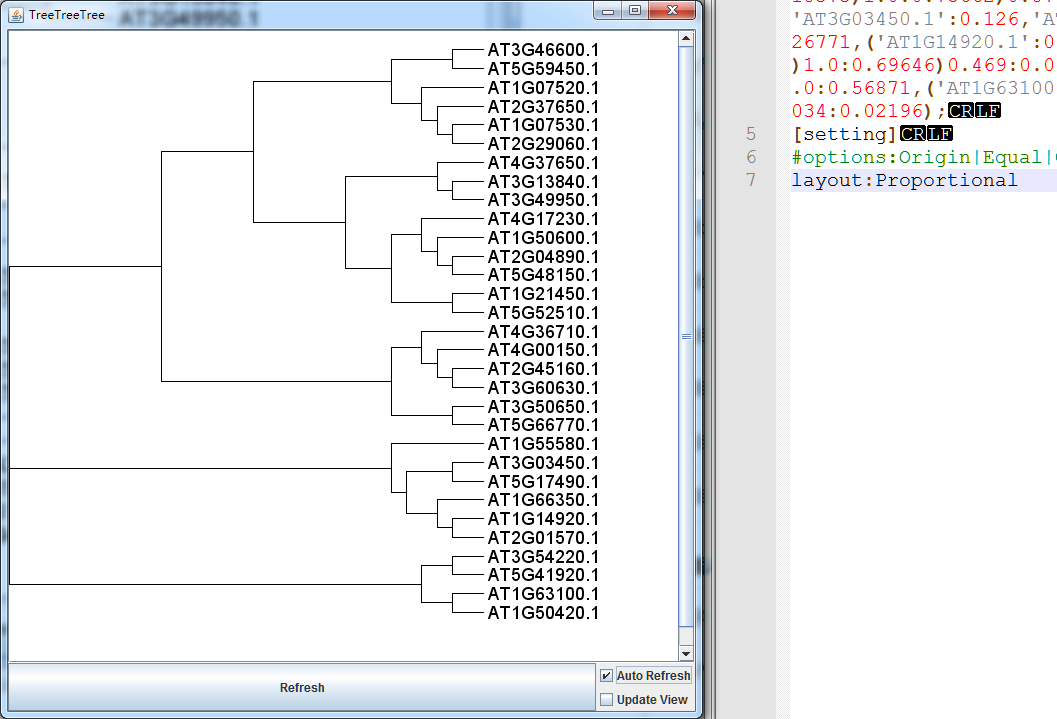
当然,最常见的还是直接使用Cladogram。
类似的,我们可以对一些属性进行修改,比如增加以下内容
# options:yes|no# 显示branchlabel,一般就是bootstrap值branchlabel:yes# 是否显示叶片名字leafname:yes# 叶片文本的一些设置leaffontfamily:arialleaffontbold:yesleaffontitalic:yesleaffontsize:14leaffontcolor:100,100,100
保存可见,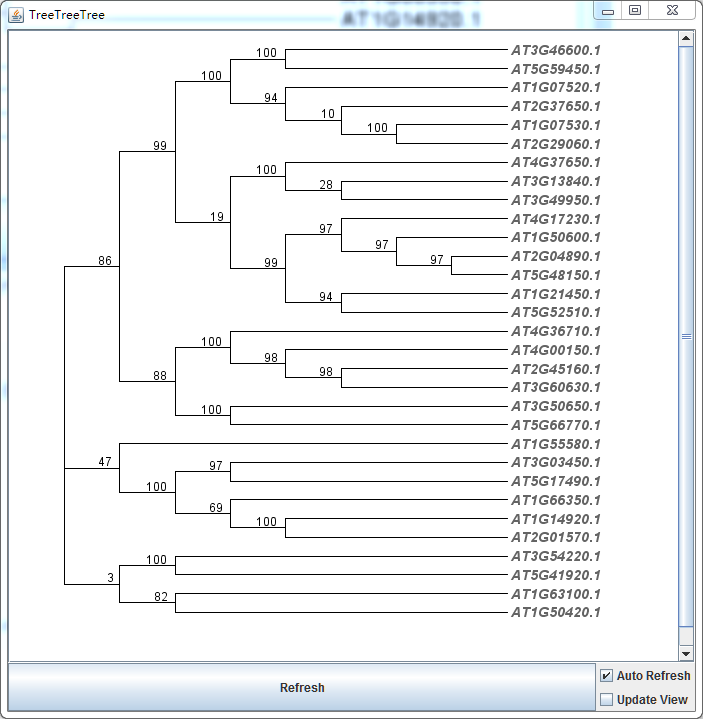
你可以随意调整一些内容,并保存,即可实时看到结果,事实上就相当于,
你使用文本在操作一个进化树的可视化。
但是篇幅有限,这里不做展开。关于可控操作,都已经在配置文件模板中。这类直接给出树部分的配置信息如下。
[setting]#options:Origin|Equal|Cladogram|Proportionallayout:Origin# options:yes|noholdername:yes# options:yes|nobranchlabel:yesleafname:yesleaffontfamily:arialleaffontbold:yesleaffontitalic:yesleaffontsize:14leaffontcolor:100,100,100# alignleaf:yes# polarautopolar:noinnerstep:10startangle:30endangle:330# alignleafalignleaf:no# lineLen###dashLen###lineLen...aligndashpattern:3###2###1
可能值得提的还有一点,亦即,你想要对分支进行上下翻转,上述参数holdername:yes事实上就是显示方便操作的信息。你会看到这个图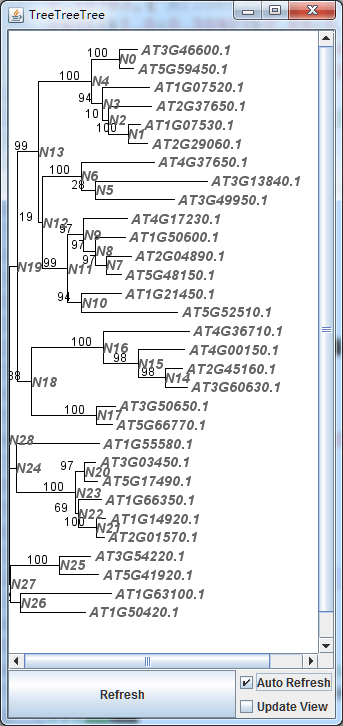
其中N+数值其实就是每个分支节点的名字,你可以在配置文件中添加操作信心,从而实时翻转一些分支,如下
[manipulation]flipnode:N13flipnode:N28flipnode:N22flipnode:N24
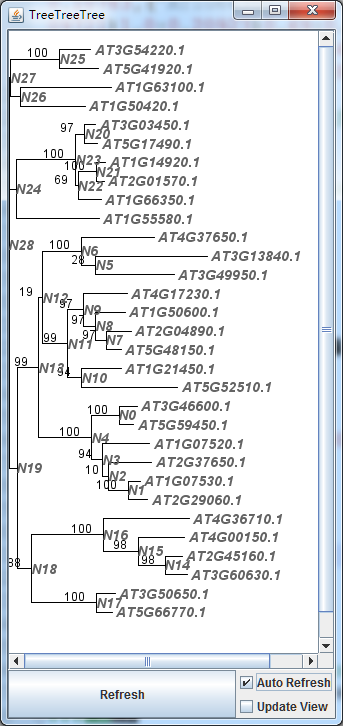
于是,你看到数值结构已经改变。我们似乎忘记对叶节点进行对齐,可以修改上述参数
# alignleafalignleaf:yes# lineLen###dashLen###lineLen...aligndashpattern:3###2###1
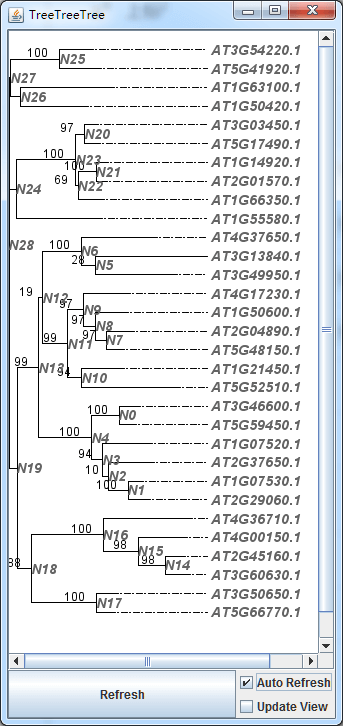
恩,似乎不对齐更好看一点。修改回来。当然,我们希望对一些分支进行高亮,或者是分支线条进行颜色或说风格修改,那么你可以添加或修改以下信息,
###### 进化树分支注释参数#### 首先,定义风格,如Example Style 1即风格1的模式[cladestyle]=Example Style 1=hightLightColor:123,123,0hightLightAplpha:0.6lineColor:0,0,255lineStroke:2leaffontColor:123,0,0=Example Style 2=hightLightColor:0,200,200hightLightAplpha:1lineColor:0,123,255lineStroke:4leaffontColor:123,123,0# 然后,对不同的节点,使用你所想要使用的风格[cladeprofile]N13###Example Style 1N9###Example Style 2N27###Example Style 2
得到如下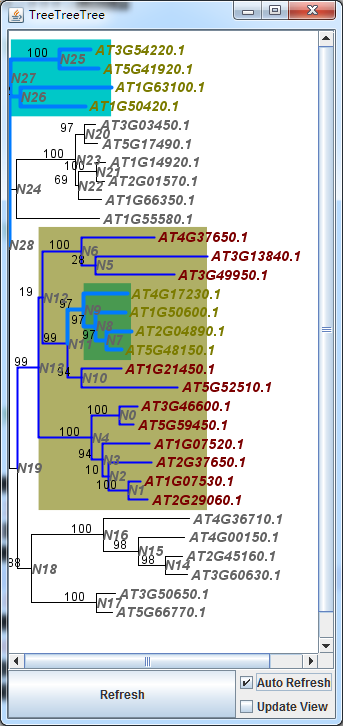
可以看到,有两个分支应用了同样一个Style。当然分支N13遮盖了N9。恩..此处就不关心了….类似的操作,我们会想要对一些叶节点,此处是基因做一些简单的标记,比如三角形,矩形,圆形等,那么可以添加一下内容
###### 进化树叶片注释参数#### 类似的,第一步,定义不同标记的风格[nodestyle]=s1=# Options:Rectangle|Eclipse|RoundRectangle|DonwTriangle|UpTriangle|TowardWestRect|TowardEastRectshape:Rectanglefillcolor:0,255,255size:0.6=s2=shape:Circlefillcolor:255,255,0drawcolor:0,0,0size:0.3=s3=shape:DonwTrianglefillcolor:255,0,0drawcolor:0,0,0size:0.3=s4=shape:Rectanglefillcolor:0,255,0drawcolor:0,0,0size:0.3# 类似的,第二步,对不同的叶节点应用风格[nodeprofile]AT2G45160.1###s1AT3G60630.1###s4AT3G50650.1###s2AT5G66770.1###s3AT4G37650.1###s4AT3G13840.1###s2AT3G49950.1###s3AT4G17230.1###s4AT1G50600.1###s2AT2G04890.1###s3AT5G48150.1###s2
得到如下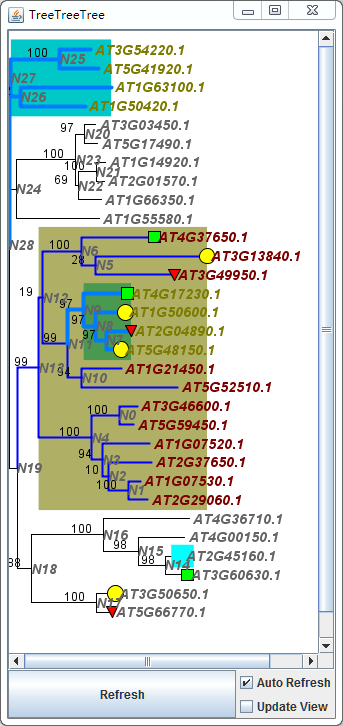
添加热图信息
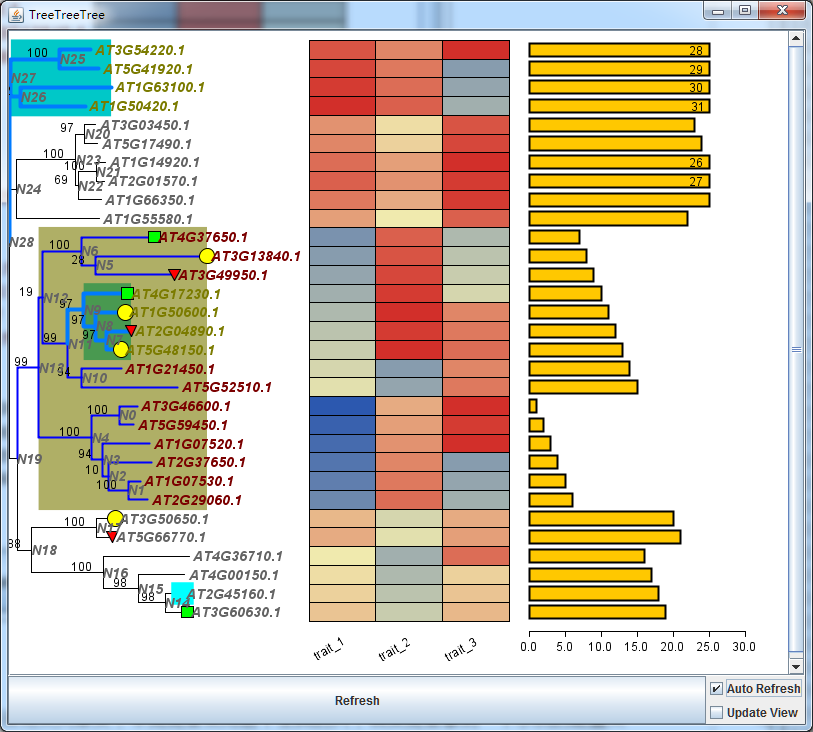
简单的进化树可视化,其实并不是我想做的事情。我是希望在在进化树上下文的基础上,显示一些信息。而这些信息能够实时跟随进化树的变化而做对应的位置调整。比如,节点翻转,那么热图也跟着翻转。之前TBtools的Amazing Simple HeatMap在做类似的事情是,让我比较失望,所以此处,TreeTreeTree必须是我得到补偿。所以他也支持热图信息,你可以按照以下格式田间内容,从而绘制对应的热图。
######################################## 添加热图 ######################################### 定义类型为热图,图形的起始坐标和终止坐标,注意Y坐标是有树决定的[TYPE]:HeatMap[STARTX]:300[ENDX]:500# 定义一些风格,比如颜色[style]=trait_1=firstcolor:44,88,175secondcolor:240,234,174thirdcolor:210,47,42borderwidth:2# 提供数值矩阵,注意,制表符分隔,而不是空格分隔[profile]trait_1 trait_2 trait_3AT3G46600.1 1 21 31AT5G59450.1 2 22 30AT1G07520.1 3 23 31AT2G37650.1 4 24 8AT1G07530.1 5 25 9AT2G29060.1 6 26 10AT4G37650.1 7 27 11AT3G13840.1 8 28 12AT3G49950.1 9 29 13AT4G17230.1 10 30 14AT1G50600.1 11 31 24AT2G04890.1 12 30 25AT5G48150.1 13 31 26AT1G21450.1 14 8 24AT5G52510.1 15 9 25AT4G36710.1 16 10 26AT4G00150.1 17 11 18AT2G45160.1 18 12 19AT3G60630.1 19 13 20AT3G50650.1 20 14 21AT5G66770.1 21 15 22AT1G55580.1 22 16 27AT3G03450.1 23 17 28AT5G17490.1 24 18 29AT1G66350.1 25 21 30AT1G14920.1 26 22 31AT2G01570.1 27 23 30AT3G54220.1 28 24 31AT5G41920.1 29 25 8AT1G63100.1 30 26 9AT1G50420.1 31 27 10
你会得到如下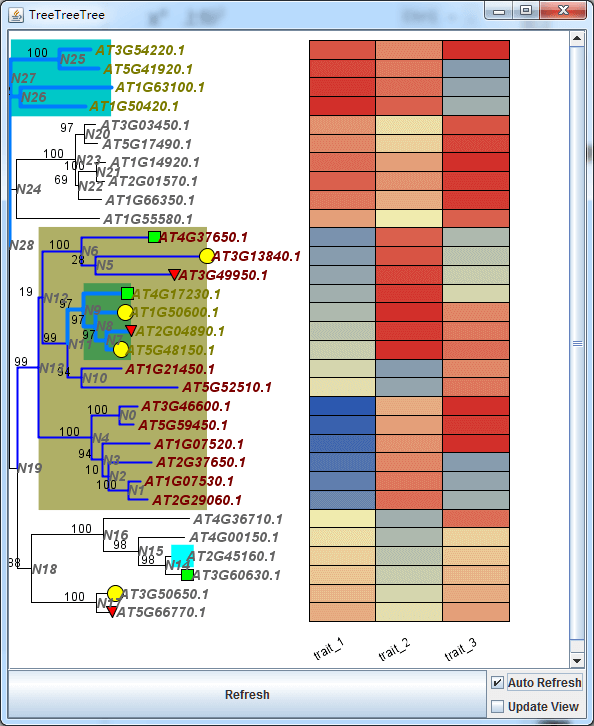
此时,如果你对某个分支,比如N18进行翻转,
[manipulation]flipnode:N13flipnode:N28flipnode:N22flipnode:N24flipnode:N18
当你Ctrl+S保存配置文件的同时你可以看到,热图的行也同样发生了改变。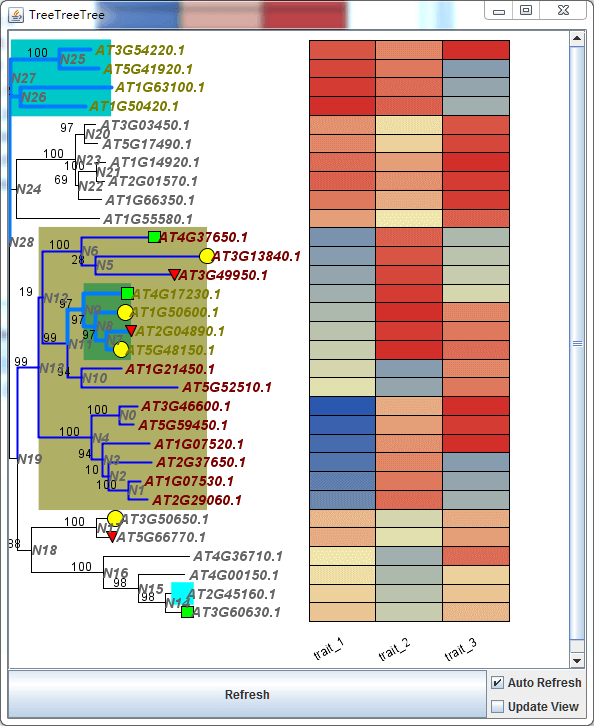
添加柱形图信息
剩下的,我就不贴配置信息了,似乎没有太大意义,毕竟需要使用这个工具的人,确实需要拿到一个示例的配置文件,而你确实会有。热图只是一个测试,我事实上需要的是柱形图,在补充信息文件后,我可以得到
看起来还不错。对于柱形图,其实存在一个参数,限定最高值。当超过这个数值,那么会截断并显示具体数值。
添加堆叠柱形图
有时候,我们有多一维的信息,需要进行展示。此时,堆叠柱形图成为必要。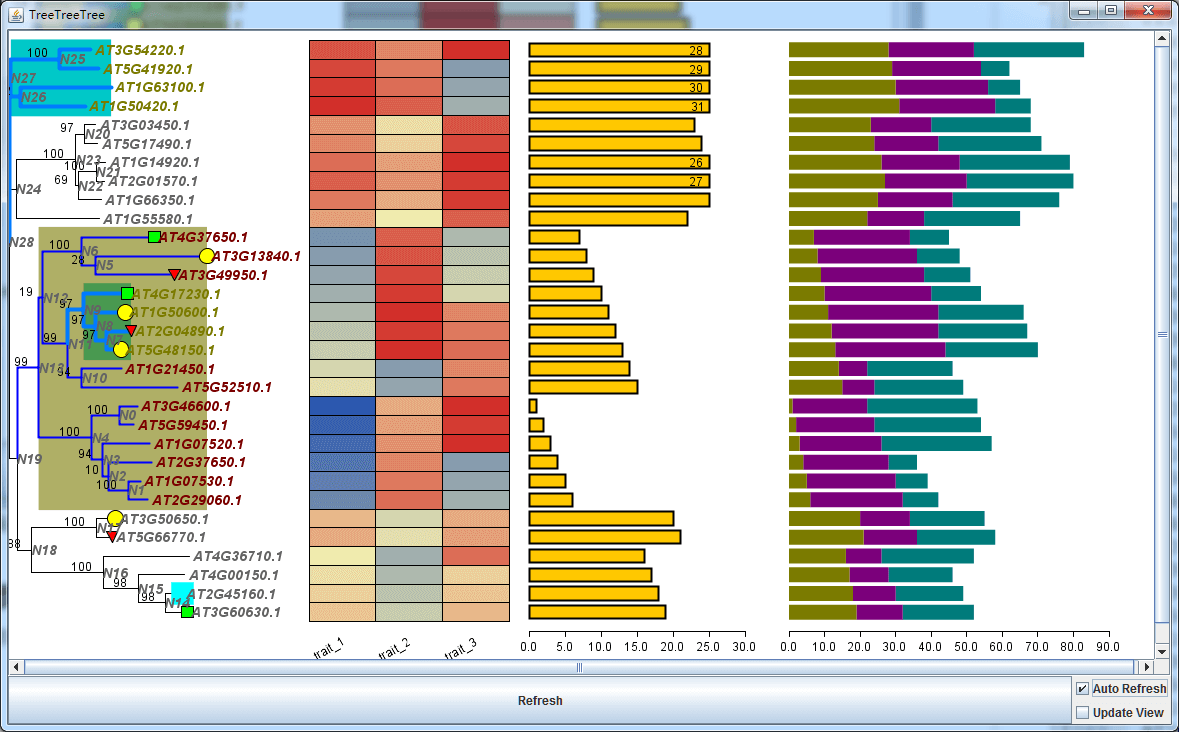
实时手动绘制结构域
这里似乎还是要摆出基本配置信息,如下,添加后保存
#################################################### 添加手动编辑输入的生物序列 ####################################################[TYPE]:ManualAssigned[STARTX]:1140[ENDX]:1380# 同样,先定义结构域的风格[style]=Example Domain 1=# Options:Rectangle|Eclipse|RoundRectangle|DonwTriangle|UpTriangle|TowardWestRect|TowardEastRectshape:Rectangleheight:0.8borderwidth:1bordercolor:123,0,123fillcolor:123,234,123fillalpha:0.6fiilgradient:No=Example Domain 2=shape:Eclipseheight:0.8borderwidth:1bordercolor:0,123,123fillcolor:123,123,123fillalpha:1fiilgradient:No# 对于每一个需要显示结构域信息的序列,给出序列长度是需要的[seqlength]AT2G29060.1:1000AT2G01570.1:1500# 针对序列,应用结构域信息,代替格式是 起始坐标=风格=终止坐标[profile]AT2G29060.1###100=Example Domain 1=180###500=Example Domain 1=800AT2G01570.1###10=Example Domain 1=180###600=Example Domain 2=900
保存,可以看到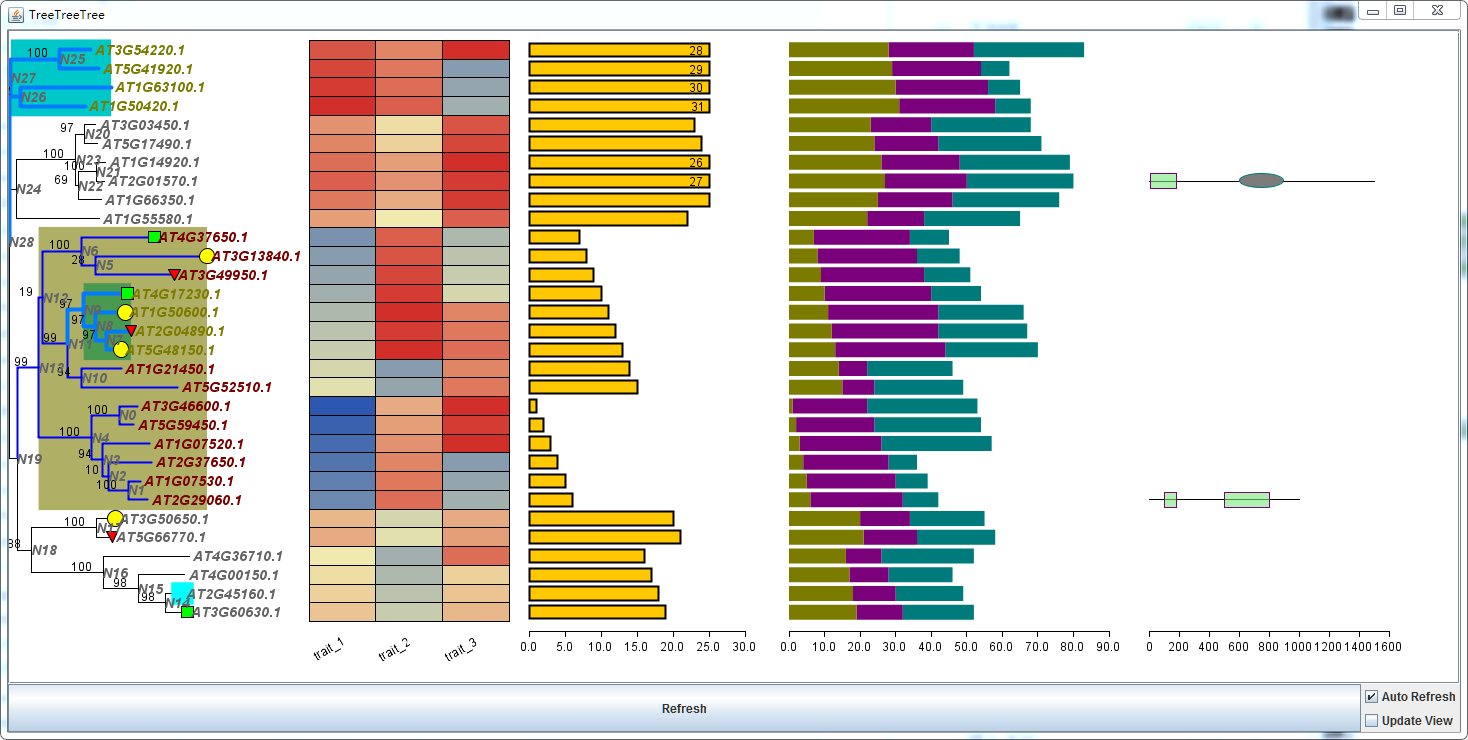
你可以实时针对某个序列,添加一些结构域信息
[seqlength]AT2G29060.1:1000AT2G01570.1:1500AT1G55580.1:1200# 针对序列,应用结构域信息,代替格式是 起始坐标=风格=终止坐标[profile]AT2G29060.1###100=Example Domain 1=180###500=Example Domain 1=800AT2G01570.1###10=Example Domain 1=180###600=Example Domain 2=900AT1G55580.1###200=Example Domain 1=540###600=Example Domain 2=900
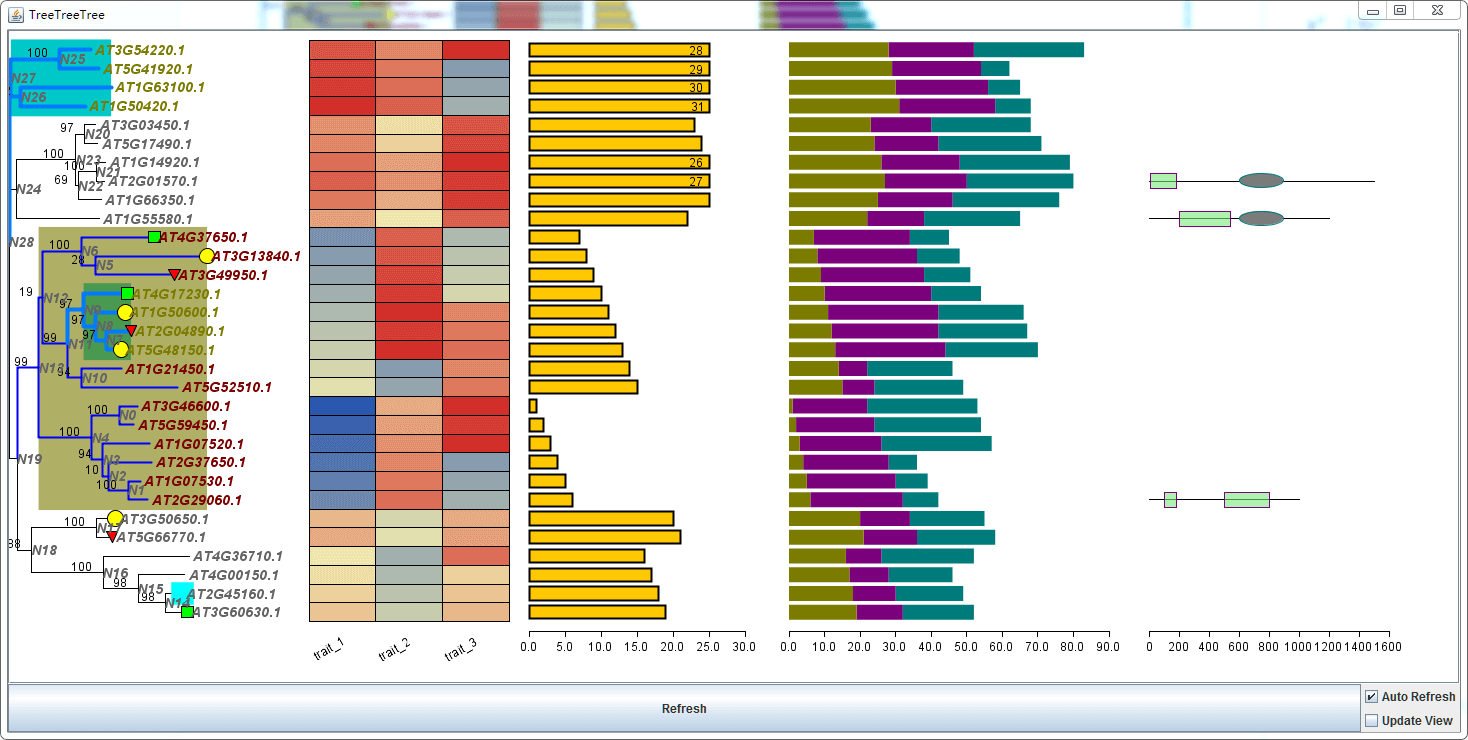
可视化结构域信息
当然了,可能在多数时候,你想要可视化的是一些结构域信息,而不是手动编辑,那么,类似的,你可以田间如下
################################################ 添加结构域以及位置信息 ################################################[TYPE]:Domain[STARTX]:1420[ENDX]:1700[style]=GRAS=# Options:Rectangle|Eclipse|RoundRectangle|DonwTriangle|UpTriangle|TowardWestRect|TowardEastRectshape:Rectangleheight:0.8borderwidth:1bordercolor:123,0,123fillcolor:123,123,0fillalpha:1fiilgradient:Yes=DELLA=shape:Eclipseheight:0.8borderwidth:1bordercolor:0,0,0fillcolor:0,123,123fillalpha:1fiilgradient:Yes[seqlength]AT1G07520.1 695AT1G07530.1 769AT1G14920.1 533AT1G21450.1 593AT1G50420.1 482AT1G50600.1 597AT1G55580.1 445AT1G63100.1 658AT1G66350.1 511AT2G01570.1 587AT2G04890.1 413AT2G29060.1 694AT2G29065.1 630AT2G37650.1 718AT2G45160.1 640AT3G03450.1 547AT3G13840.1 510AT3G46600.1 583AT3G46600.2 453AT3G46600.3 551AT3G49950.1 410AT3G50650.1 542AT3G54220.1 653AT3G60630.1 623AT4G00150.1 558AT4G08250.1 483AT4G17230.1 529AT4G36710.1 486AT4G37650.1 531AT5G17490.1 523AT5G41920.1 405AT5G48150.1 490AT5G52510.1 640AT5G59450.1 610AT5G66770.1 584[profile]AT1G14920.1 169 529 GRASAT1G14920.1 28 94 DELLAAT1G07530.1 393 764 GRASAT1G07520.1 315 692 GRASAT1G55580.1 41 445 GRASAT1G50600.1 227 597 GRASAT1G21450.1 223 593 GRASAT1G66350.1 152 506 GRASAT1G66350.1 32 95 DELLAAT1G50420.1 54 479 GRASAT1G63100.1 274 654 GRASAT2G37650.1 344 713 GRASAT2G04890.1 50 413 GRASAT2G29060.1 318 691 GRASAT2G45160.1 268 639 GRASAT2G01570.1 221 581 GRASAT2G01570.1 44 111 DELLAAT3G46600.1 209 579 GRASAT3G50650.1 195 542 GRASAT3G13840.1 145 510 GRASAT3G49950.1 27 407 GRASAT3G60630.1 243 622 GRASAT3G54220.1 296 650 GRASAT3G03450.1 180 545 GRASAT3G03450.1 45 109 DELLAAT4G37650.1 143 529 GRASAT4G17230.1 155 525 GRASAT4G36710.1 122 485 GRASAT4G00150.1 205 554 GRASAT5G66770.1 217 584 GRASAT5G48150.1 119 490 GRASAT5G17490.1 157 516 GRASAT5G17490.1 34 100 DELLAAT5G52510.1 266 640 GRASAT5G41920.1 40 400 GRASAT5G59450.1 224 598 GRAS
于是你得到了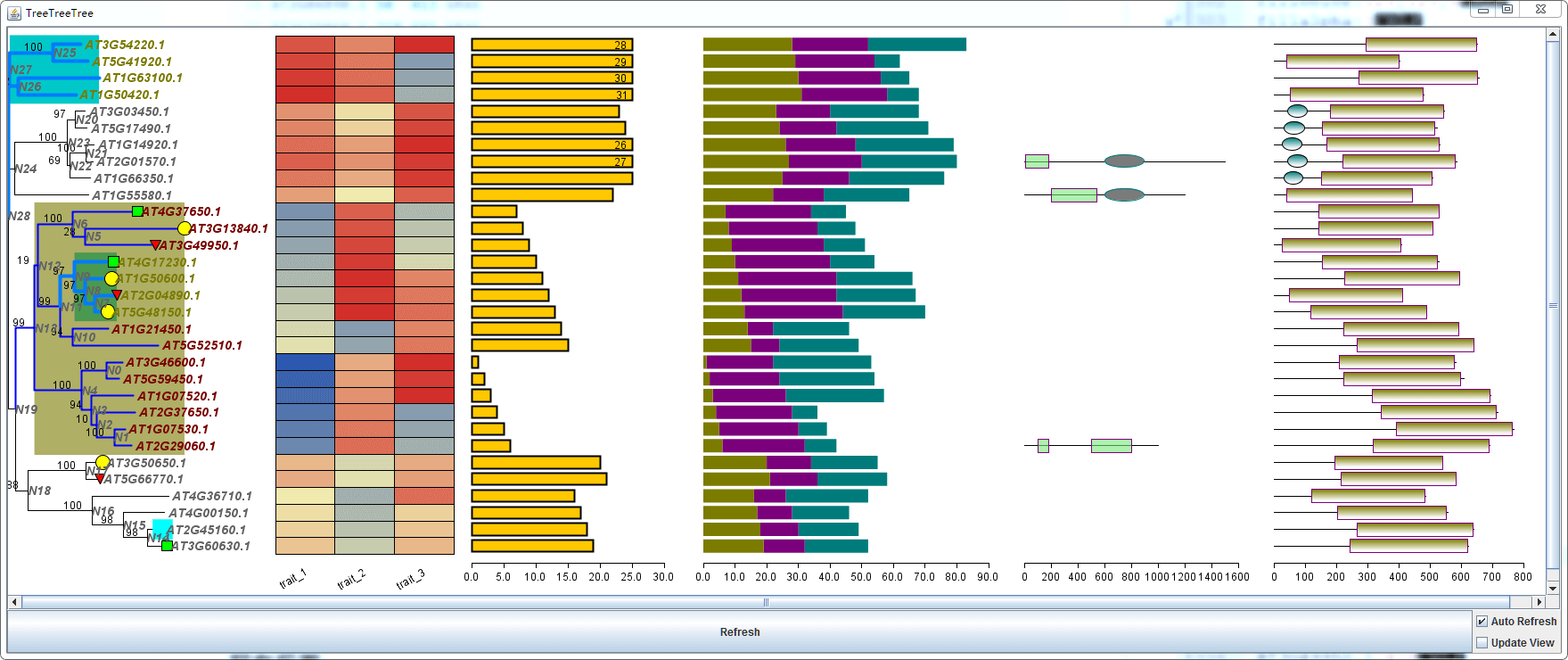
掰弯图片
TBtools的图片,常常是可以掰弯的,你需要调整的参数 ,已经在上面给出了。只需要修改
# polarautopolar:noinnerstep:10startangle:30endangle:330
即可掰弯,不过,掰弯时,一些panel直接的重叠,那时要手动调整StartX和EndX。总的来说,我当然相信,你可以得到下图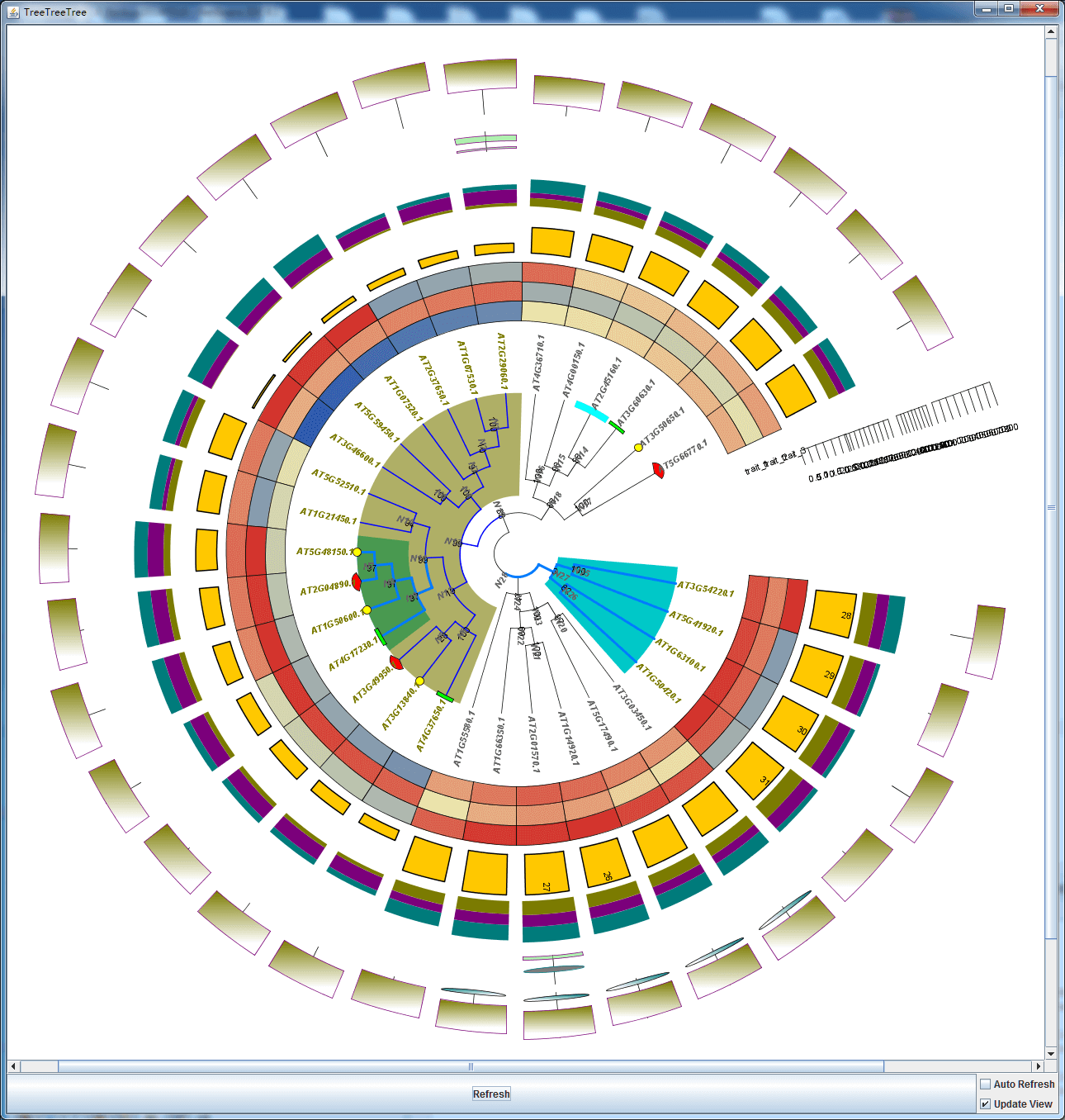
当然了,我完全有理由相信,其实,你需要的只是一个进化树和其中的某一类,那你就只给那一类的信息,并调整好panel的坐标就好了,比如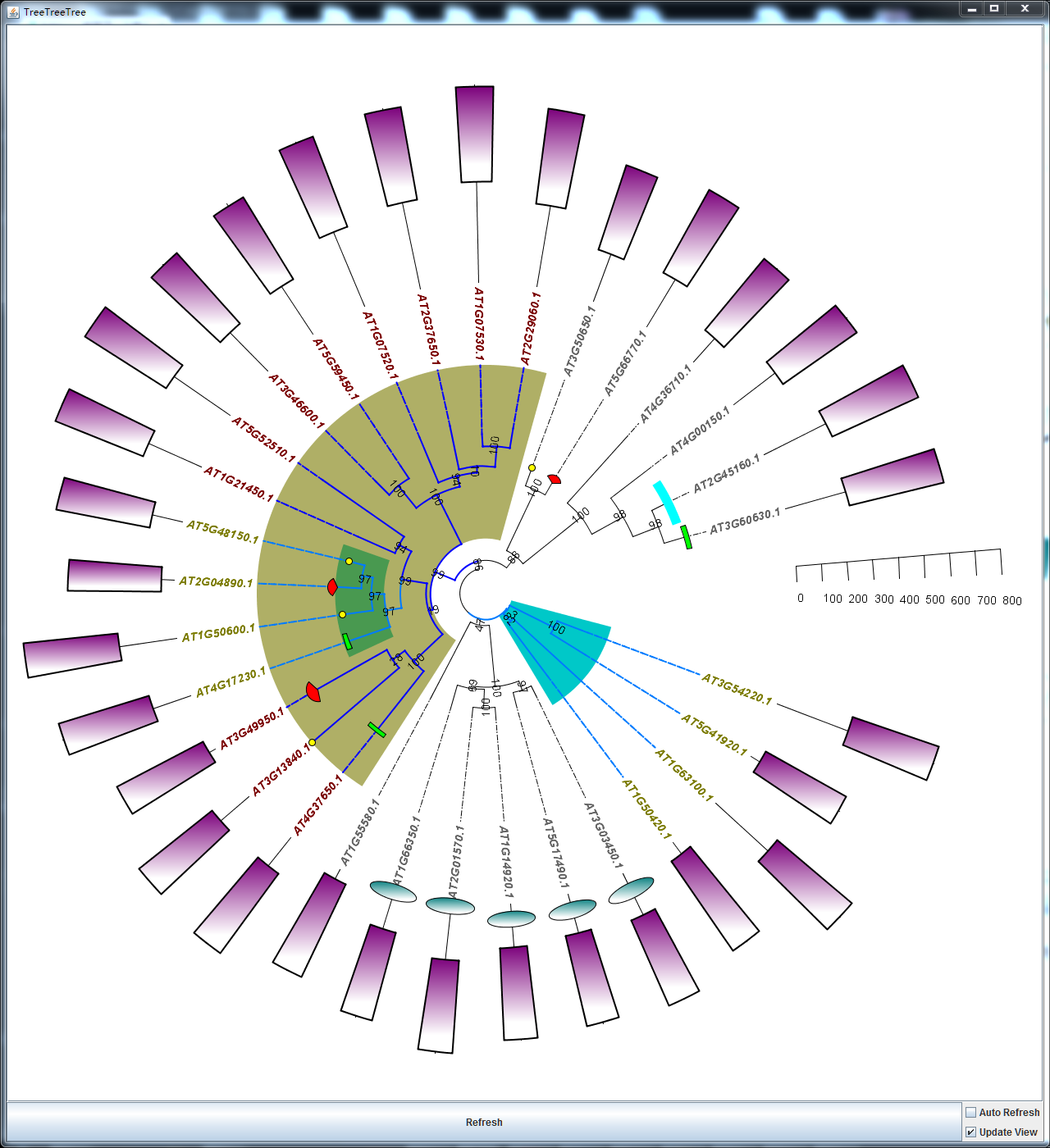
但是,如果你只是要画这么一个图片,为什么不用Amaing Optional Gene Viewer吗?如果是热图,那么为什么不用Amazing Heatmap呢?
或许,一旦你有了文件,你就一定能重复这个图。或者一旦你需要修改什么,那么你就只需要对文件进行细微的修改,就可以得到更好看的图,这就是原因。简单来说,你有配置文件,你就有图。
图就是文本,文本就是图
写在后面
我没什么想说的了,估计又要基本停更了,因为我已经开始赶下一个课题了。
今天是教师节,那就祝大家教师节快乐吧!
顺便中秋节也快乐!

