到底还是写了一篇推文,却着实想不到一个合适的题目。可能这是我写过最长的一篇推文,约莫万字。这篇推文的主要内容应该包括:
- TBtools 开发为了什么?
- TBtools 是怎么开发出来的?
- TBtools 版本迭代历史?
- JIGplot <- JJplot2 <- JJplot 的迭代过程?
- 我是如何接触生信的?
- 本硕博均是园艺果树专业,如何一步一步自学并入门生信。
- 一份生信编程入门书籍清单 和 社群与论坛推荐。
- 一些不堪回首的往事。
- 感谢陪伴我和帮助过我的人。
PS:推文在 TBtools 文稿 Online 前后,已经基本成型。原定在期刊官方公号推文后再行推出,但一周以来,身边和用户朋友一再催促。也罢,推文也是我硕博生活至今小结,就此推出,用互联网帮我记住这些经历。
写在前面
2020年06月22日,TBtools 终于还是发表在正式期刊(Molecular Plant)上。
这不仅仅是我一个人的开心,而是一小群人的快乐。这群人,或者对 TBtools 的开发极度支持,或者看着 TBtools 一步一步,一步又一步成长成现在模样。五年有余,说长不长,可以覆盖了一部分用户的硕士博士阶段。说短,其实也不短。软件开发不同于湿实验开展,前者周期往往足够短,甚至两三个月可以完成和测试。文稿的撰写到发表,从某个角度来说,是在用户朋友和生信小圈子一些朋友的催促下完成。总的来说,这应也算一个工作的阶段性成果,所以值得记录一下。华南农大园艺学院果树系,本科 —> 硕士 —> 博士,Emmm,是我常常不务正业的十年。而我折腾的与生信擦边的各项,正好也是TBtools 的开发过程。以下,用相对碎片化地方式,整理一下这些那些我还记得的片段。
遇到了第一个生信坑
我觉得我这些图表,其实还做得不错。这已经是2016年的事情了。
合伙买了二手服务器
折腾数据,必然是需要计算资源。正好那会浩哥(即 TBtools 论文二作)也计划开始搞分析,于是我们二人合伙从淘宝搞了一个二手服务器。如果我没记错,加上我们自己买的民用硬盘和其他,大概也就五六千。配置是相对奇葩的状态,似乎是1T还是3T,内存排满72G,线程16个。那会这个配置其实可以满足转录组数据分析的需求了,所以我们用得还是开心。
服务器的费用是两人各出一半,即每人3k左右。那会园艺学院硕士补助,多半是100~200。所以,或许很多人不知道 ,我出的这一半其实是我硕士三年的补助,或者说饭钱(估计浩哥也不知道 )。以至于,后来有那么大半个月,我其实是靠着我女朋友的饭卡吃饭的(没有更好的方式)。
学习了 Perl 语言
解读公司的结题报告,是一件麻烦的事情。2014年那会,其实做科研服务的公司很多,但都不太成熟 。公司返回的结果,往往是一些大文件或者大表格。整理起来比较麻烦。于是,多少学习编程语言成为必须。那会没有太多纠结,在浩哥的推荐下,我学起了perl。如果我没有记错,有那么一段时间,可能是个把月,我就只呆在宿舍,常常门也不出,等着其他人给我带饭。我只是刷编程书(其实后来我还看了不少perl相关电子书,不过大骆驼我是买的纸质书),试试运行代码,反反复复。用perl写了不少脚本,做了不少有趣的事情。我甚至只用 perl CGI 架了一个小网站,提供序列批量获取和BLAST的功能(命名为xxxblast,对标 wwwblast;我没有用框架,因为确实也不够机智)。
我印象深刻的,或许还是写出来的第一个我自认为有用的脚本。那个脚本的功能非常简单,批量提取Fasta序列。说起来还是有点搞笑(公众号也曾提及),那天晚上我写到凌晨三点,终于认为脚本稳定且正确,具体逻辑是:
- 用一个脚本把原始的Fasta序列整理成一行ID一行序列的格式
- 使用另一个脚本读取一个ID列表,然后遍历1. 中格式化好的序列,匹配的输出
这个功能的低能实现,让我兴奋了好一会。
现在想想还是有趣,因为 Perl脚 本我早已不写,而 Perl 只活在我的命令行里面。批量提起Fasta序列?我早已能够随手写一行 perl 命令搞定。不仅仅是批量提取序列,甚至包括更为复杂的分析。
Emmm,回想起来,我甚至已经忘了,到底那本 Perl入门经典小骆驼,是浩哥的,还是我的。多半应是他的,而我忘了也没打算还他了。
学习 Linux
那是一个寒假,我第一次没有回家里过年。借了浩哥的一本《鸟哥的Linux私房菜》,硬是啃了一个星期(还是两个星期?)。啃完就折腾服务器,边折腾边重新啃,来来回回,应是翻了不下三遍,书中也被我用铅笔写满的标注。也因此,我自认为现在的 Linux 基础起码比不少自称搞生信的要扎实一点。
当然,这还得益于另外一些 Linux 操作相关书籍的阅读。Linux的学习,似乎主要还是对文件系统的了解。其次才是一些shell下的 工具或命令。由于我先学了 perl,所以更多工作我会写 perl 单行来完成,而不会用 awk 或者 sed 等。说得自大一点,我认识的人里面,没有一个 perl 单行写得比我更好。一是我写得早,二是我写得久,三是我天天写。
学习 R 语言
Perl 学了,Linux 也基本掌握了,那么生信数据分析的文本处理,已经基本得到解决。可视化成为当务之急。事实上,那会我和小庭子有大体的分工,我搞Perl,他学R语言,后面合伙就可以更快地做点事情。当然,后来各自课题和生活上事情较多,我还是自己学了一下。提起R语言入门,我从来推荐《R语言初学者指南》。
Emmm,那么到底这本书是我的还是浩哥的,我还是忘记了。不过,可以认为这本书确实是简单。看完了,再看看 《ggplot2:数据分析与图形艺术》,那么似乎真的没什么统计图表是不能在短时间内完成的。后来我发现,R语言真正方便的应该是统计,而可视化是附加的事情。只能说生态赋予了R在统计可视化上的完美优势。
开发了密码子偏好性分析流程 R2CUB
很少人知道我做过密码子偏好性分析。因为我的课题中并没有这一部分,而那会只是一时兴起。应是研二研三了,我估摸着花了个把星期的时间,开发了一个流程。这个流程在数据分析上是纯粹的 Perl 语言 Naive 实现,所以并没有任何其他依赖,只需要电脑上安装基础版本的 Perl 语言解释器即可。文档,手册等等,我也整理完善,甚至喊了其他朋友测试鲁棒性。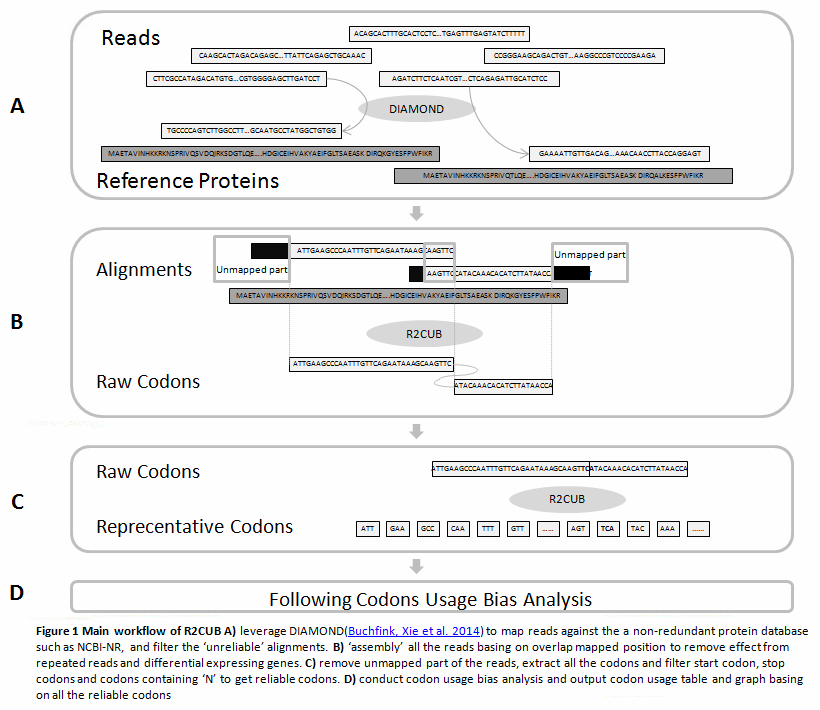
是的,这是那会我把文章都写了,最后似乎没有投稿。DIAMOND,那会才刚刚出来,网络上没看到多少应用(如果说看到广泛使用的,那至少也一年后了)。可能也是我用得早,所以对他当时版本的局限有一定的了解。后面我再也没有关注,主要还是用回 BLAST。分析结果出来的,RSCU 和 ENI 等数值计算出来了,那么可视化就用 R 脚本。
可以看到具体到科属还是分得比较好的,单纯只用RSCU做了一个聚类。其实这个流程还是不错的。我曾经在园艺学院生物技术所(现在似乎已经改成其他名字了)组会上,开始分享进展时,回车一键化流程,分享结束了,也就可以看结果(其实跑起来一个物种也就几分钟)。当然,最后我并没有从中看出有用的生物学意义 ,所以流程和文稿都打入冷宫,不再启用。鼓捣了这个流程,我便意识到一个问题,即常规流程,命令行运行,实在太麻烦。
学习界面化软件开发
这里涉及到一个点,为什么我一定要搞界面化软件。前述已经提及,硕士生活补助全部投到了服务器(换句话说,其实已经不够钱吃饭),所以在一段时间内,我做过一些现在看起来有点坑的业务,比如代写 Perl/R 脚本,五块或者十块钱一个,接了不少。而后来,接转录组测序数据分析业务,才发现其实搞分析原来没那么廉价。当然,做这些事情,是其他更为艰难的生存无奈所迫,具体便不做开展。回到主题,了解我的人,知道我做买卖有两个原则:
- 只卖我觉得好的东西
- 只卖给有需要的人
既然接了一些分析业务,常常就会遇到客户问题(当然,自己分析结果自己课题组使用起来,也一样)。其中最经典的就是Fasta序列提取。现在回想起来,我仍然想不明白,那会做生信擦边课题的人真的很多,而搞科研服务的公司也有不少,为什么就没有人做一个界面友好,功能实用的小软件。仅仅是从转录组组装结果中批量提取Fasta序列(如某几个差异表达基因的序列),就必须要求客户安装 Perl 解释器,然后用 perl 脚本….
于是,我还是决定下来写界面化工具。我没有记错的话,那会我买了一本Java 入门书籍和一本 C++ 入门经典教材。
翻了一天多Java,用notepad++,写了简单的界面化软件,大体是一个sci-hub链接自动获取的功能(Emmm,是的,没有用IDE,这主要是我之前写perl的习惯没有调整过来;sci-hub在国内似乎才刚刚兴起,并不像现在这样到处是文献获取神器)。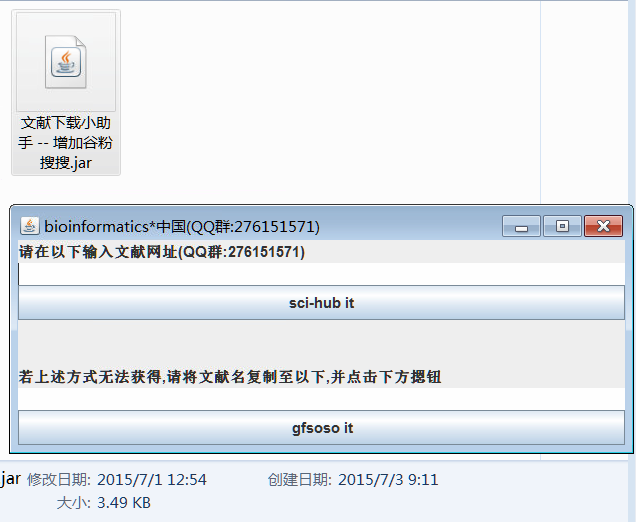
总体上,感觉写Java代码的效率明显不如perl。于是,我又翻了两天C++。
写了几个命令行小工具,却没有搞明白如何更好地打界面,尤其是打造跨平台的界面化软件(注意,我们永远无法确定用户到底是Windows,MacOS,还是Linux)。在这样的背景下,我又重新转向Java,然后写了一个简单的序列提取工具,随后释放到网络上。
那会可以释放的地方极少,大体上就是:
- bioinformatics*中国 QQ交流群
- PLOB
没有了。整体上,其实还得到不少有趣的评价。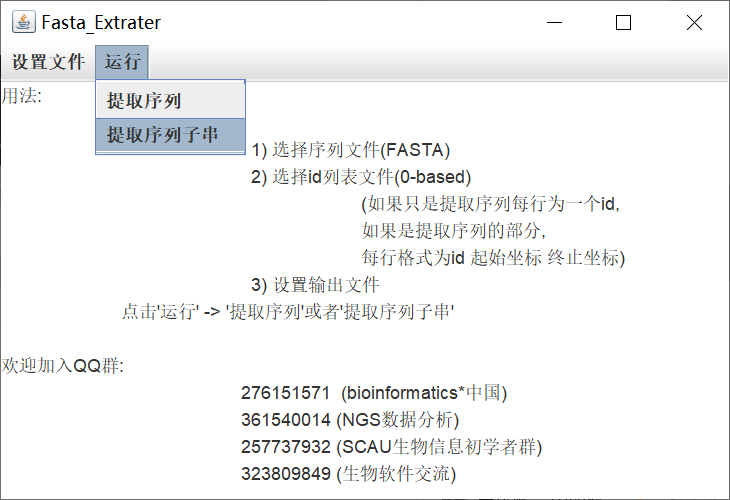
界面非常简单,Jar包是22Kb。不小也不大。
开发一点相对有用的工具
掌握了基本的界面化软件开发,我想到的第一件事就是如何更好地分发已有的教程或者简便操作。在这段时间,我操作系统转向ubuntu(Linux的一个发行版,这似乎是我第一次尝试在工作中不使用windows)。用Java写了一个感觉比较有趣的东西,大体就是可以方便地将任何命令行转换成界面工具。比如,只要编写一个配置文件,那么就可以把 BLAST 命令转换成界面化工具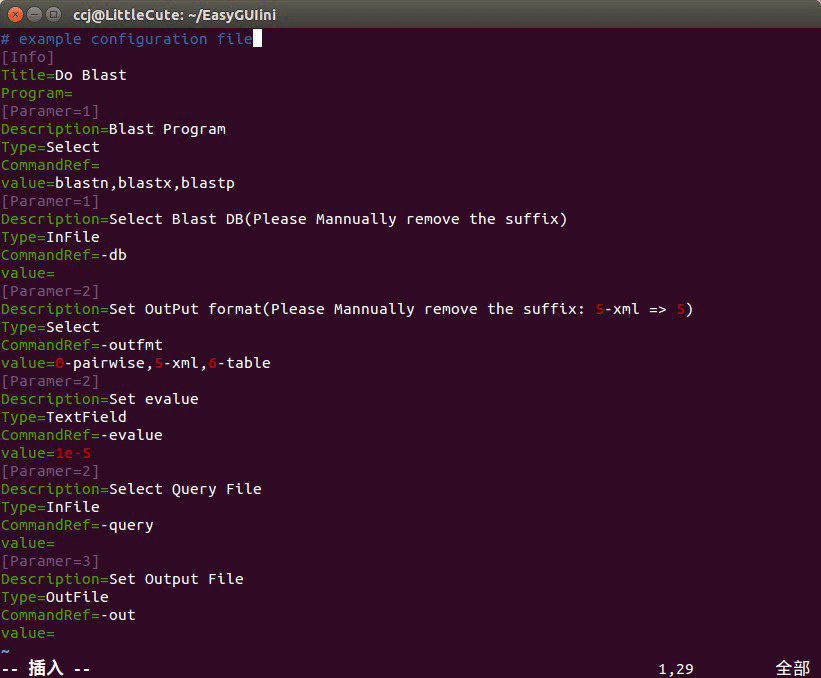
自动转换界面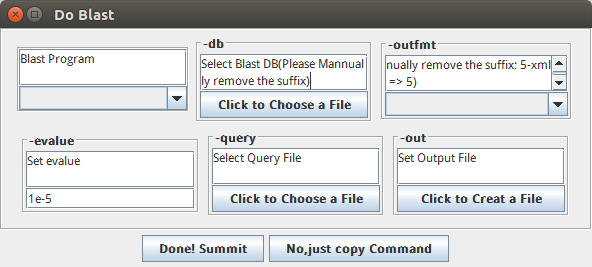
事实上,这个小工具可以认为是简单的插件化软件开发,或者是一个通用的界面化工具,比如我们只需要编写简单的配置文件,就可以把perl单行,一些命令行调用的工具,或者是 perl 和 R 画图脚本转换成界面工具。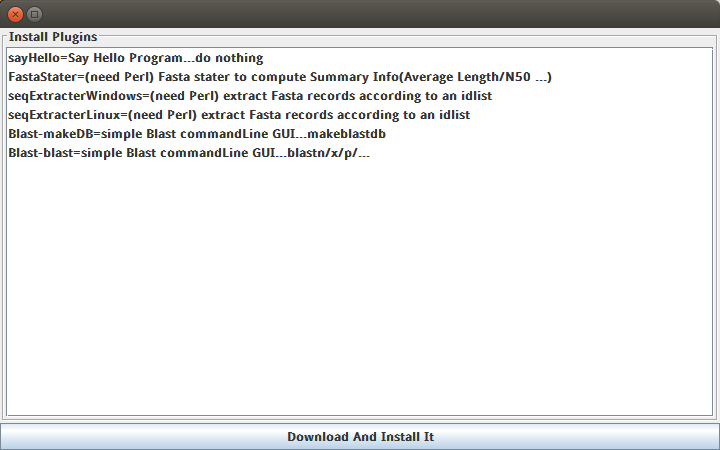
是的,我甚至还在一个服务器上部署了插件中心….
图中一语成谶,后来,我硕士确实延期了一年,现在博士也延期了【微笑脸】^_^。
后来,就没有后来了。EasyGUI 的这个工具无法很好地自动布局界面。另外实用性其实不太高,毕竟他只是一个纯粹的自动界面化接口。
在这之前,有传言 blast2go(一个强大且通用的GO注释软件)要开始收费,或者说不再提供免费版本。我大体看了下 blast2go 的文章以及他所使用的数据库,基本了解了主要逻辑后,自己写了一个 perl 版本的 idmapping,功能即将NR等蛋白序列库注释转换为GO注释。事实上,用起来还是不错。看着bioinformatics群不时讨论到GO注释相关问题,一时兴起,我又写了一个 Java 界面化工具,并直接命名为 blast3go。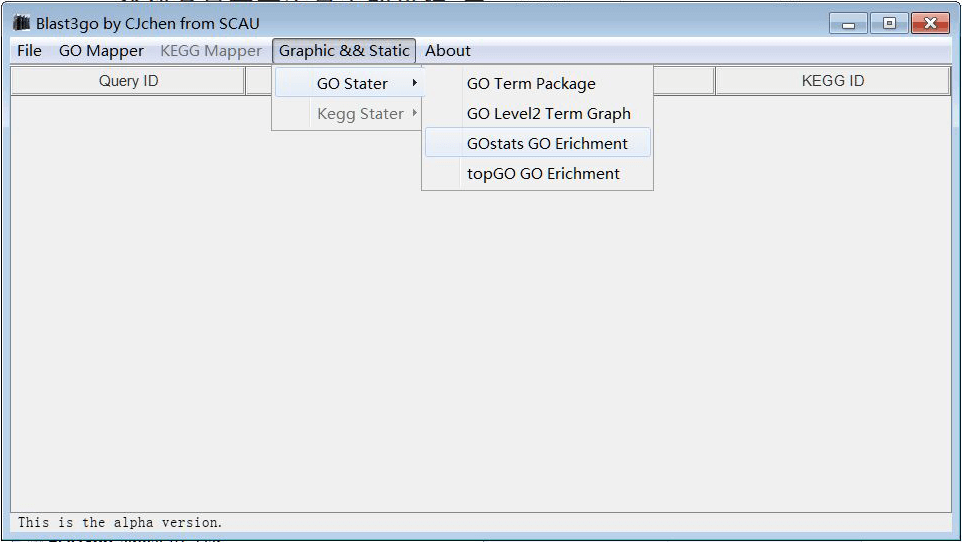
这个工具,还是比较有趣,GO注释逻辑是自己实现。而富集分析以及可视化,使用的是 R 语言包,包括 GOstats 和 topGO (那会我还没认识大湿兄,也不知道 ClusterProfiler)。效果图如下,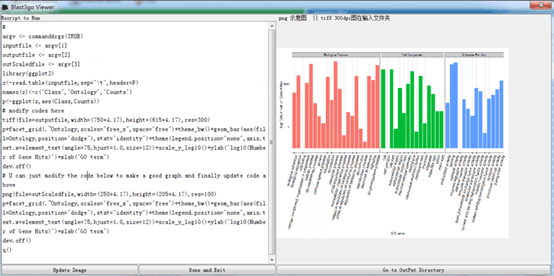
是的,打包的就是我自己写好的脚本….我尝试了几乎所有可用的 java 和 R 语言交互接口,最后发现效果都不如直接调用 Rscript。这段经历,事实上也让我彻底对*打包 R 甚至使用 R 语言失去偏好。
认识大佬,初识生信
上述,我提及数次,bioinformatics*中国 这一 QQ 社群。在那里,我认识了一群人,真正意识到生物信息其实不是我认知的样子。他们之中,有专门做算法开发的,软件设计的,流程部署的,科研应用的。这些人,改变了我对生信的认知,甚至是让我真正明白,什么是生信入门。由于我个人相对积极地参与群里的交流讨论,后来做了群管,并担任了一届群主(后来由于 TBtools 社群管理需要,届满我就退群了)。期间,我们在群里举办了两三年每周 seminar,一次20期(可惜那会没有做充分的录屏)。四五年前的事情,我自认为那至少是国内最早开展的生信交流网络直播。后面过了一些时日,才看到各类公司或者社群搞起类似的事情。总的来说,我从其中学习了很多。可惜的是,这个社群那会早已满2000人,所以对入群感兴趣的,可能早就没有机会(因为,我自己也回不去了)。由于我们只想做精不想做大,所以清理人出群是我们的日常工作。这一习惯,现在沿袭到所有 TBtools使用交流群。
值得提的是,在这期间,我有幸在 Zinky (赵齐,现任职于中肿)的带领下,蹭了中山大学任间教授课题组的两三次组会。我也因此明白,其实生信软件开发,有非常多值得做的事情,而 Java 这门语言,似乎存在很大希望(虽然,五年后的今天,传言他们已经几乎不再使用 Java 做界面化软件开发了,可能 python+Qt?或者专注网页工具)。
偶然合作,共同开发 EasyCodeML
如果让我一定要推荐质量高的生信交流QQ社群的话,那么有且只有两个:
- bioinformatics*中国
- 生物软件交流群
后者应是福建农林大学高芳銮老师(Raindy)组建并后续由几位大佬一起维护的QQ群。前者重点在大数据,后者重点在群体演化以及传统的生信数据分析。在一次偶然的讨论中,Raindy 提到正选择位点分析,是常见分析,但很多做进化分析的朋友,却常常搞不清楚如何操作,单纯正确的分析逻辑,可能需要数周甚至上月入门。降低门槛,让这类分析不再困难,于是他提出了 CodeML 分析的界面化可能。那会我正好没有手上没有更多值得开发的项目,也就一拍即合。当然,后来的事情就显得复杂。因为我并没有进化分析的背景,所以我们经过了艰难的沟通,才勉强完成了第一个使用版本(图示为最终版本,图片来源:http://wap.sciencenet.cn/blog-460481-1163040.html?mobile=1)。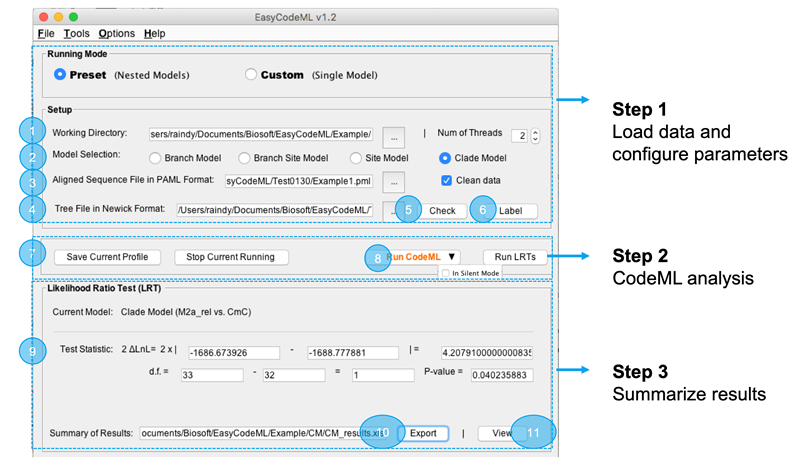
关于 EasyCodeML 的开发,我其实测试了不少方法。单纯在进化树可视化以及分支选择上,我直接使用了 JavaScript 的 D3.js 库,从零画树,并实现浏览器的交互。第一版 EasyCodeML,其实是 Java 同时打包一个 Chrome 的浏览器驱动,用户进行分支选取,其实是调用本地 Chrome,在网页浏览器搞定。随后才跳转回 Java 界面。可以注意到:
- 从交互的角度,R 语言出图自然是不行的,那也没什么合适的 R 语言进化树可视化包
- 从便利的角度来谈,调用本地网页浏览器,让人很难受 。
当然,这是最早期的版本….早已废弃。我们合作的最新版本,在2018年已经发表。这也是我第一篇共一以及共通讯的生信软件类文章。
着手开发自己的绘图引擎
在几乎相同的时间上,由于课题的需要,我也受够了 blast3go 的不实用性。开始了 BioCJava 项目,主要实现了序列批量提取和GO注释,以及GO富集分析的功能(这个时候 ,富集分析已经是我自己纯粹的Java实现,所以跟 R语言不再有交集)。
一方面EasyCodeML的进化树分支选取上需要更好地实现,另一方面 BioCJava 项目只能做纯粹的文本处理,实用性不高。似乎还是一个寒假,甚至又是过年在家的时候 ,我读了《The Grammer of Graphics》这本书,重新理解了图层语法。
那时的我,觉得图层语法实在太流弊。Java也应该有一个。于是我自己着手写了 JJplot (参考R语言ggplot2的名字)。如果我没记错话,我为此还画了一些UML,毕竟实现还是没那么简单。说是图层语法,其实实现下来还是naive。不过足够我用于完成 EasyCodeML 的进化树分支可视化选取 以及 GO Level 2 注释可视化。
TBtools 开发伊始
所有的无奈,全部的认知,让我在 TBtools 的实现,有更细致的思考。参考前述,一开始 TBtools 的名字是 BioCJava。但两三次被认为是 biojava 的界面打包后(不能怪别人看不到 CJ 二字,但实在不爽)我临时决定修改程序名字为 TBtools,意指 Tools for Biologists。后来在大群释放后 ,被传布成了 Tao Bao tools,意思是 淘宝工具。嗯,多少可以蹭点淘宝热度,其实也不错。误打误撞,我也没有再修改他的名字。
这是一个相对早期版本的 BioCJava,时间是 2015年08月31日(项目开始的一个月后)
注意到,这个时候 TBtools 的 Logo 已经存在。这个 Logo 还是有点故事,主要贡献的人是前述提到的女朋友,即借我饭卡,我白吃白喝了个把月的那个。也可以看到,BioCJava 只有三个基本功能,谁也不会想到,五年后的几天,TBtools 界面上就是 140+ 个功能(更不提背后的 360+ 个命令行功能)。那会用户其实非常少,应不到10个人,主要还是找我帮忙分析数据的老师或者课题组同学。
TBtools 开发第一年
在这个时间点上,我仍然还在大群(即 bioinformatics*中国)活跃。所以 TBtools 主要推广还是在大群。基于群里的交流讨论,我也不断改进和开发 TBtools。
这已经是开发约莫一年后的样子。主要只是拓展了GO注释,富集分析,以及GO层级统计的功能。为什么一年时间才写这些功能?因为我也有生物学实验和其他课题要开展,TBtools 的开发从始至终,可以说是一个上班摸鱼或业余消磨时间的方式 。当然,这一年的跨度,可能还是在 JJplot 的开发上。
TBtools 开发第二年
这一年,我硕士延期了。不过这个延期是我自己要求的。因为我刚刚解决了一些不得不解决的现实问题,可以开始更有尊严的活着。于是,我那会打算是争取出国(当然,最后没有出去有另外的原因,包括身体,家庭,去向以及offer来得太晚)。无论如何,这一年却也是 TBtools 成长飞快的一年。我暑期在家,想明白了一点事情,于是将 JJplot 全盘推倒,重新写了一个更具图层语法意义,支持全图缩放和交互的 JJplot2。于是才会有系列其它可视化内容,包括韦恩图以及共线性可视化等。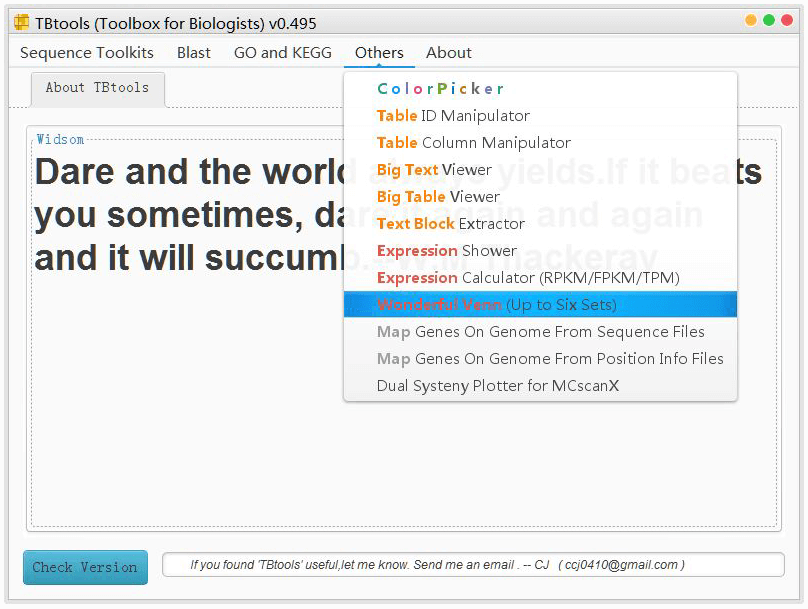
值得一提的是,Omicshare.com 论坛开设不久,我也就跑到论坛上做了一些宣传,最后还当了一个版块的版主(当然,现在还是)。现在,我没有继续主动在上面推广 TBtools,原因主要是论坛重点在于生信讨论,不太适合过多推广TBtools。无论如何,我觉得 Omicshare 论坛,仍然是我极力推荐的生信论坛(可能得到充分的公司资金支持,所以可以较好地运转)。感兴趣的可以点击这个链接 http://www.omicshare.com/user/register.php?uhash=1659401951,跳转并注册账号。 。对应的也有一些云平台免费或者收费的小工具可以使用。
。对应的也有一些云平台免费或者收费的小工具可以使用。

JIGplot 的到来
转眼就是2017年,我注册了并开始运营《生信札记》微信公众号(当然用心经营是一年后)。在上面撰写了一些生信学习经验,而后来便主要用于分享TBtools 使用实例。TBtools 社群在接下来的一年间快速增长,很快便满人。有了bioinformatics中国的管理和发展经验,我试图尝试另外的发展方式,即若有必要,就开设新群。但很快,两个2000人群的名额已经用完,大群任期也临近。我便换届卸任,将重心转移到接下来的博士课题和TBtools社群管理上。
或许2017年的整整一年,是我相对轻松地一年,那边硕士毕业没有问题,这边博士也定了去处。我重新思考了绘图引擎的实现。值得提及,这段时间,大湿兄(Y蜀黍,现任职于南方医科大,博导)在他的公号上不时更新了一些推送。我现在只有大体模糊的记忆,其中一句话,对我的触动相对较多,总结一下,*所有的计算机图形可视化,都可用 点 决定。两个点就是一条线,三个点就是一个图形。这本身只是一个初中几何知识,但我却用了两三年才想明白。于是,在接下来的半年到一年内,我又一次完全推翻了 JJplot2 实现,开发了 JIGplot。
这一次,我不再执着于图层语法的实现,当然思想上我依然有所参考。相应实现的思考,在一个PPT,我可能已经打开,思考,调整,关闭了不下百次(Emmm,我不小心都快被自己感动哭了)。所有的思考和努力,熔铸在 JIGplot。
从此,TBtools 的可视化相关功能得到绝对地拓展。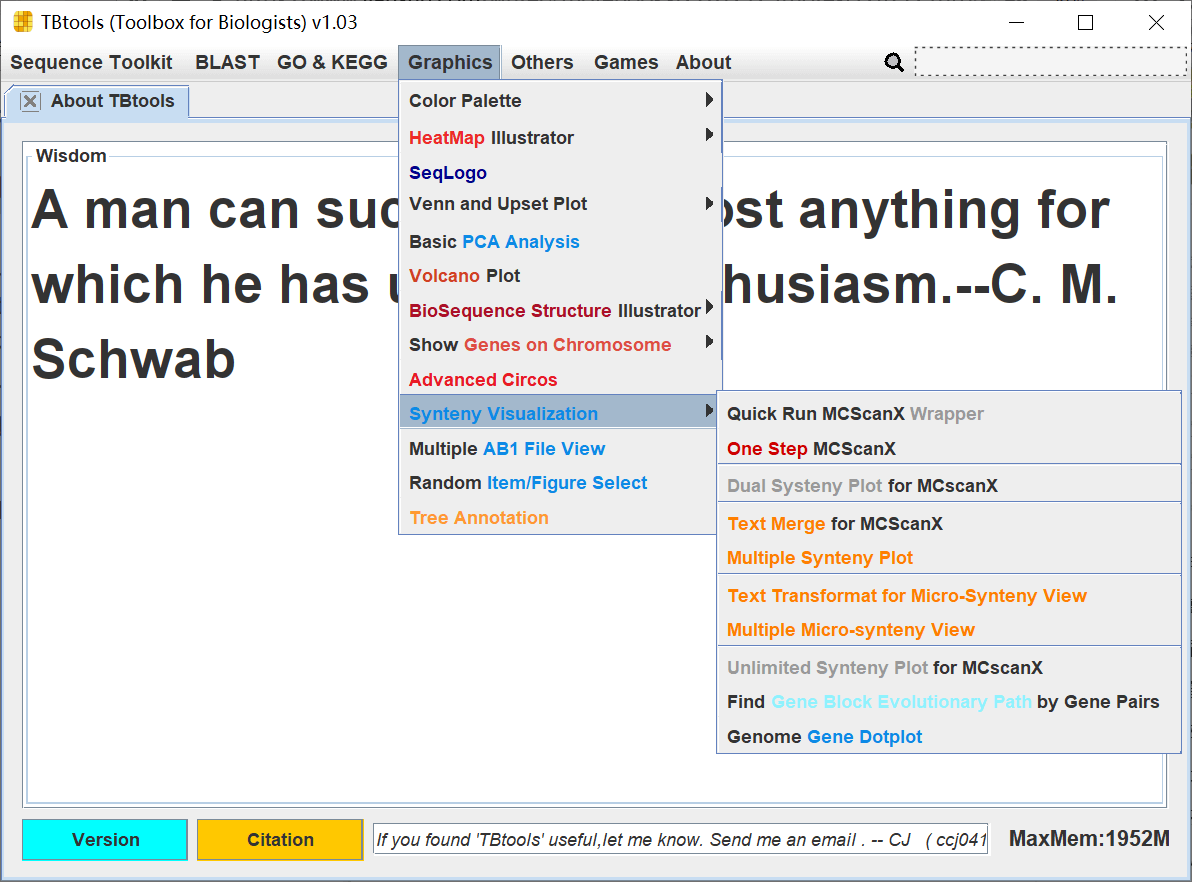
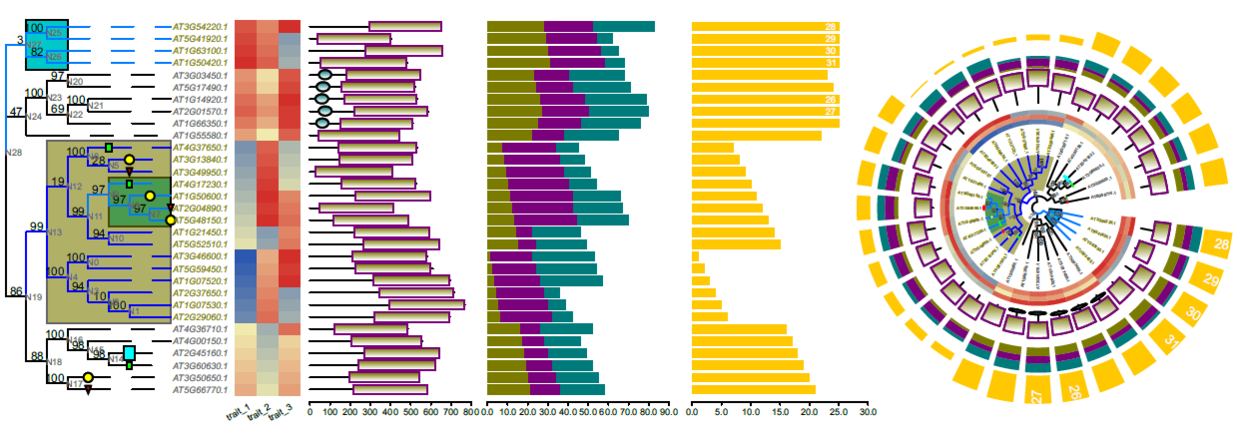
博士研究生阶段
时间飞逝,硕士一年的延期结束,我也开始了博士研究生生活。在这一期间,硕士课题组做了一部分菠萝基因家族分析的工作,同时博士课题组(我尚未毕业)也在做一些家族分析工作。我看着他们做一些结构域预测可视化,MEME motif可视化,甚至于基因结构可视化时,总是难受。因为效率真的太低,可能搞完了要两三天。既然有了JJplot2,我反手就是一个生物序列可视化功能,无论是结构域,MEME还是基因结构,直接继承一个类,于是变成三个独立可视化功能。Emmm,感觉这个功能一下子帮大家省了不少时间。
JJplot2 的实现,并不支持拼图。而 JIGplot 天生就是图层化。博士期间我进一步开发的 MetaGeneStructurePlot,支持一键化,一张图同时展示 ,进化树,MEME motif,基因结构,结构域,甚至其他。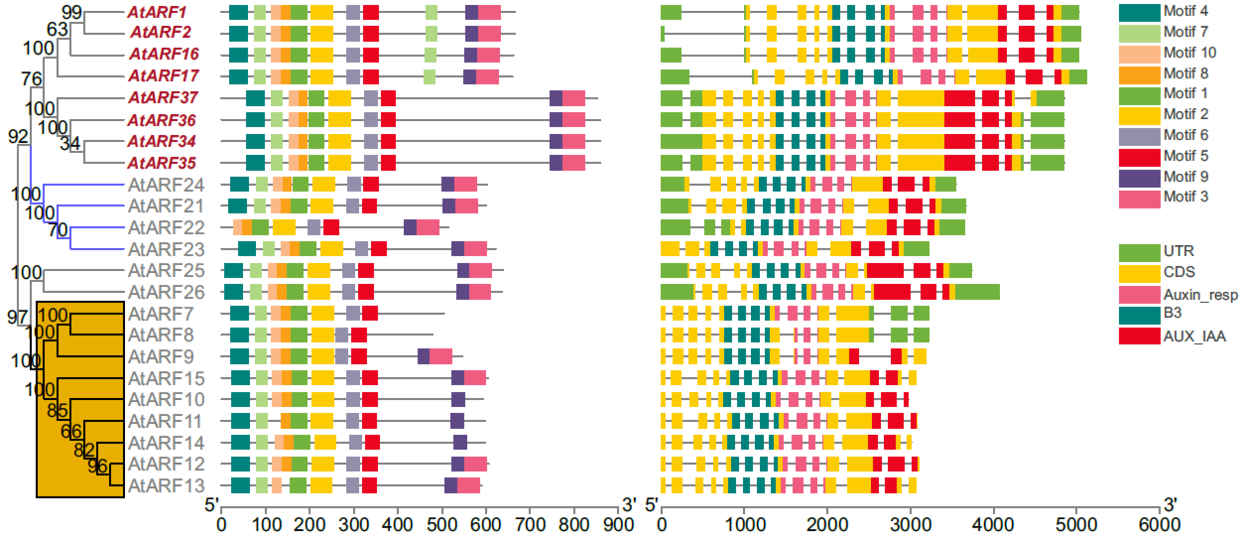
当然,可能也是这个功能的推广,TBtools一度(或者甚至一直)被人认为就是专门做基因家族分析的工具。对于这个,我也很无奈。
博士研究生这三四年,事情确实多了许多,课题也多了很多。TBtools 的开发主要是基于我个人或者课题组课题需要,所以功能也在不断丰富。其中包括一些比较基因组相关工具。当然,更多的其实并没有开放出来,所以可能知道的人不是很多。总的来说,虽然这两三年没有非常明显的阶段性突破(如还再推翻绘图引擎 JIGplot),但更多功能得到开发,优化,增强和拓展。如果说我硕士阶段对应了 TBtools 的出生和儿童时期,那么博士阶段对应的则是 TBtools 长大成人,并办了成人礼(TBtools文章终于有了归宿)。
TBtools 功能现状
TBtools 能够有如此丰富的功能,有赖于我所在课题组(博士-夏瑞教授课题组和硕士-何业华教授课题组),TBtools 三十多位群管 以及 目前 2万有余用户的意见和建议。相关的推送已经有很多,这里附上一张简图,
目前已开放的GUI功能140+,而这个数字,事实上还在增长。
TBtools 投稿经历
前述已基本理清 TBtools 项目开发始末。可以说,TBtools 从一开始就是我个人课题或者分析需求导向开发;说得直白一点,那么是顺手写一写。未曾想过他会走到今天,更没想过将其撰写成文并发表。
第一次撰写成文,已经是2018年年初(开发了约三年),在较多用户的催促下,草草整理了一份 PDF 文档。文档相对粗糙,我花了两三天就做完,就放到 bioRxiv,因为其他课题也在开展。
其中的图表是这样的
可以看到,基因结构的图片也只是朋友华南植物园王洁雨(演化生物学-公众号那个老王)画的,我截图拿来用了。图中还带着水印。很明显,那会的想法只是放上去,不要再被用户怼说链接不好引用。
就这样,过了半年,我们手上项目暂有停歇,另外多少我对论文发表有了新的认知,应是包括《园艺研究》主编程宗明老师的一次报告提及,既然工作做了,就还是要发表。我与夏老师商量后,决定还是花点时间,尽量投了。我们又花了几天,写了一篇感觉还不错的短文,Application Note,投到《Bioinformatics》。大体是这个状态(这些可以在 bioRxiv查看到)
其实这张图表也挺好看的。若是做一点生物信息的,多少还是有一种执念,即发一篇《Bioinformatics》( 二区 ),那时我也有(此前,已有一区刊物的朋友邀请投稿,我们婉拒了)。没有太多意外,Bioinformatics 编辑回信,大体是“我们稿件太多,懒得理你”。明显只是客套话,但也没办法(后来Bioinformatics分数 IF 一跌再跌,也就再也没想法更新并投过去了)。值得注意的是,投过去时,JIGplot已经开发并放进去了,我对 TBtools 的工作其实比较有信心,至少创新点是有的。但悲剧是没办法的。我与夏老师就这个事情讨论过一次,大体原因是,文稿内容和图片确实无法反应出 TBtools 的创新点和优势所在,换句话说,怪不了别人。此后不久,我们接到了另一个生信相关期刊(三区)邀请投稿,我们婉拒了;也接到国内国外其他期刊约稿,我们还是婉拒了。期间,不少朋友给了这些那些的建议,可惜,那时的我确实不在状态。
就这样,又过了一年。2019年年末,我家中有事离开学校,于是有了一段相对长的空余时间可以做点事情。夏老师和我前前后后讨论了数次,多半是“激烈讨论”(其实这个对我们两个是常态,但无论如何争吵,在课题或者学术的角度上,目标是一致的)。最终,我们努力打磨,加上一起参与到 TBtools 界面,文档,文稿工作的老张(张翼),Margaret 和 Hanna,终于还是得到一个不错结果,新的文稿基本可以展示 TBtools 的部分主要特性。

一共是三个图,这也是某种意义上的 TBtools 文稿完全重写的第三版。当然,更为幸运的是,分子植物期刊(Molecular Plant)的编委老师们愿意花时间了解 TBtools 相关工作,我们终于也得到了文稿送审的机会。在编辑和审稿人的建议下,我们再次完全重写了TBtools文稿(这应算作第四版),并补充了两张图。前前后后,我们几个人花了挺多时间和精力。最新的这版文稿,展示了 TBtools 更多的特性。静态图片终归是静态图片,无法完全展示,但我觉得这应该已经是极限。
经过了大半年的努力,文稿被接收发表。对于这四个 TBtools 文稿版本,感兴趣的可以直接在 bioRxiv 上下载并与 Mol Plant 期刊的文稿对比。
争了一口气
生活,或许如此。我们不需要过多理会别人的看法,而只坚持做自己觉得值得的事情。TBtools 开发早期,遇到过不少质疑,甚至是诋毁。甚至于一些人身攻击。五六年前的事情,也不值得详细提及。不过,看不上 TBtools 的,依然大有人在。这是一件理所当然的事情。从 TBtools 表面上能做的事情来说,确实并没有多少明显的超越。因为我一直看重的是降低甚至抹除数据分析的门槛,节省生物科研工作者的时间。所以 TBtools 主要开发目标一直是,将繁杂的分析过程简约化,常用的分析自动化。TBtools 里面几乎每一个功能都包含了我个人思考,即使是 BLAST 等界面化功能,我也专门做了序列类型的预判,从而节省了软件参数设置的时间。相应的优化还有很多,但是这些并不能被评判者们所看到。
有时候想想,你的汗水,你的努力结果,都在大多数人看不到的地方,那么并不能怪别人看不上你。只能说,可能你做的东西,本身就不容易被认可。当然,我运气比较好,一是 TBtools 用户朋友看得到,二是 博士导师和硕士导师的帮忙,三是 Molecular Plant 期刊的编委和审稿人愿意花时间了解 TBtools 的工作。三者结合,即天时地利人和,于是 TBtools 终于得以见刊。
当我知道文稿被接受的时候,多少我是松了一口气(甚至有早期用户与我提及他都要哭了)。或许这是因为我们已经自认优秀太久,终于还是争了一口气。我们打了那些看不上我们的人一记响亮的耳光,我们也不再需要承受无理的质疑:都没发文章,软件能用吗?预印本靠谱吗,不敢引用云云….
感谢
TBtools 的开发,完善,到现在阶段性的成果出来。事实上,需要感谢的人或者社群很多。以下我大体列举出来,但依然会存在遗漏,请注意到的朋友提醒下我。顺序按我脑海里浮现的顺序排列,不对应感谢的份量:
- 华南农业大学何业华教授,2009年本科入学,当年十月份,我便进入他的课题组,组培,分子,大田都接触了不少。硕士也顺利推免,此间一些我个人无奈但也不可抗拒的事情发生。何老师给与了我充分的自由,让我得以有足够的时间鼓捣数据分析,并最终找到解决办法。
- 湖南农业大学陈浩博士,跟着浩哥走,于是我有机会学习并了解生物信息数据分析。
- 贵州师范学院吴亚,前述提起多次的女朋友(现在是我的妻子),我们一起经历了生病却不够钱买药的阶段,一起租过半夜漏水的房子。每每想起,觉得委屈了她。她帮我设计了Logo,在早期界面设计和图形配色上给与了不少建议(比如MEME motif 可视化的配色方案)。
- bioinformatics*中国 QQ交流群的所有群管,如果没有他们,或许我从未明白生信分析到底是啥。千言万语,不如我们在群里开车的默契。此处不做展开。

- 中山大学的任间教授以及我齐哥(Zinky),他们让我看到 Java 开发可视化工具的无限可能。或许没有他们,那么我在 Java 学习上,可能一直无法入门。
- 南方医科大学余光创教授(我大湿兄,你们Y蜀黍),他不停地在博客或者公众号上分享的计算机绘图逻辑,触发了我的思考,从而才有 JIGplot 的充分实现。
- 华南农业大学夏瑞教授,TBtools 的完善得益于博士课题以及课题组其他成员的需求,建议和意见。TBtools 文稿构思,写作,投稿甚至于最终发表,我们一起耗费了大量时间和精力。前前后后 ,一个软件,鼓捣了两年文稿,全部推倒重写了三版。同时还要感谢张翼,Margaret 和 Hanna 在其中的时间和精力付出。
- TBtools 三十多位群管,当 TBtools 在交流群里,在其他社群里,在公众号上被怼的时候,是他们一直鼓励我,相信我,并支持我,于是我得以坚持下去。

- OmicShare论坛及其各个版主,虽然我退群有两三年,不过OmicShare活跃的那段日子,同样也是回忆,也是 TBtools 成长的一个阶段。我仍然相信,OmicShare论坛可以也值得一直办下去。必要的商业化,是保证公益可持续不可或缺的部分。
- 早期曾经捐助过 TBtools 开发的用户朋友,我印象中至少有三个人,现在应该都在高校工作了吧,他们捐助了加和起来估计有 500 块钱,那会正是我经济窘迫,而 TBtools 开发不被认可,孤助无援的时候。他们让我觉得这些工作,其实还是值得。我认为,一定的物质激励,或许恰恰是成事的关键。这里还需要感谢南京大学的一个老师,我们来来回回聊了几次,他似乎习惯把我拉黑,以至于我确实没搞清楚,他到底是姓汤还是姓周?
- SCAU生物信息交流群,这是我和浩哥一起组建的华南农大生物信息交流群。其中成员仍然不多。浩哥和我开始做生信的时候,我们自认为校内并没有做生信的老师。六年后的今天则有所不同。五年大招人和人才引进,华南农现在应是有一些侧重生信数据分析相关研究的课题组。当然,这并不影响我们组建的这个社群存在的意义,虽然他一直没有太多人。但, TBtools 的开发,尤其是菜单的重构,我有印象是在群里讨论中有了决定。
- 支持并帮助推广 TBtools 的系列公众号,这里不便一一列举,但我认为你们应该知晓。
- 参与 TBtools使用 和 基因家族分析 讲演的朋友们,你们的资助使得我有足够的资金购置安装器授权,使得所有用户尤其是新手用户,有更好的 TBtools 安装和使用体验。
- TBtools 的 2万+ 用户。TBtools 的开发和完善,得益于社群的使用意见和反馈。如果没有用户的意见或建议,TBtools 不可能有现在的状态。
写在最后
洋洋洒洒,不小心就码了一个长文。互联网,是有记忆的。多年以后,我回头来看,这篇推文,或许会更为有趣。感谢冥冥中,你着实让我相信,一切搞不死你的,会让你变得更强。
最后,还是用我的 QQ 签名作为结束语吧。


