一、降维
1D,2D和3D数据可以可视化。但是数据科学领域并不总是能够处理一个小于或等于3D得数据集,我们肯定会遇到使用高维数据得情况。对于数据科学专业人员来说,有必要对工作数据可视化和深入了解,以便更好地完成工作,我们可以使用降维技术。
降维技术得另一个最受欢迎的用例是在训练ML模型时降低计算复杂度。通过使用降维技术,数据集的大小已经缩小,与此同时,有关原始数据的信息也已经应用于低维数据。因此,机器学习算法已从输入数据集中学习既简单又省时间。
PCA-主成分分析是降维领域最主要的算法。它最初是由皮尔逊(Pearson)在1901年开发的,许多人对此做了即兴创作。即使PCA是一种广泛使用的技术,但它的主要缺点是无法维护数据集的内部结构。为了解决这个问题,t-SNE出现了。
二、t-SNE工作原理
首先,它将通过选择一个随机数据点并计算与其他数据点的欧几里得距离来创建概率分布。从所选数据点附近的数据点将获得更多的相似度值,而距离与所选数据点较远的数据点将获得较少的相似度值。使用相似度值,它将为每个数据点创建相似度矩阵
。
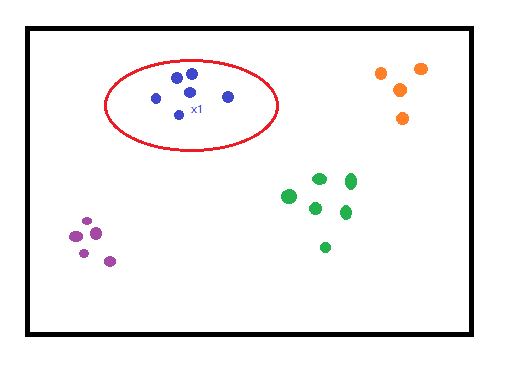
图1. 根据距离创建相似度矩阵
因为不可能将超过3维的数据集可视化,所以为了举例的目的,我们假设上面的图是多维数据的可视化表示。
这里需要说明的是:相邻指的是与每个点最接近的点的集合。
由上图可知,我们可以说的邻域
,这意味着
和
是
的邻居。它将在相似度矩阵
中获得更高的价值。这是通过计算与其他数据点的欧几里得距离来计算的。
另一方面,像上图中的绿色点集离最远的点,在相似度矩阵
中将获得较低的值。
其次,它将根据正态分布将计算出的相似距离转换为联合概率。
通过以上的计算,t-SNE将所有数据点随机排列在所需的较低维度上。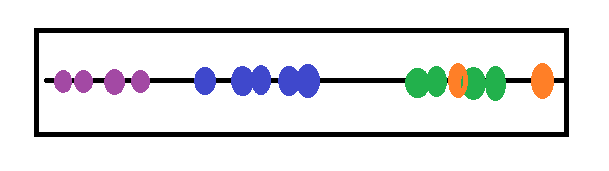
图2. 映射到较低维度上
t-SNE将再次对高维数据点和随机排列的低维数据点进行所有相同的运算。但是在这一步中,它根据t分布分配概率。这就是名称t-SNE的原因。t-SNE中使用t分布的目的是减少拥挤问题(后面与PCA对比可见)。
请记住,对于高维数据,该算法根据正态分布分配概率。
t分布->视觉上t分布看起来很像正态分布,但尾部通常更胖,这意味着数据的可变性更高。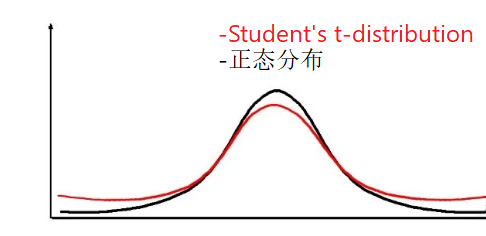
图3. t分布和正太分布
对于较低维的数据点,还将创建一个相似度矩阵。然后该算法将
与
进行比较,并通过处理一些复杂的数学运算来使
与
之间有所不同。包括使用两个分布之间的
(KL散度)作为损失函数运行梯度下降算法。使用KL散度通过将两个分布之间对于数据点位置的值最小化,帮助t-SNE保留数据的局部结构。
在统计学中,散度是对一个概率分布与另一个概率分布如何不同的度量。梯度下降算法是各种机器学习算法中用于最小化损失函数的一种优化算法。
最后,该算法能够得到与原始高维数据相对相似度较好的低维数据点。我们可以使用sklearn.manifold.TSNE()实现t-SNE算法。
要点:
- t-SNE算法具有扩展密集簇并缩小稀疏簇的特点
- t-SNE不会保留群集之间的距离
- t-SNE是一种不确定算法或随机算法,这就是为什么每次运行结果都会略有变化的原因
- 即使它不能在每次运行中保留方差,也可以使用超参数调整来保留每个类之间的距离
- 该算法涉及许多计算。因此,该算法需要大量空间和时间来计算
- 困惑度(perplexity)是控制数据点是否适合算法的主要参数,推荐范围是
- 困惑度应始终小于数据点的数量
- 低困惑度->关心本地结构,并关注最接近的数据点
- 高困惑度->关心全局结构
- t-SNE可以巧妙地处理异常值
三、简单的实现
我们使用kaggle的一个数据集,确定是有毒还是可食用蘑菇的分类问题(数据集下载地址)archive.zip ```matlab import pandas as pd import numpy as np import matplotlib.pyplot as plt import seaborn as sns
df= pd.read_csv(‘D:\谷歌下载\mushrooms.csv’) df.head()
```matlabX = df.drop('class', axis=1)y = df['class']y = y.map({'p': 'Posionous', 'e': 'Edible'})cat_cols= X.select_dtypes(include='object').columns.tolist()for col in cat_cols:print(f" col name : {col}, N Unique : {X[col].nunique()}")
col name : cap-shape, N Unique : 6 col name : cap-surface, N Unique : 4 col name : cap-color, N Unique : 10
col name : bruises, N Unique : 2
col name : odor, N Unique : 9
col name : gill-attachment, N Unique : 2
col name : gill-spacing, N Unique : 2
col name : gill-size, N Unique : 2
col name : gill-color, N Unique : 12
col name : stalk-shape, N Unique : 2
col name : stalk-root, N Unique : 5
col name : stalk-surface-above-ring, N Unique : 4
col name : stalk-surface-below-ring, N Unique : 4
col name : stalk-color-above-ring, N Unique : 9
col name : stalk-color-below-ring, N Unique : 9
col name : veil-type, N Unique : 1
col name : veil-color, N Unique : 4
col name : ring-number, N Unique : 3
col name : ring-type, N Unique : 5
col name : spore-print-color, N Unique : 9
col name : population, N Unique : 6
col name : habitat, N Unique : 7
for col in cat_cols:X[col] = X[col].astype("category")X[col]=X[col].cat.codesX.head()
3.1 使用PCA降维可视化
from sklearn.preprocessing import StandardScalerfrom sklearn.decomposition import PCAX_std = StandardScaler().fit_transform(X)X_pca = PCA(n_components=2).fit_transform(X_std)X_pca = np.vstack((X_pca.T, y)).Tdf_pca = pd.DataFrame(X_pca, columns=['1st_Component','2nd_Component', 'class'])df_pca.head()
画图
plt.figure(figsize=(8, 8))sns.scatterplot(data=df_pca, hue='class', x='1st_Component', y='2nd_Component')plt.show()
图3. PCA降维数据集
3.2 t-SNE降维可视化
import pandas as pdimport numpy as npimport matplotlib.pyplot as pltimport seaborn as snsfrom sklearn.preprocessing import StandardScalerfrom sklearn.manifold import TSNEdf= pd.read_csv('D:\\谷歌下载\\mushrooms.csv')df.head()X = df.drop('class', axis=1)y = df['class']y = y.map({'p': 'Posionous', 'e': 'Edible'})cat_cols= X.select_dtypes(include='object').columns.tolist()for col in cat_cols:print(f" col name : {col}, N Unique : {X[col].nunique()}")for col in cat_cols:X[col] = X[col].astype("category")X[col]=X[col].cat.codesX.head()X_std = StandardScaler().fit_transform(X)tsne = TSNE(n_components=2)X_tsne = tsne.fit_transform(X_std)X_tsne_data = np.vstack((X_tsne.T, y)).Tdf_tsne = pd.DataFrame(X_tsne_data, columns=['Dim1', 'Dim2', 'class'])df_tsne.head()plt.figure(figsize=(8, 8))sns.scatterplot(data=df_tsne, hue='class', x='Dim1', y='Dim2')plt.show()
图4. t-SNE分类结果
3.3 PCA VS t-SNE
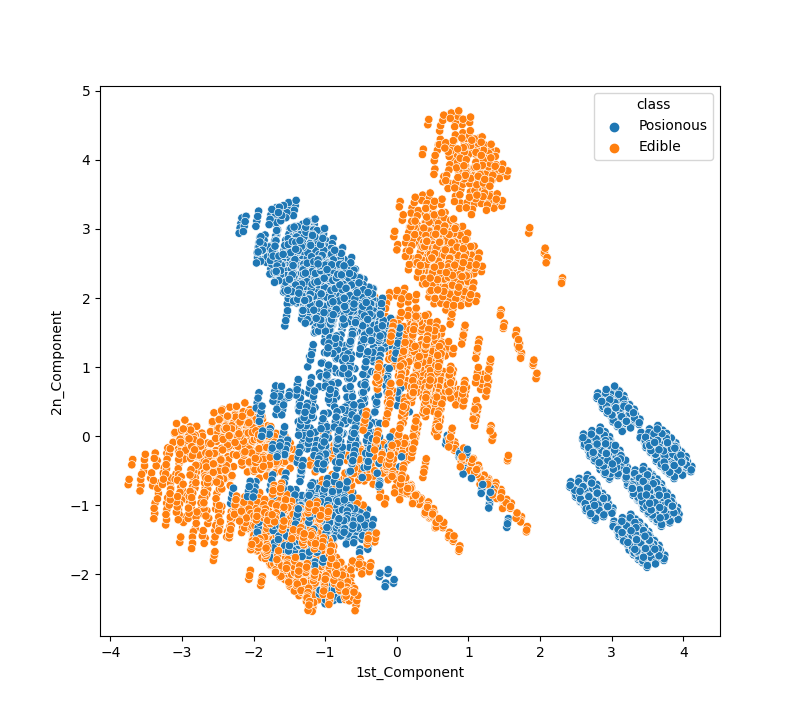
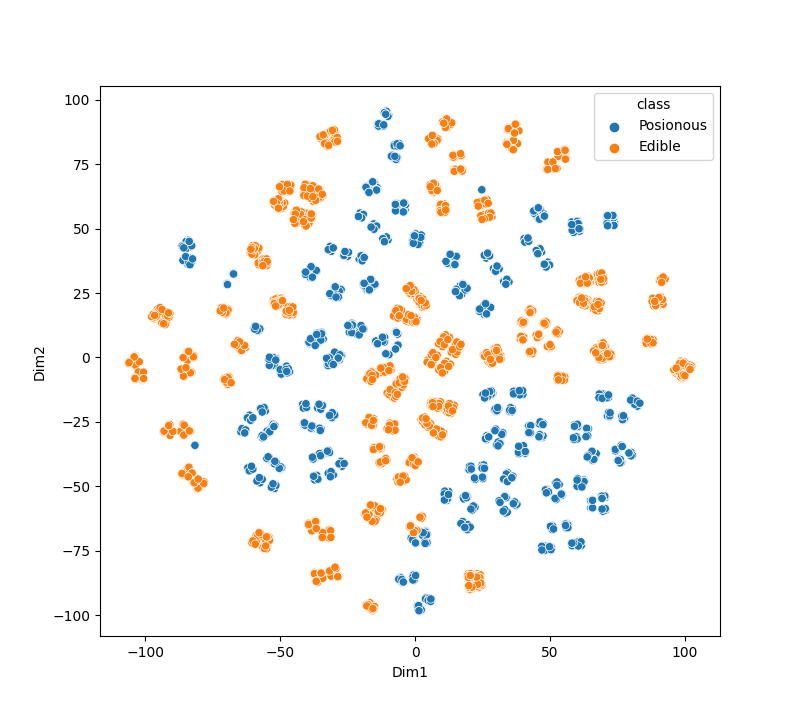
图6. PCA V.S. t-SNE
根据以上结果,可以说与PCA的性能相比,t-SNE的表现要好。
t-SNE算法将有毒和可使用蘑菇聚类,没有任何重叠;PC无法将蘑菇完美分类。
要知道在哪里可以得到稳定的形状,我们应该使用困惑度和n_iter参数,在困惑度=30且n_iter=5000之后,聚簇形状变得稳定。
四、总结
与PCA不同,t-SNE可以更好地应用于线性和非线性良好聚类的数据集,并产生更有意义的聚类。尽管t-SNE在可视化分离良好的群集方面非常出色,但在大多数情况下它无法保留数据的整体几何形状。

