**前言:**秩和检验(Rank sum tes)的定义和应用范围这里不多复述,网上有各种资源,需要注意的是两点:①秩和检验是一种**非参数检验法**, 它是一种用样本秩来代替样本值的检验法;②秩和检验**可以用于样本容量不相等**的两个或多个样本。<br /> **本文介绍如何在R上进行Kruskal–Wallis秩和检验。**<br /> 参考文献:<br /> [基于R软件的多组独立样本秩和检验的多重比较_金英良.pdf](https://www.yuque.com/attachments/yuque/0/2019/pdf/119869/1551270418162-8c76a1e3-b89b-4856-9dbb-91c47bb46722.pdf?_lake_card=%7B%22uid%22%3A%22rc-upload-1551270384124-3%22%2C%22src%22%3A%22https%3A%2F%2Fwww.yuque.com%2Fattachments%2Fyuque%2F0%2F2019%2Fpdf%2F119869%2F1551270418162-8c76a1e3-b89b-4856-9dbb-91c47bb46722.pdf%22%2C%22name%22%3A%22%E5%9F%BA%E4%BA%8ER%E8%BD%AF%E4%BB%B6%E7%9A%84%E5%A4%9A%E7%BB%84%E7%8B%AC%E7%AB%8B%E6%A0%B7%E6%9C%AC%E7%A7%A9%E5%92%8C%E6%A3%80%E9%AA%8C%E7%9A%84%E5%A4%9A%E9%87%8D%E6%AF%94%E8%BE%83_%E9%87%91%E8%8B%B1%E8%89%AF.pdf%22%2C%22size%22%3A171203%2C%22type%22%3A%22application%2Fpdf%22%2C%22ext%22%3A%22pdf%22%2C%22progress%22%3A%7B%22percent%22%3A0%7D%2C%22status%22%3A%22done%22%2C%22percent%22%3A0%2C%22card%22%3A%22file%22%7D)
1.示例数据
现在有三组数据A,B,C需要进行Kruskal–Wallis秩和检验,检验这三组数据是否差异显著,三组示例数据如下:
A:3.5,4.0,6.7,5.6,8.9,7.8**(样本容量为6)
B:4.5,3.0,8.0,5.4,6.9,2.8,7.7,3.9**(样本容量为8)
C: 2.1,4.8,3.3,8.8**(样本容量为4)**
2.安装相关包
分析前,先在R上安装包**“spdep”**和包**“pgirmess”。**
install.packages(“spdep”) install.packages(“pgirmess”)
3.分析方法
先使用该软件的核心包(自带的)进行多组独立样本Kruskal–Wallis秩和检验(即先总的检验多个样本数据的差异是否显著),再采用R语言软件的“pgirmess”统计包的kruskalmc函数进行Kruskal–Wallis秩和检验的两两样本比较。(即再对多个样本进行两两差异的显著性分析)
**
4.Kruskal–Wallis秩和检验
(1)方法一(直接法)
这种方法是直接在R语言的命令界面里写入要处理的数据,适合小数据集。
library(spdep) library(pgirmess) A=c(3.5,4.0,6.7,5.6,8.9,7.8) B=c(4.5,3.0,8.0,5.4,6.9,2.8,7.7,3.9) C=c(2.1,4.8,3.3,8.8) jianyan=list(A,B,C)
kruskal.test(jianyan) #①Kruskal-Wallis检验,先打印总体比较的p值
运行结果如下: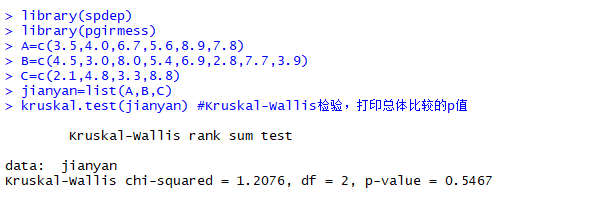
然后进行两两样本的秩和检验:
resp<-c(3.5,4.0,6.7,5.6,8.9,7.8,4.5,3.0,8.0,5.4,6.9,2.8,7.7,3.9,2.1,4.8,3.3,8.8) #将A,B,C三组数据依次放在一个向量里 categ<-factor(rep(1:3,c(6,8,4))) #6,8,4依次为A,B,C对应的样本容量
kruskalmc(resp, categ, probs=0.05) #②再使用kruskalmc函数做两两样本的比较
运行结果如下: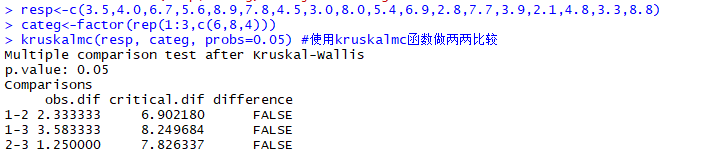
————————————————————————————————————————————————————
【题外话:Kruskal–Wallis秩和检验有时得不到精确的p值,对于Kruskal–Wallis秩和检验差异显著的多个样本,想知道具体差异在哪些样本之间同时又想获得精确的p值,可以采用Nemenyi进行两两样本检验,方法如下】
install.packages(“PMCMRplus”) library(PMCMRplus) library(PMCMR) posthoc.kruskal.nemenyi.test(jianyan) #未校正
运行结果如下:
posthoc.kruskal.nemenyi.test(jianyan,dist=”Chisq”) #校正
运行结果如下: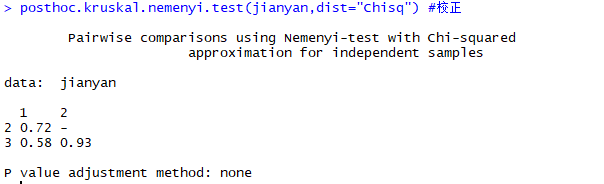
—————————————————————————————————————————————————————-
(2)方法二(表格数据导入法)
先将三组数据放于.csv表格里,表格里的数据的格式如下:
示例数据附件如下:
table.csv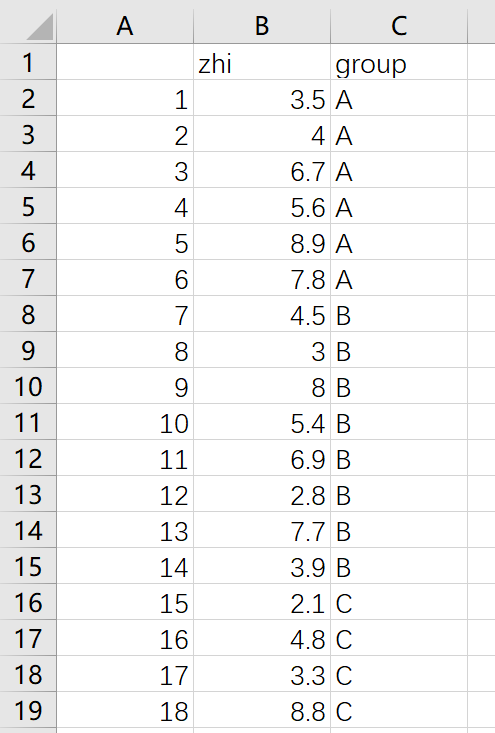
R中输入代码如下:
cd <- read.csv(“table.csv”)#读入数据,建立数据集cd,此处假设table.csv文件已经放在工作目录之下 cd$group <- as.factor(cd$group) kruskal.test(zhi~group, data=cd) #①Kruskal-Wallis检验,先打印总体比较的p值
运行结果如下:
此时假设 “spdep”包和“pgirmess”包已经安装,再进行Kruskal–Wallis秩和检验的两两样本比较如下:
library(pgirmess) library(coin) library(multcomp) kruskalmc(zhi~group, data=cd, probs=0.05) #②再使用kruskalmc函数做两两样本的比较
运行结果如下: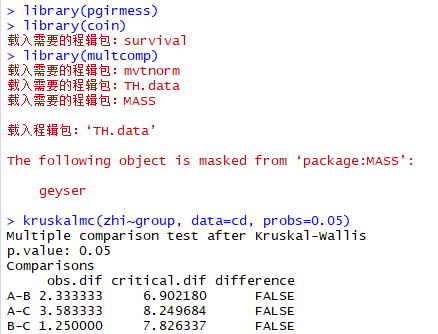
——————————————————————————————————————————————-
PS:使用table.csv里的数据进行Nemenyi检验流程如下:
library(PMCMR) posthoc.kruskal.nemenyi.test(zhi~group,data=cd) #未校正
运行结果如下: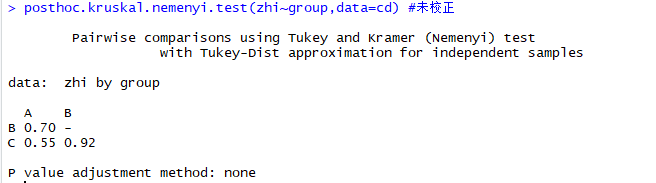
posthoc.kruskal.nemenyi.test(zhi~group,data=cd,dist=”Chisq”) #校正
运行结果如下: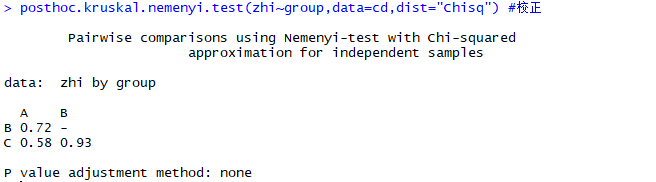
————————————————————————————————————————————————
2019年2月27日初稿
转载请注明出处。如有错误,欢迎及时与我反馈,一起交流学习。

