- 1. 树文件格式
- 2.加载树文件
- 3. 构建映射关系
- 4. Load to Solver
- 5. Set Costs
- 6. Set Host Switch Parameters
- 7. Set Polytomy Parameters
- 8. Set Advanced Genetic Parameters
- 9. Solve Mode
- 10. Solution Viewer
- 11. Viewer Basics
- 12. 寄生物树上的节点可拖动
- 13. Support Values
- 14. Viewing Polytomies
- 15. 修改解决方案(Solutions)的分步指南
- 16. Saving and Retrieving Solutions
- 17. Stats Mode
- 18.Linux版本
以病毒为例,病毒经常发生跨宿主传播,主要发生在近缘物种之间 。许多人类病毒流行病是由动物跨宿主传播而来,如SARS、MERS、高致病性禽流感等。 然而,有些病毒可以和宿主在很长的时间尺度内共进化,例如乳头瘤病毒可能和其猫科宿主共进化1000多万年。<br /> 欲研究跨宿主传播和与宿主共进化这两种模式在病毒进化中发挥的作用,可以进行宿主和病毒系统发生关系相关性分析,Jana便是这样一个不错的软件。<br /> Jane需要的最少数据量是**一对系统发育树**(以下称为“host”树和“parasite”树,即宿主树和寄生物树,但实际上它们可以是任何一对树,例如物种树和基因树,或其他)。 这些树的分支之间的映射,不必是一对一的。Jane允许将多个“parasite”映射到单个“host”,也可以单个“parasite”映射到多个“host”。<br />** 参考文献**:<Jane: a new tool for the cophylogeny reconstruction problem.><br /> ** 官网链接:**[https://www.cs.hmc.edu/~hadas/jane/tutorial.html#solve](https://www.cs.hmc.edu/~hadas/jane/tutorial.html#solve)
1. 树文件格式
树文件格式为.tree或.nex格式,也可以使用Jane的图形用户界面“Tree Editor”功能(稍后以两种格式之一保存)绘制一对树及其尖端映射。
2.加载树文件
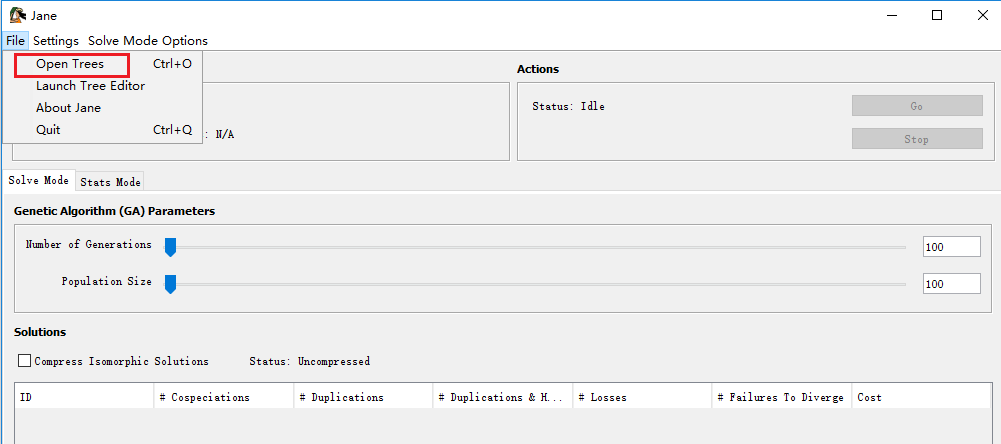
如图,从 “File”菜单栏选择“Open Trees”加载树文件。如果没有树文件,可以使用树构建器“Tree Editor”创建,接下来介绍“Tree Editor”的使用。
3. 构建映射关系
该工具可以从头开始构建树,也可以以.tree或.nex格式加载树。 树构建器可以同时加载完全指定的树和部分指定的树(例如,仅“host”树、仅“parasite”树、或两个树但没有尖端映射)。<br />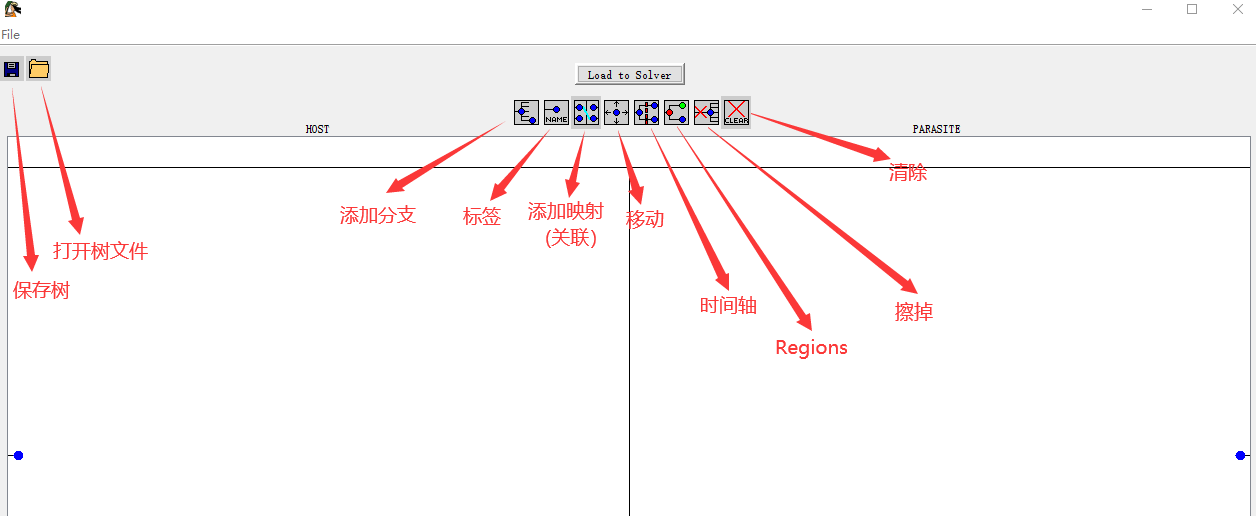<br /> 该工具各个按钮的使用说明见上图,我们可以导入或者自己依据已知的拓扑结构创建两棵树,一棵病毒的进化树,一棵宿主的进化树(当然,它们不一定要满足一一对应关系),然后输入标签,创建两棵树之间的映射,**示例**如下(注意两棵树的位置):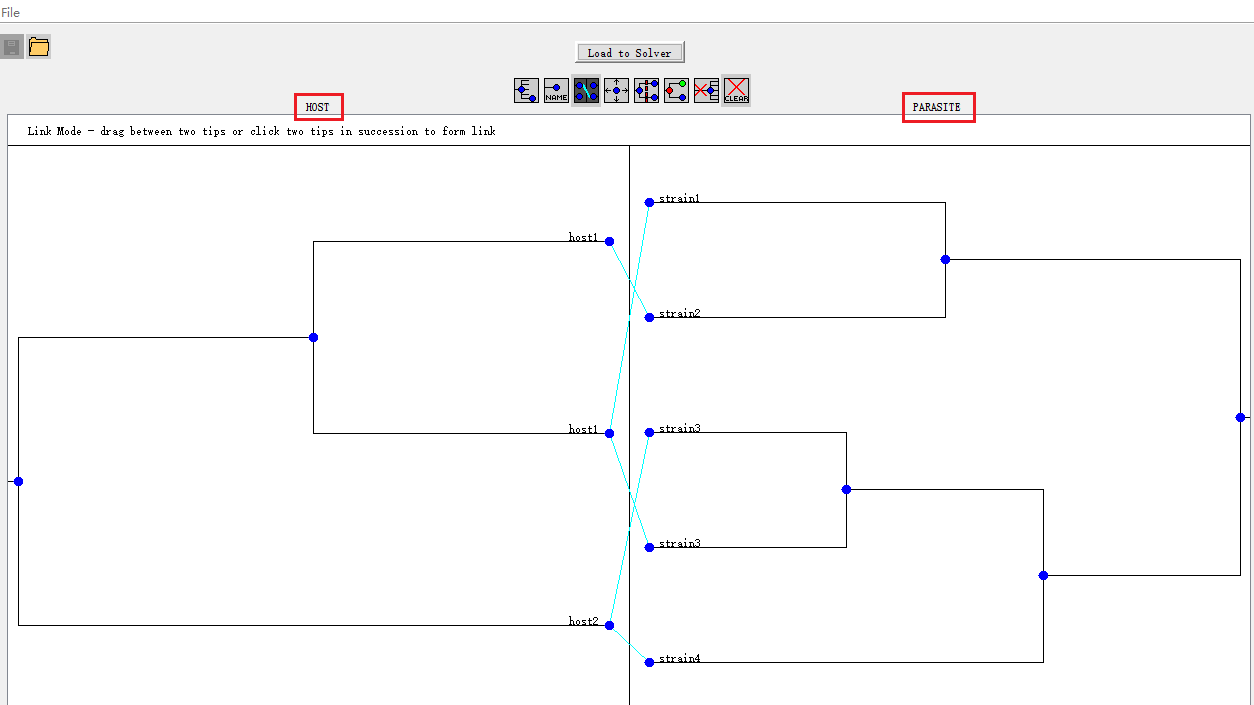<br />** 这里顺便说下“时间轴”这个选项:**<br />** 如果(请注意这个词)**要添加时间轴,请点击“时间轴”按钮,然后输入所需的时区数量。如果在添加时区后希望更改时区数量,请单击左上角的“Change Number of Time Zones”按钮,然后输入新的时区数。输入时区数量后,时区将由红色虚线分隔。要设置节点的时区,请将节点拖到所需的时区中。每个节点的两侧都会有一个红色箭头,将这些箭头拖到另一个时区中可为该节点设置时区。** 再顺便提下“Regions”这个选项:**<br /> Jane软件还可以指定某些节点属于某一区域(例如地理区域),一旦指定了区域,就可以将两个不同区域之间的宿主切换事件的成本设置为任何值。这样,可以对宿主切换事件从一个区域到另一个区域的复制施加不同的“惩罚”。具体而言,**宿主在不同区域的复制和跨宿主传播所需的成本=复制和跨宿主传播所需的成本+特定区域的宿主切换所需成本。**<br />** 如果(请注意这个词)**要添加区域,请单击“**Regions**”按钮切换到区域模式。进入“区域模式”后,鼠标左键拖动选择一组节点,然后输入区域编号。为节点分配区域后,该区域将出现在树上方的key中,并且该节点将更改为相应的颜色。分配区域后,可以通过选择“Edit Region Switching Costs”按钮并输入所需成本来设置区域转换成本。从一个区域到另一个区域的切换将是基于跨宿主传播的成本和额外的区域间宿主切换成本的总和。**表示“无穷大”的成本“ i”可用于阻止所有宿主在两个区域之间切换**,区域切换成本将显示在区域键旁边。<br />**注意:**.nex格式的树文件不支持“**Regions**”功能。
最后说下“擦掉”按钮,可用于删除节点或链接。而“清除”按钮则可以清除整棵树。
4. Load to Solver
树映射关系搭建完成后,可以使用“Load to Solver”按钮保存文件并在Jane的主界面中打开它(保存后会自动打开)。 如果尚未保存文件,系统将提示您保存文件,然后文件将在主界面中打开,可以在“[Solve Mode](https://www.cs.hmc.edu/~hadas/jane/tutorial.html#solve) ”模式或者 “[Stats Mode](https://www.cs.hmc.edu/~hadas/jane/tutorial.html#stats)”模式下运行该文件。这两种模式后面会提到。
5. Set Costs
即不同事件的成本设置。我们首先来了解一下事件,Jane 软件将寄生物与宿主之间的相互关系分为五个事件 :Cospeciation, duplication, duplication with host switch, loss, and failure to diverge.<br /> (1)当寄生物树上某一个代表物种形成事件的节点与宿主树上某个节点相对应时,即视为发生共进化事件(Cospeciation);<br /> (2)当寄生物树的两个节点与宿主树同一个宿主节点对应时,则被发生了一次复制(duplication)事件;<br /> (3) 当寄生物树发生复制事件的两个节点对应宿主树上不同顶点时 , 即视为发生了复制和跨宿主传播 (duplication with host switch)事件;<br /> (4)当寄生物树没有相应节点与宿主树上对应宿主节点对应时,就会发生丢失(loss)事件;<br /> (5)寄生物树有支系未能分叉成功直接导致的是分枝失败(failure to diverge)事件 。<br /> Jane 对每个事件都赋成本值(cost)。赋值成功之后进行最小化总成本的计算。通过产生随机寄生物树来评估宿主与病毒系统发生一致性。<br /> 在“settings”菜单栏里选择“Set Costs”,如下:<br /> 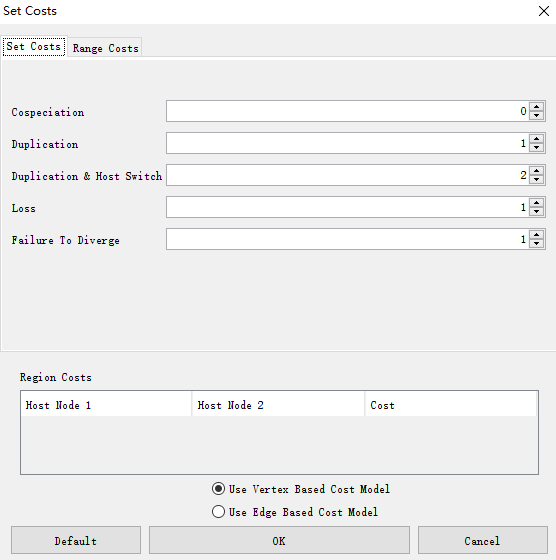<br /> Jane要求事件成本为整数。 请注意,如果希望获得更高的精度,则可以通过简单地按比例增加成本(例如,“100”和“101”的成本类似于“ 1.00”和“ 1.01”)来模拟。<br /> 由于通常很难准确估计事件的相对成本,因此Jane还支持设置成本范围。 例如,我们可以指定共进化成本为0,复制成本为1至3,复制和跨宿主传播成本为3至5,等等。然后Jane将为给定范围内的每种成本组合计算解决方案,之后可以浏览这些成本组合,找到每种事件的最佳解决方案。要使用此功能,请选择“Range Costs”选项,如上图所示。 请注意,Jane的运行时间将根据在给定范围内可能产生的成本组合的数量而增加。<br /> 如果要解决的树具有区域信息,则可以通过“Set Costs”对话框底部的“Region Costs”部分来更改区域之间的复制和跨宿主传播所需成本。
6. Set Host Switch Parameters
设置跨物种传播参数。除了可以自定义跨物种传播的成本外,Jane还允许完全禁止在树中“相距太远”的部分之间进行任何跨物种传播。 特别是,通过“Setting”菜单的“Set Host Switch Parameters”选项,可以在宿主开关上设置最大距离。 如果沿着树边缘的两个节点之间的图论距离大于给定值,Jane将不允许发生跨物种传播。<br /> 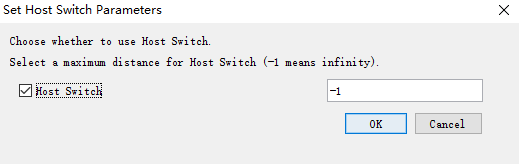<br /> 如图,-1意味着无穷大的距离。
7. Set Polytomy Parameters
Jane inteprets polytomies (multifurcations) as soft polytomies - polytomies that should be ultimately resolved into a sequence of bifurcations. Jane attempts to resolve polytomies in both the host and parasite trees in a way that minimizes the total cost of the resulting cophylogeny reconstruction.<br /> 我们可以在“Set Polytomy Parameters”菜单中控制两个选项。 “Ensure Sequential Resolutions”选项(默认设置)可确保每个导致“快速连续”发生的多歧分枝结构(三分枝以上)的分枝解析度,在多歧分枝结构的两次分叉之间,树的任何部分都不会同时发生其他物种形成事件。 相反,通过关闭此选项,可以将软多歧分枝结构分解为一系列“缓慢”发生的分叉,这意味着树中的其他事件可以在时间上与这些分叉交错。 这两个选项可能会导致处理方案略有不同。<br /> 同样,可以切换“Prevent Mid-Polytomy Events”选项。 如果此选项为开(默认),则不会有复制或跨物种传播事件涉及为将多歧分枝结构分解为一系列分叉而创建的边缘。
8. Set Advanced Genetic Parameters
即设置高级遗传参数。大多数人永远不会点击这个菜单。 但是,如果你好奇或想调整Jane的处理方式,请继续往下看。<br />** **<br />** **<br /> **菜单栏:Settings > Set Advanced GA Parameters 可用于设置遗传算法中的突变率和选择强度。**<br />** **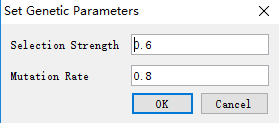<br />** **突变率是介于0和1之间的值,其中0永远不会发生突变,而1总是会发生突变。 选择强度的最小值为0,它对应于随机选择的总体,并且此参数没有上限。 **大多数用户发现默认参数效果很好,因此选择不调整这些参数的值。**<br /> **“Selection Strength”**这个参数确定我们对适应度函数的信任程度。值1表示我们应该只使用前几代中的最佳个体来构建后代,值0表示我们在不考虑适应度函数的情况下从前几代中随机选择,而介于两者之间的值意味着我们从前几代中随机选择,但是偏向于最好的选择方案。<br /> ** “Mutation Rate”**这个参数确定我们在遗传算法中更改时序的频率。 值为1表示在每次复制后都进行突变,值为0表示不进行突变,而介于之间的值给出了突变率。
9. Solve Mode
这个选项,Jane在宿主树上找到了寄生物树的良好重构,并可以查看这些重构。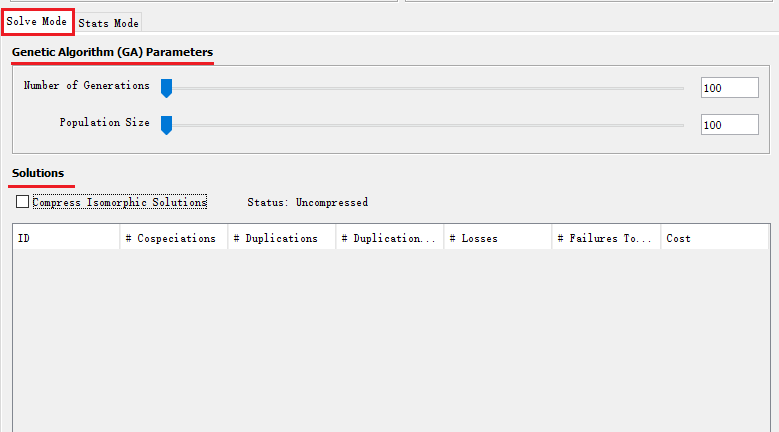
(1)Genetic Algorithm (GA) Parameters
Jane通过生成宿主树内部节点的许多随机相对时序并求解最佳关联,然后应用遗传算法修改时序并生成新的宿主时序候选者,来找到宿主树与寄生物树之间的低成本关联。
在遗传算法中,有两个主要控制参数,分别称为“population size”和“number of generations”。这些参数与算法的内部工作方式相关,与宿主/寄生物系统中正在研究的种群无关。简单地说,“population size”是算法每次迭代时要考虑的不同解的数目,“number of generations”是算法在寻求寄生物树到宿主树的良好重建时执行的迭代次数。
为这两个参数选择较大的值通常会更好,直到增加该值不会造成任何影响的某个点。在Jane里,影响最大的参数是种群大小和世代数。尽管最初的Jane论文建议种群数量设置为世代数量的两倍,但对较大树(200多个节点)的经验表明,种群数量的数量级大于世代数量是最好的。例如,我们发现对于200条序列的树,“最佳点”似乎是30代,1300大小的种群。
(2)The Solution Table
解决方案表,显示有关在解决方案模式下找到的每个解决方案的信息的列表,包括每个事件的发生次数和总成本。可能我们会看到,Jane的许多解决方案似乎都是相同的。 实际上,尽管解决方案看起来相同,但它们基于宿主树中物种形成事件的不同“时间”或相对顺序。 要压缩这些看似相同的解决方案,以便仅显示真正不同的解决方案,请选中“Compress Isomorphic Solutions”按钮。
在“Solve Mode”模式下单击“Go”后,Jane找到了新的解决方案, 找到的最佳解决方案将添加到解决方案表中。需要注意的是:如果在前面设置不同的“Range Costs”,那么这里得到的解决方案也会不一样。下图为上面的映射关系对应的最佳解决方案(默认参数):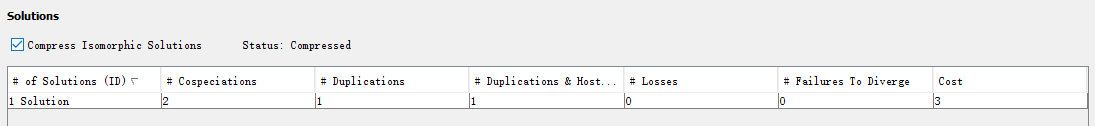
由于每次运行中都会找到大量不同的最佳解决方案,因此可以通过以下方式指定保存的解决方案数量:
Solve Mode > Adjust Number of Solutions
10. Solution Viewer
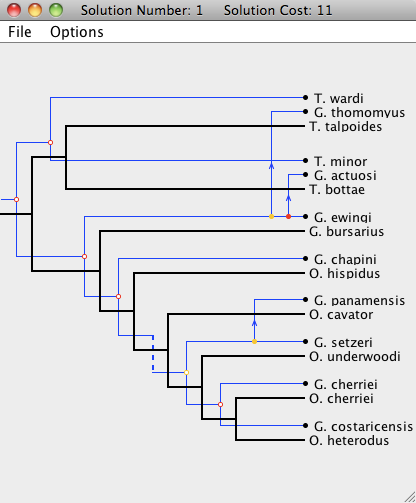
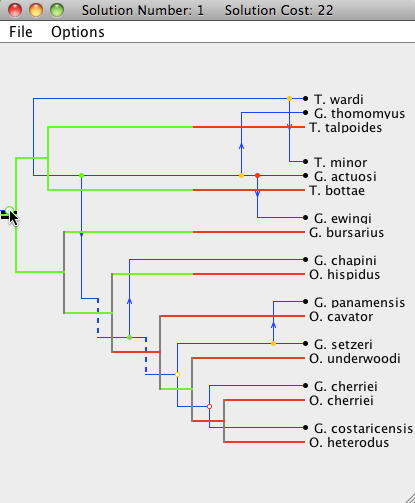
要打开解决方案查看器,只需鼠标左键双击其中一个解决方案即可调出解决方案查看器,**示例**如下图所示:<br /> 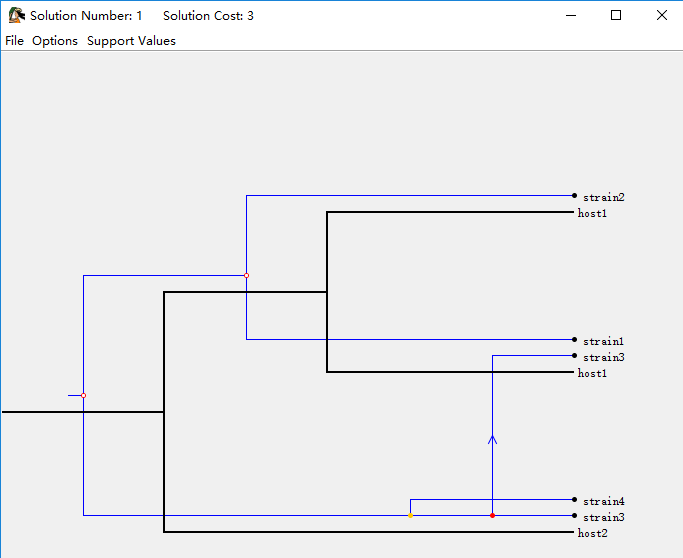<br /> 上图的各线条含义如下:<br /> 
11. Viewer Basics
在**“ Solution Viewer”**里,宿主树用黑色线条绘制,寄生物树用蓝色线条绘制。 事件有五种类型:共进化,复制,跨宿主传播,丢失和分枝失败。<br /> **解决方案查看器将按以下方式呈现每个事件:**<br />** (1)共进化事件(Cospeciation)以空心彩色圆圈标记:**<br />** **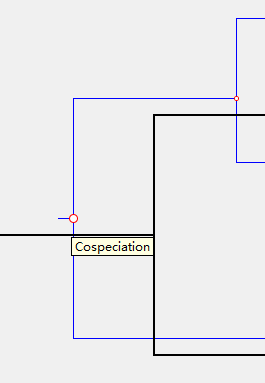<br />** **<br />** (2)复制事件(Duplication)以一个纯色的圆圈标记:**<br />** **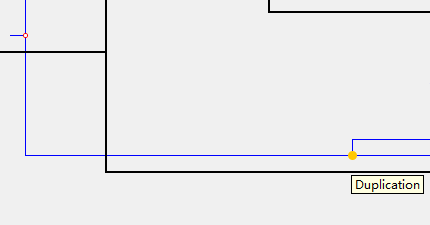
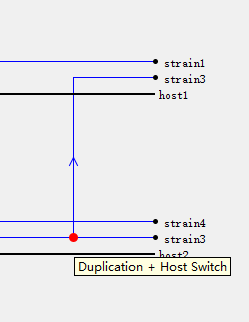
(3)复制和跨宿主传播事件以带有跨物种传播轨迹的箭头及复制事件进行标记:
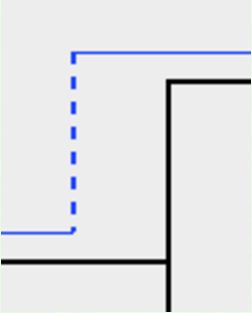
(4)丢失事件用虚线进行标记(本截图来自官网,非上面的示例图)
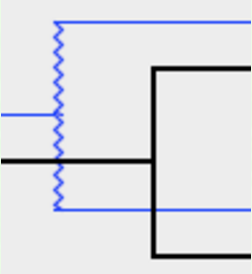
(5)分枝失败事件用锯齿线进行标记(本截图来自官网,非上面的示例图)
注意,寄生物树中的每个节点都用彩色圆圈标记。 颜色表示存在其他可能的关联位置, 绿色节点表示存在一个成本较低的位置,可以在该位置映射寄生物节点及其后代,黄色节点表示存在另一个成本相等的位置,红色节点表示所有其他可能被映射的位置成本较高。
12. 寄生物树上的节点可拖动
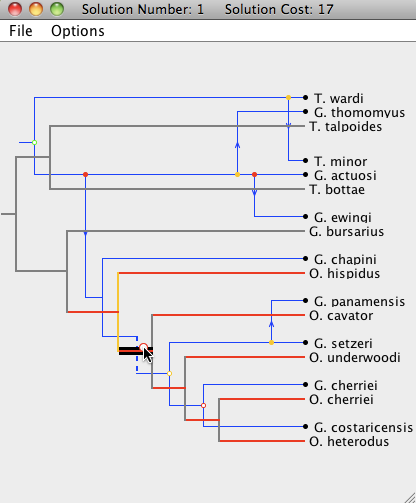
如果有需要,可以将寄生物树的节点拖到新位置,只要该新位置能导致寄生物树在宿主树上嵌入即可,同时宿主树的各个部分将以与成本变化相对应的颜色突出显示,灰色段表示当前无法将寄生物树节点移动到该位置。<br /> 在某些情况下,宿主树上可能有许多成本较低的位置来移动特定的寄生物树节点,知道哪个位置能使得整体成本最低可能很有用。只需双击一个寄生物树节点,即可将该节点“拖放”到成本最低的位置。<br /> 当将寄生物树的节点拖到某个新位置上时,该(寄生物树)节点后代的节点将自动放置在其最佳位置。因此,如果要大面积进行上述的节点移动操作,建议从寄生物树的根节点开始修改,沿树“向下”移动。
13. Support Values
Jane版本4能够在“Solution Viewer”中查看提供的支持值(Support Values),支持值给出了将寄生物树的特定事件映射到宿主树上给定位置的解决方案(solution)百分比。<br /> 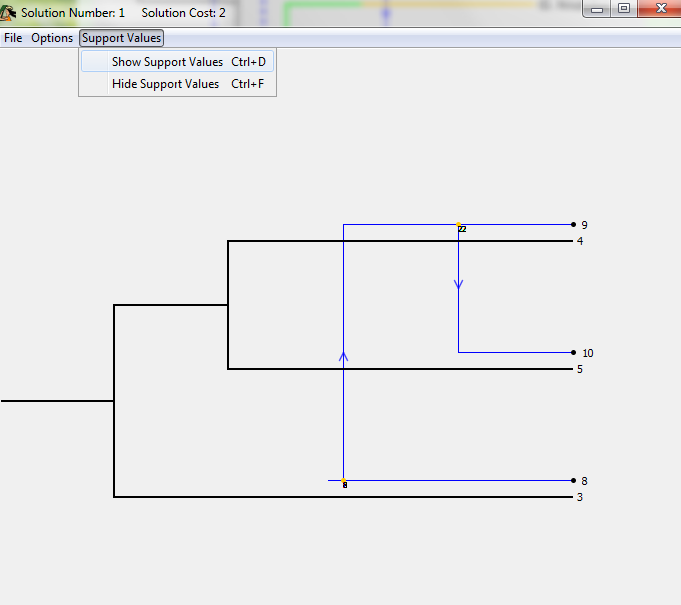
14. Viewing Polytomies
对于Polytomies(多歧分枝结构)树, Jane会尝试自动从所有可能的分辨率中找到最佳解决方案。<br /> (1) Host polytomies in Jane are displayed with edges in purple as shown below:<br /> 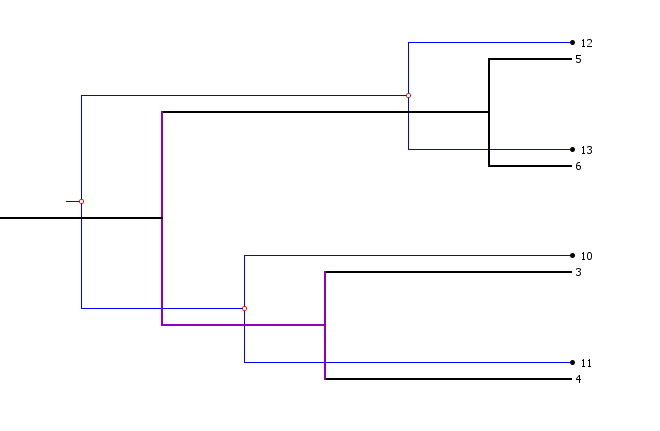<br /> (2) Parasite polytomies are displayed with edges in pink, as below:<br /> 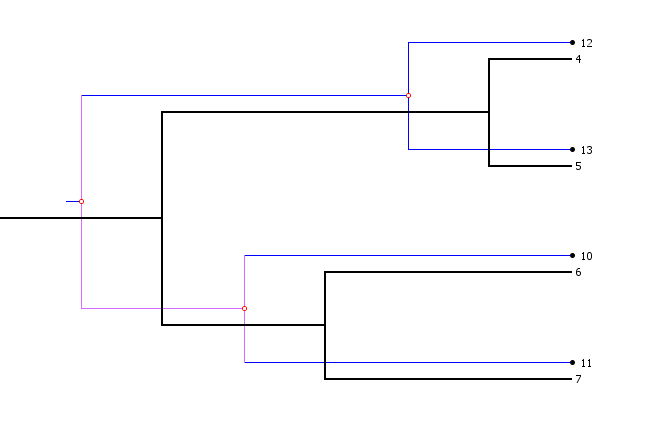
15. 修改解决方案(Solutions)的分步指南
(1)从两棵树的最佳嵌入开始(即打开“Solution Viewer”时的初始状态)
(2)第1步,将寄生物树的根节点移至新位置。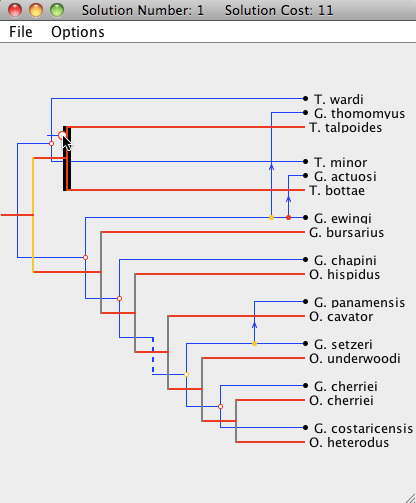
现在,被移动节点的整个子树将会最佳地嵌入到宿主树中。 由于移动节点恰好是根节点,因此将调整整个寄生物树。
(3)第2步,现在将寄生物树另一个节点拖到新位置。 注意,只有被移动节点的子树才会被重新排列,而寄生物树的其他节点将不会改变。
(4)第3步,再次移动寄生物树的根节点。 请注意,此步骤撤消了在“步骤2”中所做的更改。
(5)第4步,如果双击寄生物树的某个节点,它将移动到最佳位置(仅更改节点自身和子树的位置)。如果双击寄生物树的根节点,这将使树恢复到最佳嵌入到宿主树的状态(请参见该节第一张图片)。
16. Saving and Retrieving Solutions
Jane可以将解决方案保存到文件中,以便将来以几种不同的方式使用。当查看解决方案时,单击“File”菜单以查看选项,如下:<br /> 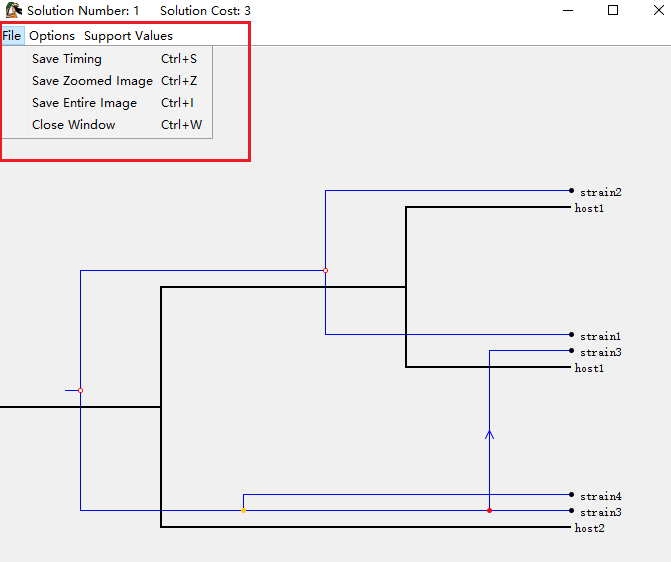<br /> <br />** “Save Timing” **这个选项是以文件格式进行保存,即后期可以再重新加载到Jane中。另外两个选项是以缩放比例或常规比例的图片格式进行保存,以供出版使用。
17. Stats Mode
在统计模式下,Jane随机生成寄生物树或尖端映射的样本,然后求解这些样本以获得其成本。 这些成本用于执行统计分析。 要使用统计模式,只需在主窗口中选择“Stats Mode”选项,然后点击“Go”。<br />
示例并使用默认参数运行结果如下: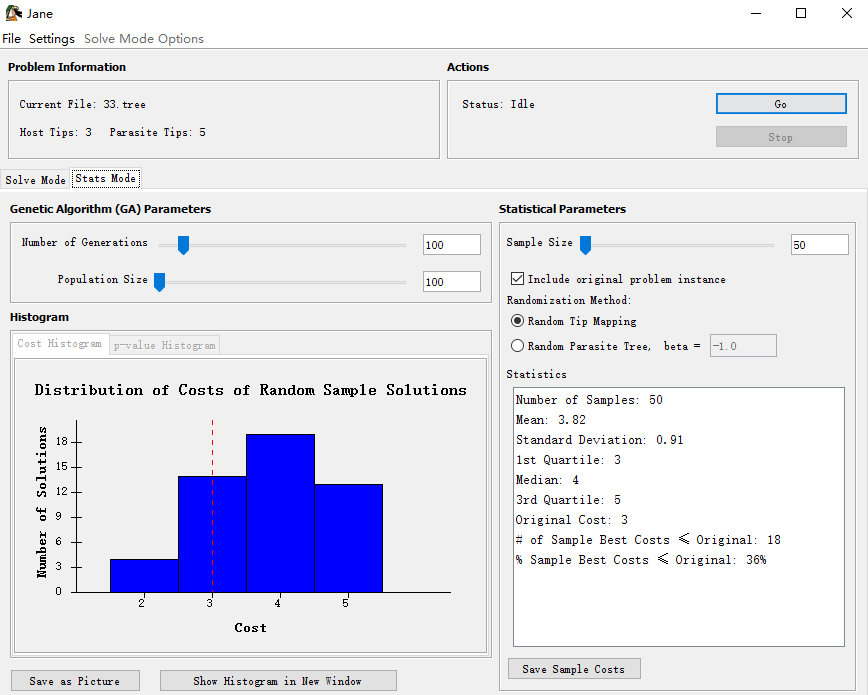
现在我们来看一看参数:
(1)样本大小和是否包含原始问题实例
在“Statistical Parameters”框中,可以设置“Sample Size”(随机样本的数量)。 如果要将原始数据包括在样本中,请选中“Include Original Problem Instance”框,将解决随机样本之外的原始数据并生成比较数据。建议勾选此选项。
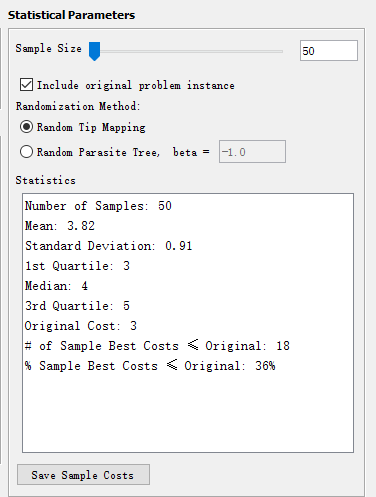
(2)随机化方法(Randomization Method)
①Random Tip Mapping
在这种方法中,尖端映射是随机排列的。每个宿主将具有与“ Original problem instance”相同程度的映射(关联的寄生物的数量)。例如,如果在“ Original problem instance”中有两个宿主,每个宿主的映射程度均为2(即每个宿主均与两个寄生物相连),则进行随机化时会保持这样的映射程度。 如果所有寄生物尖端的映射程度均为1(也就是说,每个寄生物都与1个宿主精确关联),那么将保持从宿主尖端均一地选择随机映射。
如果任何寄生物的映射程度都大于1,则随机映射将保留宿主映射程度,并确保每个寄生物的映射程度至少为1。 但是,将不再从所有此类映射中均一选择该映射,而是以宿主和寄生物之间出现的边缘概率(等于该宿主的映射程度除以寄生物总数)进行随机化。
②Random Parasite Tree
在这种方法中,根据Yule模型生成随机的寄生物树。 可以指定Yule模型“ beta”参数来控制树的平衡。 此参数的下限是-2.0,它会生成完全不平衡的树,并且此参数没有上限。 默认值设置为-1.0。
(3)Viewing Randomization Results
①成本直方图
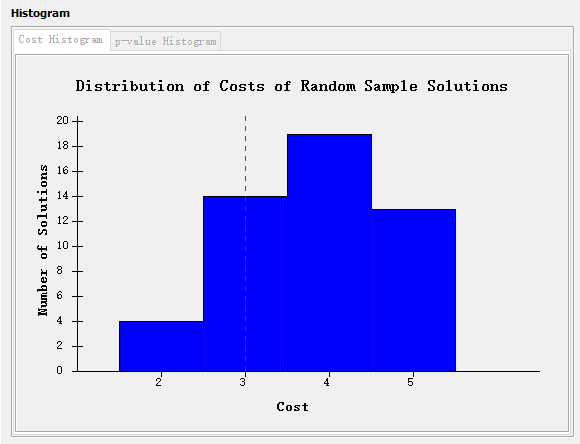
成本直方图部分将以直方图格式显示运行结果,其中横轴表示样本的成本,纵轴表示具有相应成本的样本数。 “ Original problem instance”的解决方案成本将显示为红色虚线。
②**Robustness, range costs, and the p-Value Histogram**<br /> 如果想知道统计结果的鲁棒性。毕竟,如果选择了不同的成本范围,它们的结果可能会大不相同。回想一下,在“ **Settings **”中的“**Set Costs**”菜单中,可以设置不同事件的成本范围(这些事件的成本必须是整数)。如果选择一系列成本,则在按Go之后,将在Jane窗口的底部看到这样的视图。<br />** **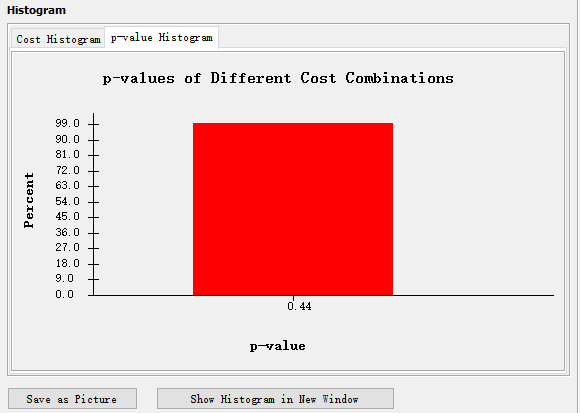<br /> p值直方图中条的颜色编码如下:红色条是当前所选成本值(从窗口底部的下拉菜单中选择的值)的p值。灰色和黑色条表示所有其他p值。 p值小于0.1的条形为灰色阴影,大于0.1的条形为黑色阴影。
18.Linux版本
Jane can be invoked from the command line rather than by launching the graphical user interface as follows. Note that treefile refers to the name of an input file in either .tree or .nex formats.
On Windows:jane-cli.exe treefileOn Macintosh and Linux:./jane-cli.sh treefileOn any platform if you download the platform independent JAR:java -cp Jane.jar edu.hmc.jane.CLI treefile
The -help flag may be used to get more information about the command line options. These options are also summarized below:
By default, Jane will solve for associations of trees described in treefile, then output the best host timing found and the association information for each parasite node. Stats Mode can be invoked by using -stats option; Jane will output the statistics obtained from the generated samples. There are options available for configuring Jane as listed below:
| Option | Description |
|---|---|
| -help | Prints help message |
| -V | Turns on verbose output - Jane will report the minimum cost and number of host timings found at each generation, rather than just at the end. |
| -C | Causes Jane to evaluate costs using a “node-based” cost model rather than the default edge-based cost model. (Note though that node-based cost model is the default in the GUI version of Jane.) |
| -c <_cosp dup switch loss ftd_> | This defines the cost vector to use; i.e. -c 0 1 2 3 4 would cause cospeciations to cost 0, duplications to cost 1, host switches to cost 2, losses/sorting to cost 3, and failures to diverge to cost 4 (default: 0, 1, 1, 2, 1). |
| -m <_value_> | Sets the mutation rate. Appropriate values fall on the interval [0, 1] with 0 being never mutate and 1 being always mutate (default: 0.6). |
| -p <_value_> | Sets the initial population size (default: 30). |
| -i <_value_> | Sets the number of generations that the genetic algorithm should run (default: 30). |
| -s <_value_> | Sets the selection strength. 0 is completely random and there is no upper bound (default: 0.8). |
| -S <_value_> | Sets the maximum host switch distance allowed. -1 causes the distance to be unlimited (default: -1). |
| -stats <_samplesize_> | Switches Jane to Stats Mode and sets the number of samples to |
| -I | When in stats mode, also does a solve of the original tip mapping/trees and prints data comparing it to the random sample. Note that the sample distribution will NOT include the cost of this solution. -stats must be invoked first. |
| -B <_value_> | Switches to parasite tree randomization using the Yule model where the value is Yule parameter. -stats must be invoked first. |
| -o <_filename_> | For Solve Mode, outputs the best host timing file to a file called |
| -silent | This causes Jane to produce absolutely no output to the console (though it will still write any files that are specified). Any operation that would normally print something out will still run, but it won’t print anything out |

