从NCBI数据库下载序列常用的有三种手段,现在一一介绍。
1. NCBI的Taxonomy库(最常用)
网站:https://www.ncbi.nlm.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=10239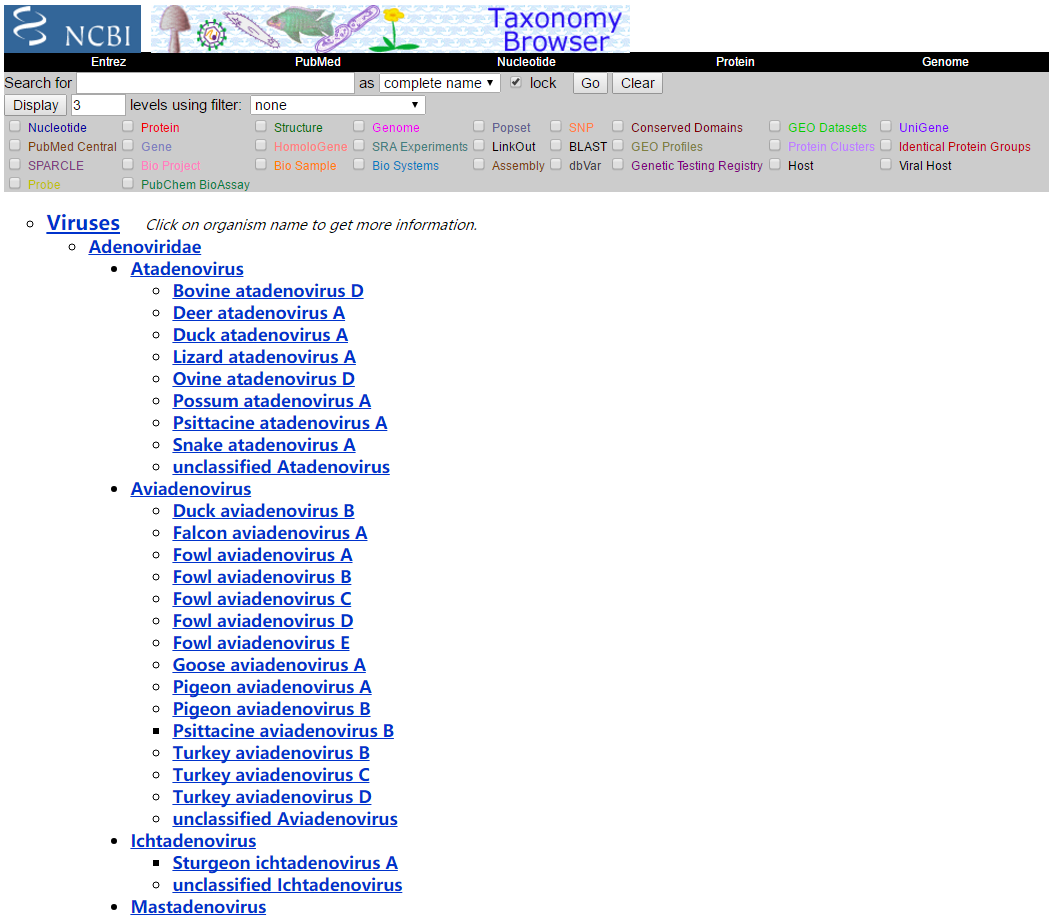
该库可以简单理解为NCBI的后台库,所有的序列如病毒序列按照科属种呈目录分布,一级级找到自己需要下载的目录就行,特点是可以下载某科(亚科)或某属(亚属)的全部序列。
以下载MERS冠状病毒为例:
(1)先在NCBI的核苷酸库里随便搜一株MERS,看看它处在什么位置: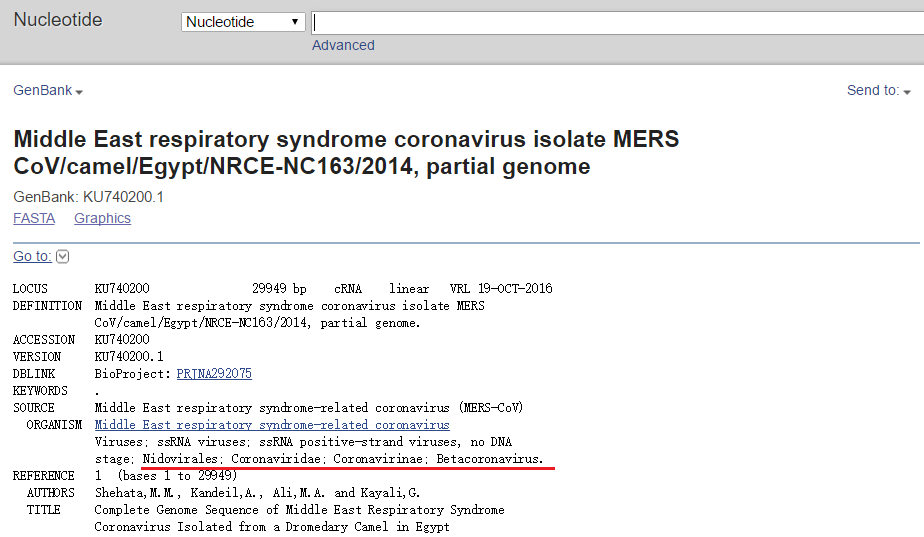
(2)在Taxonomy库的网页里通过CTRL+F键一级级找到最终的Betacoronavirus目录:
点击Cornidovirineae: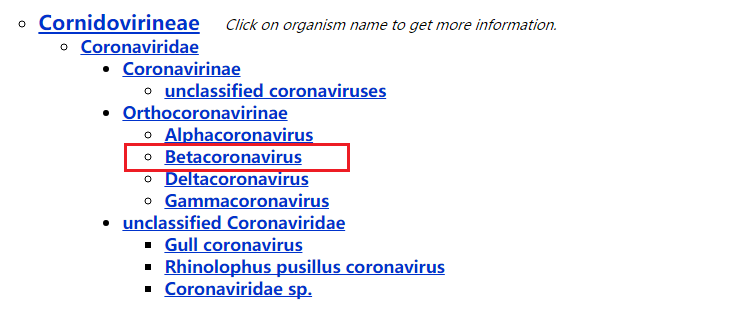
再点击Betacoronavirus:
最终找到我们需要的MERS(前提是自己对自己研究的病毒在分类学上有了解)
(3)搜寻序列
点击上图的红色方框,出现如下: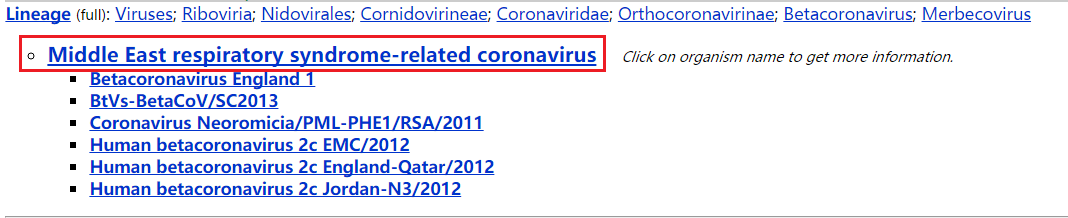
再点击红色方框,出现如下: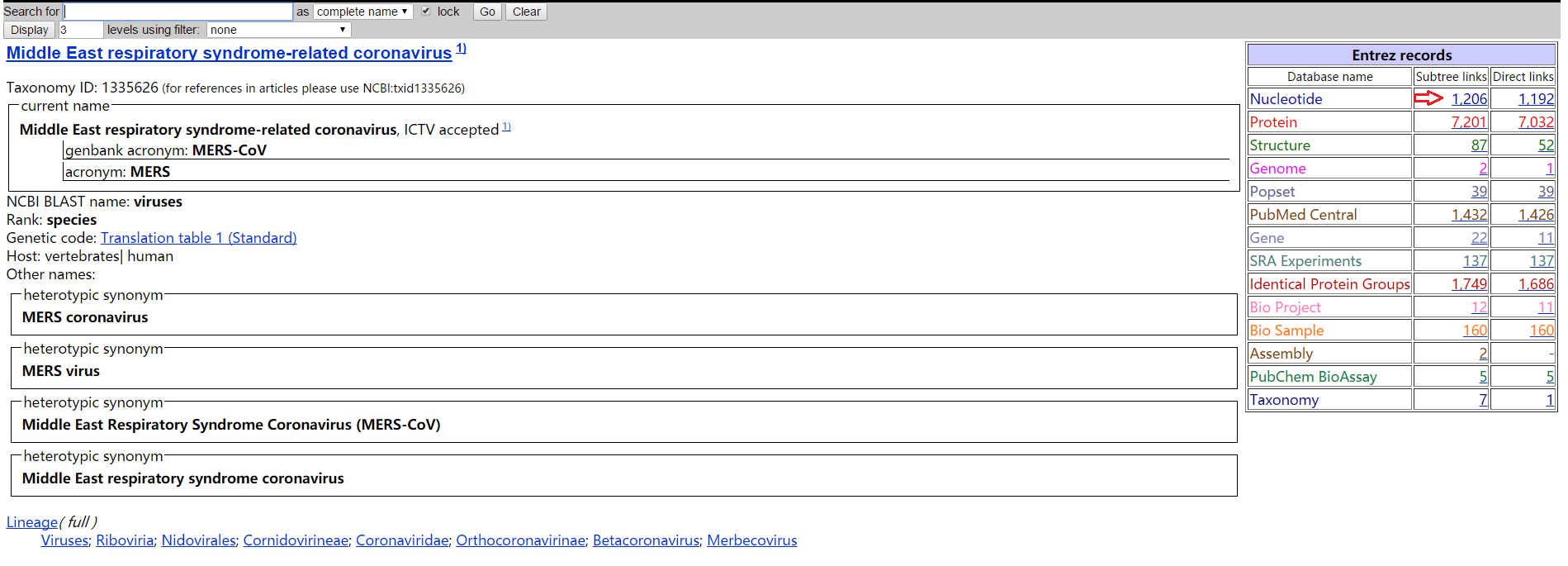
点击上图右上角Nucleotide字样后面的数字,即可进入MERS冠状病毒对应的核苷酸库: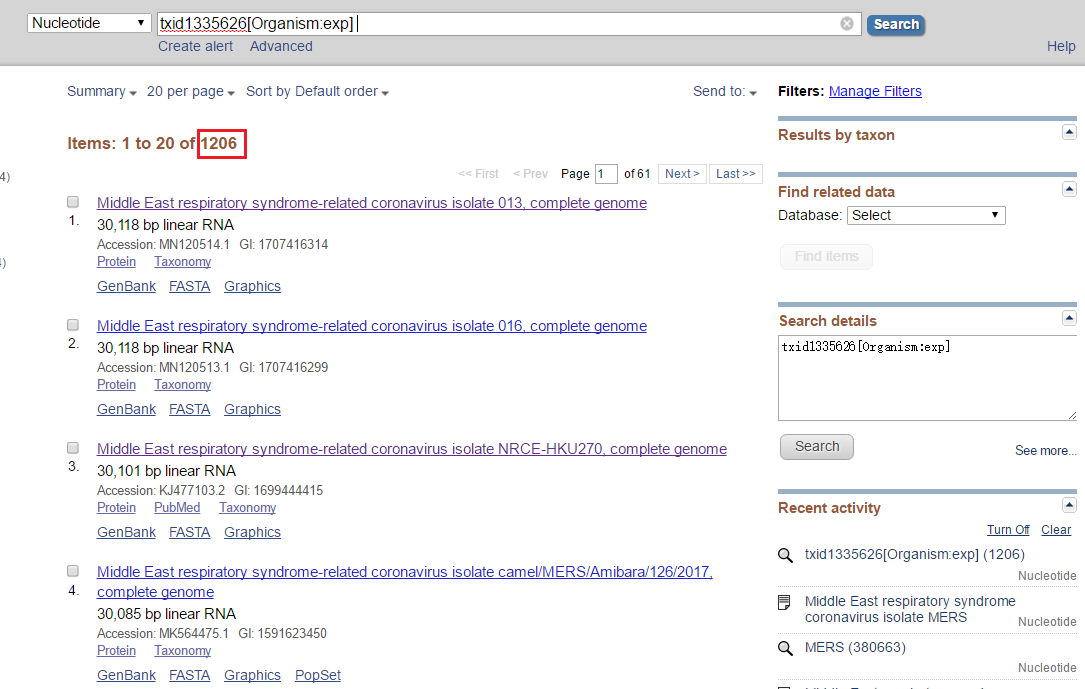
一共有1206条记录(序列),包括片段和全基因组。
如果只想下载全基因组序列怎么办,可以在上面的搜索栏后面加限制条件“complete genome”: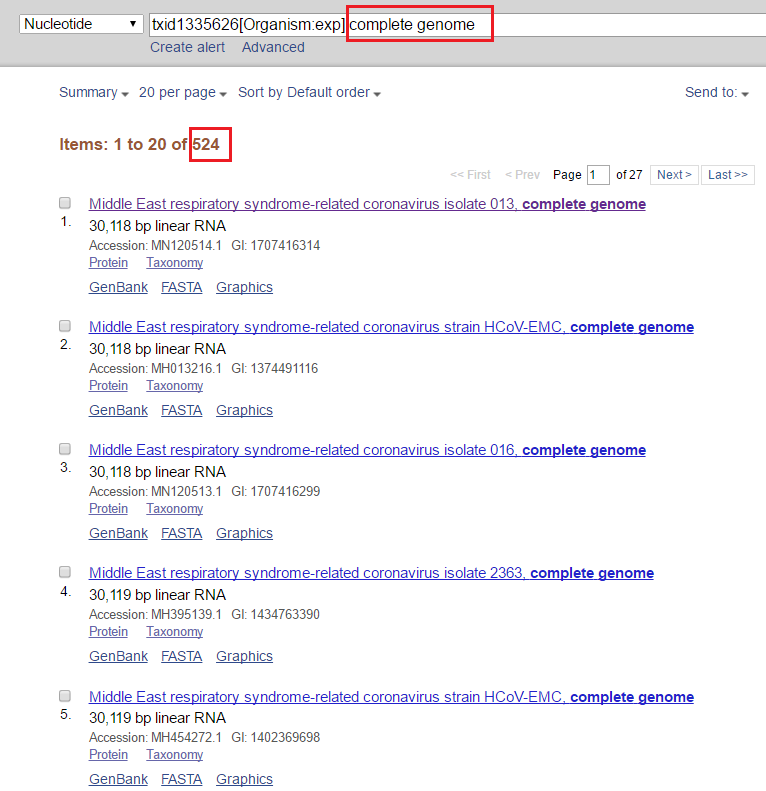
这样找到的全是全基因组序列。
但是注意,这样也有一个风险,有些是全基因组序列,可能没有“complete genome”字符,那怎么办?
我们也可以通过限制序列长度来寻找,比如MERS的全长大约在29kb-35kb,则在后面加上长度限制:
不过这种搜索方式也可能把一些非全基因组的搜索出来,如: 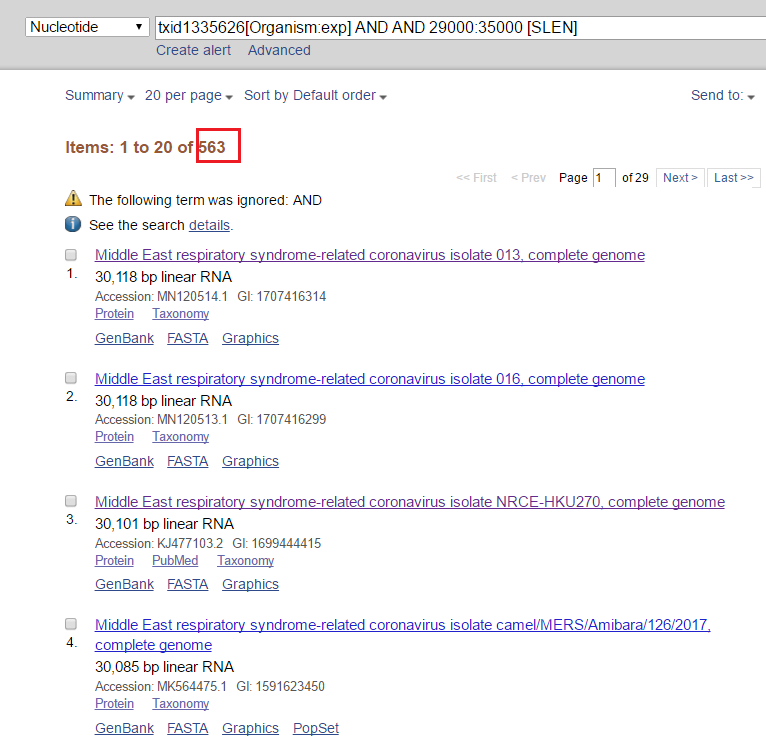
相比前面的加“complete genome”明显多了一些序列,所以后期还需要再筛选。
(4)下载序列
可以下载完整记录(或者可以理解为全基因组)的各种格式的序列: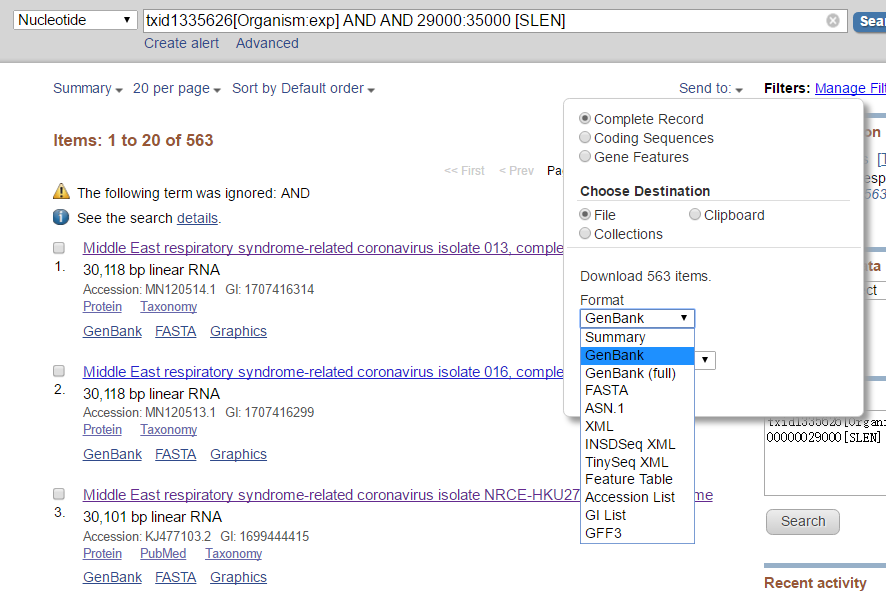
还可以下载编码区的序列,分为核苷酸和氨基酸: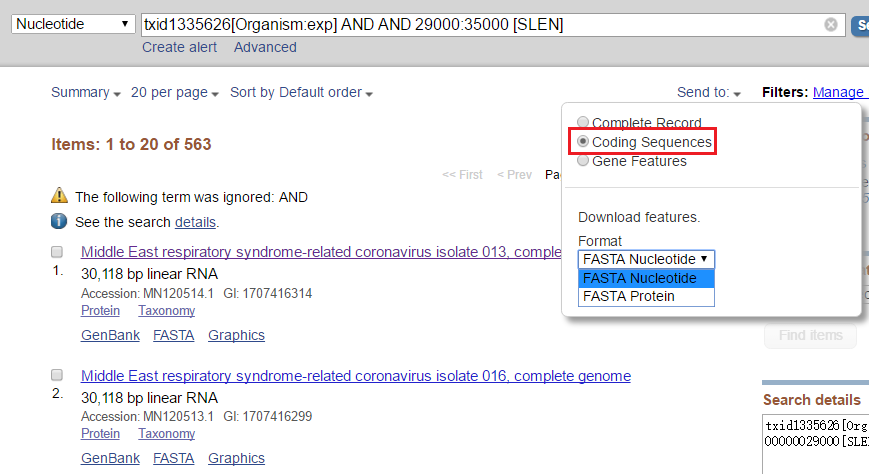
2. NCBI的Nucleotide库里直接搜索病毒名称
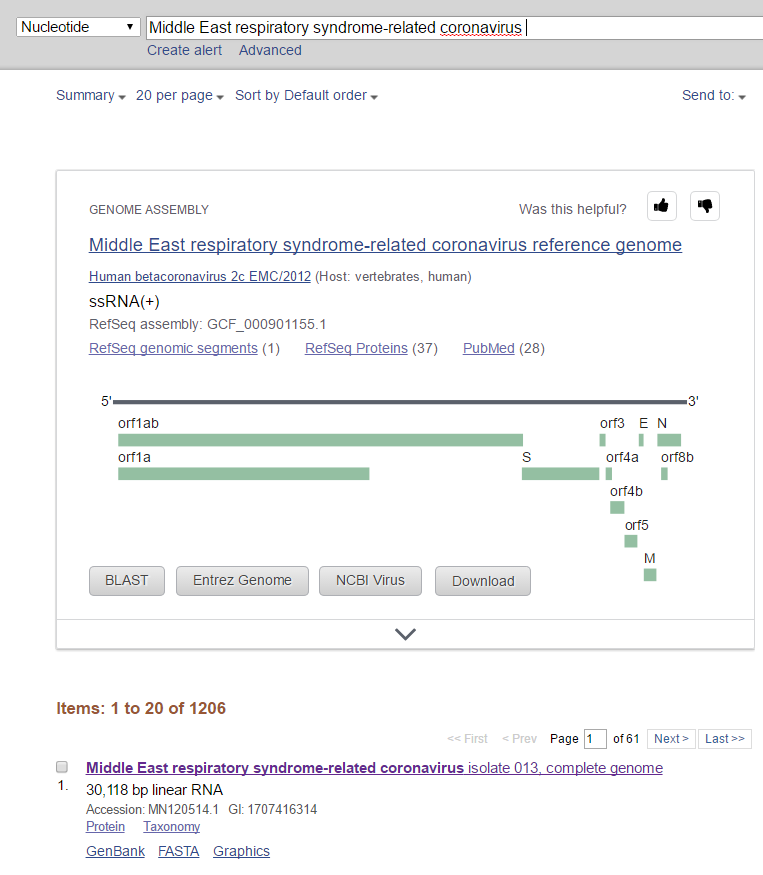
这种方式简单粗暴,但是有时找不全,不推荐,之后下载序列同上。
3. 利用已知的序列GenBank号下载
在已知一批序列GenBank号的前提下,只想下载这些指定的序列,可以利用NCBI的检索方式完成这个工作,即:
将需要下载的序列的GenBank号彼此之间用“|”隔开,放进Nucleotide库的搜索栏里,即可: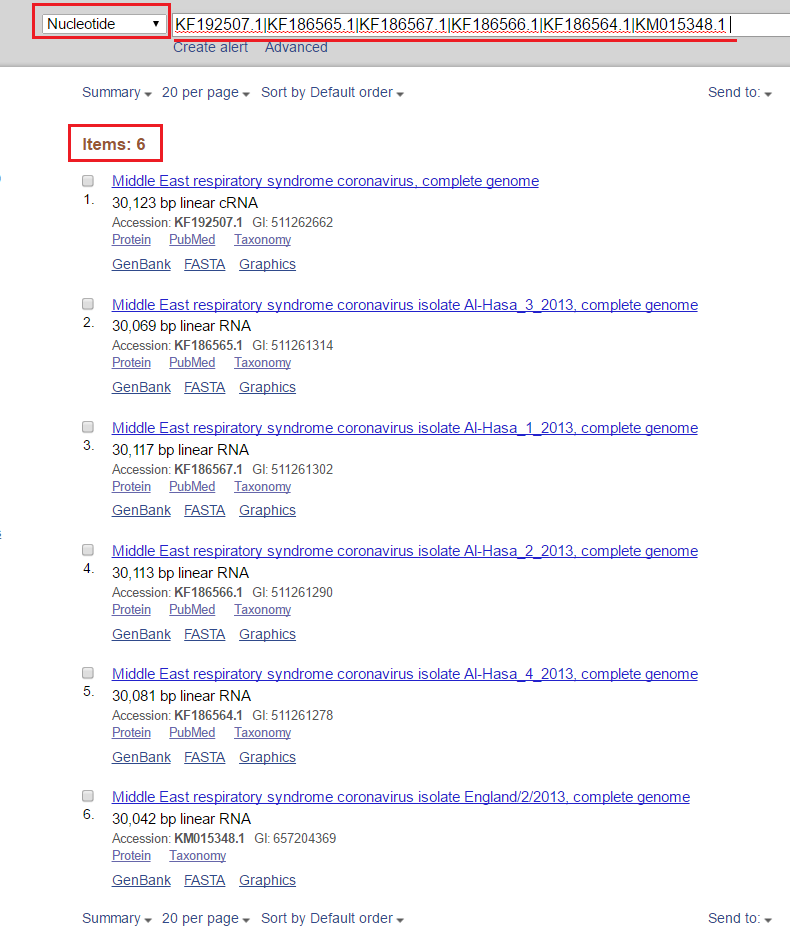
下载序列流程同上。

