前言:PhyML是一款ML法建树的老牌软件,相比RaxML针对核苷酸替换模型只支持GTR模型而言,PhyML在支持的核苷酸模型上可谓是比较多(因为可以自定义模型),本文重点介绍PhyML单线程版本的使用及各参数含义,掌握了单线程版本的使用,多线程版本就变得轻而易举了。
注意:PhyML3.1和3.3有点不同,后面会提到,本文以3.1版本示例。
参考文献:《P h y M L - M a n u a l》PhyML-3.1_manual.pdf
一.建树前先选进化模型
1.核苷酸(Jmodeltest或Modelfinder)/ 氨基酸(Prottest或者Modelfinder)
假定我们的数据是核苷酸序列,并通过jmodeltest或者Modelfinder(modelfinder是2014年的新出软件,比较快,推荐)软件依照AICc标准计算得到的最佳模型如下(下图示例为jmodeltest的结果):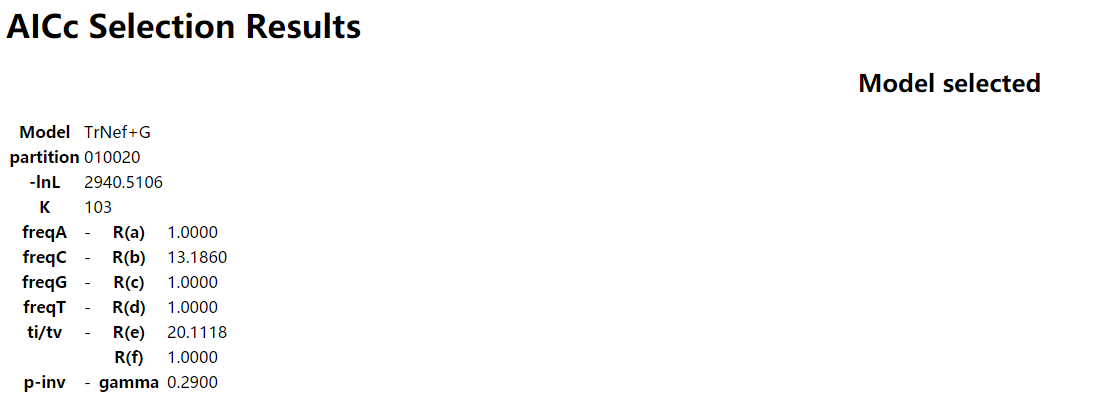
2.上述结果解读
①核苷酸替换模型:TrNef
②替换模型代号:010020
③变异速率模型:Gamma(G)
④R(a)即Rate [AC];R(b)即Rate [AG];R(c)即Rate [AT];R(d)即Rate [CG];R(e)即Rate [CT];R(f)即Rate [GT]
⑤Gamma变异速率的shape值(或叫alpha值):0.2900
这里需要注意的是,在不同软件里,模型的名字有时会不一样,但是其实是同一个模型,需要互相比对不同软件里的模型叫法,最好的方法是去了解该模型的“别称”,然后读你所使用的建树软件的说明书,找到对应模型的代号。
二.PhyML参数设置
1.如何定义外群(outgroup)
在需要设定为外群的序列的名称后面加上符号**“*”**,软件会将它自动设定为外群序列。<br />
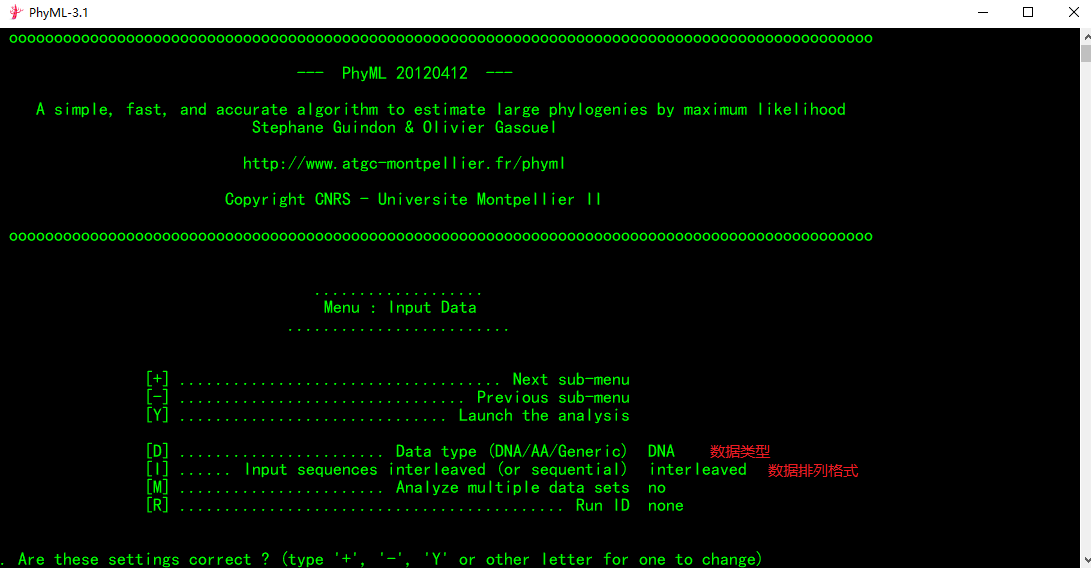
2. 输入文件
运行PhyML后,输入序列文件(.phylip格式的文件)所在的路径。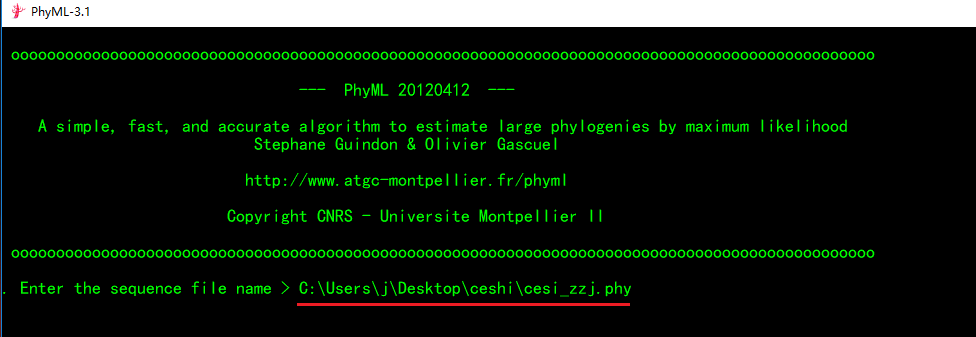
3. 按“Enter”键进入参数设置
4.首先进入的是第一页的参数设置
这一页我们常需要调节的是序列的类型和排列方式,默认是DNA和交叉式(interleaved)排列。本文示例由于我使用的数据是顺序排列式(sequential),故需要进行调整。
调整的方法为在鼠标光标处输入参数前[ ]里的字母(大小写都行),如下: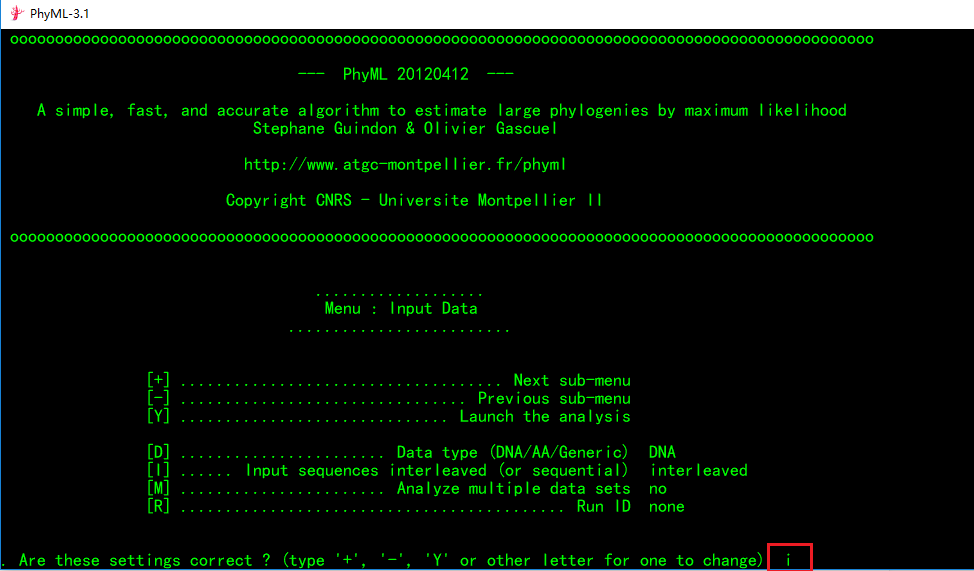
之后按回车键,可见[I]部分的参数改变了,会发现[I]项后的interleaved变为sequential。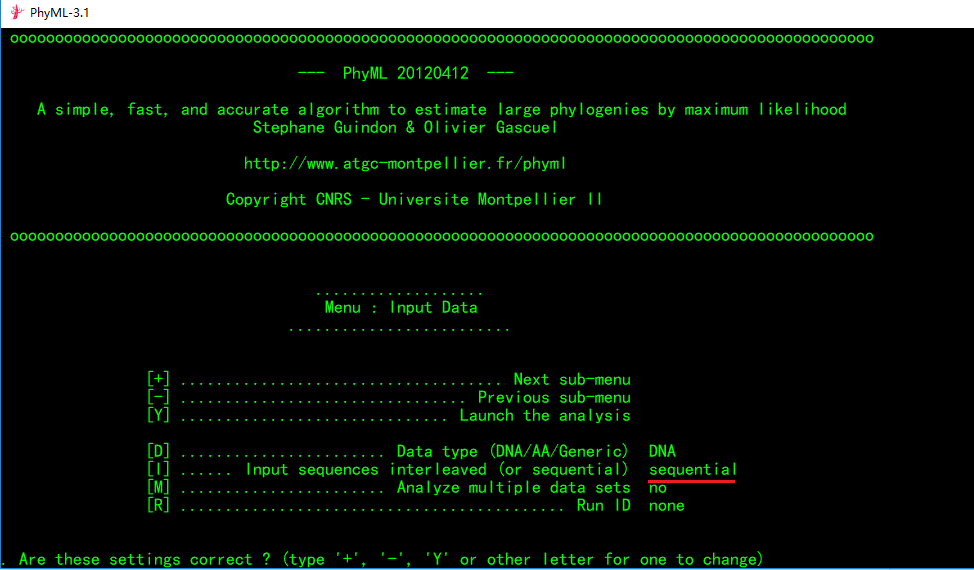
5.输入“+”进入下一页
同样再按回车键,第二页参数如下: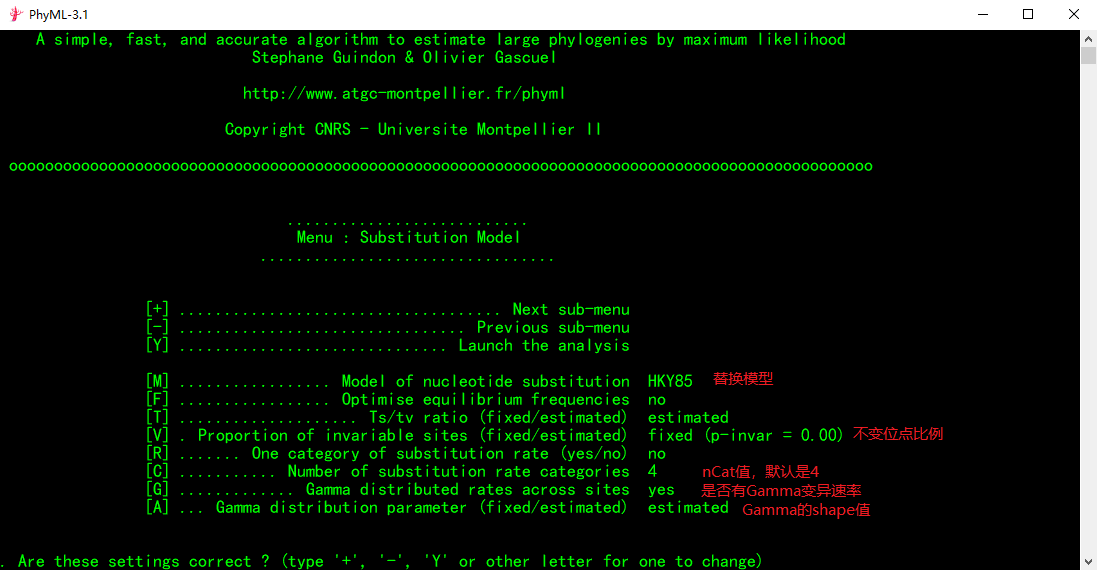
(1)设置进化模型
由于我们示例数据的最佳替换模型是TrNef,我们通过多次在鼠标光标处输入“M”再按“回车键”不断调整模型,会发现没有我们想要的TrNef模型。因为PhyML默认的模型只有HKY85、F84、TN93和GTR,其他的需要我们手动输入。**怎么手动设置替换模型?
①在光标处不断输入M,直到转换模型为Custom,如下图:**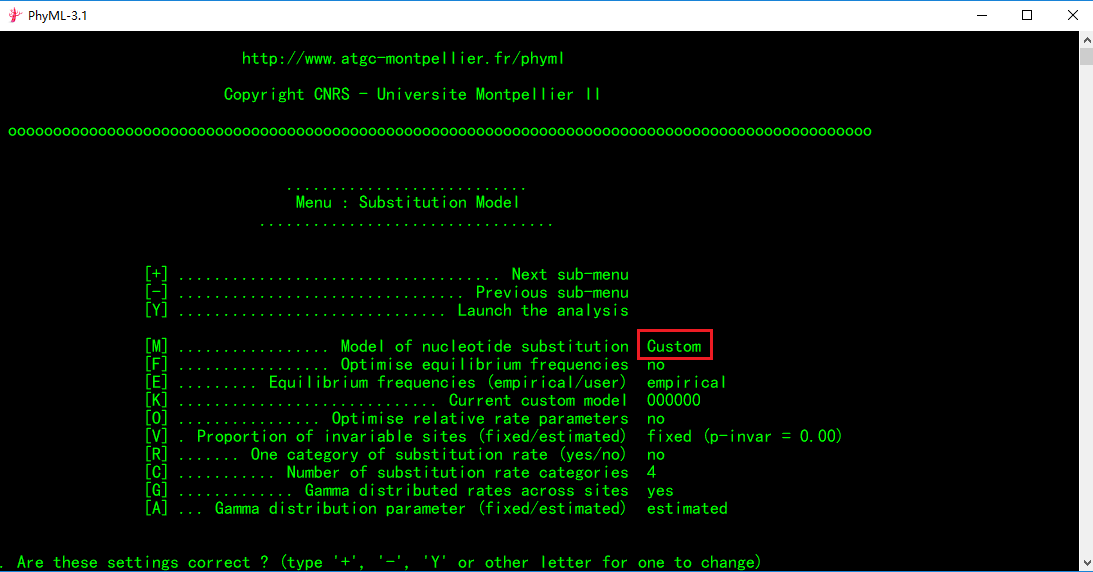
②在光标处输入K(设置[K]项,即模型设置)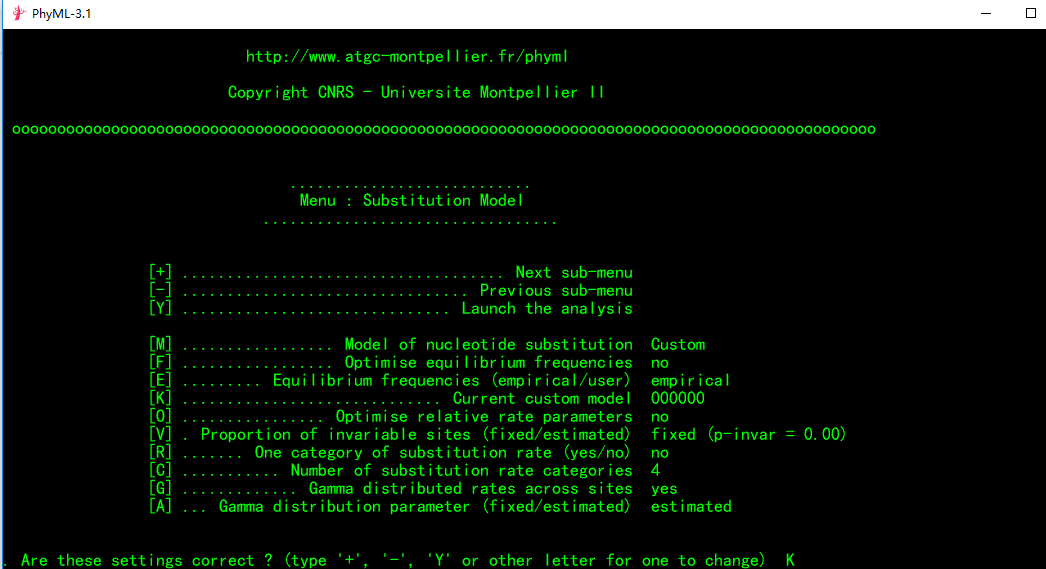
③按回车键后会出现下面: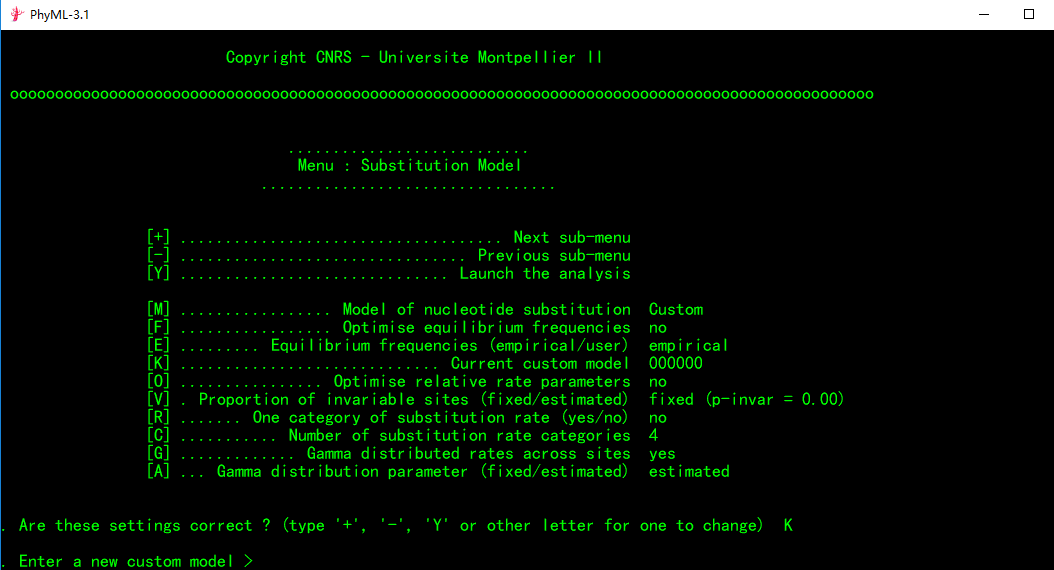
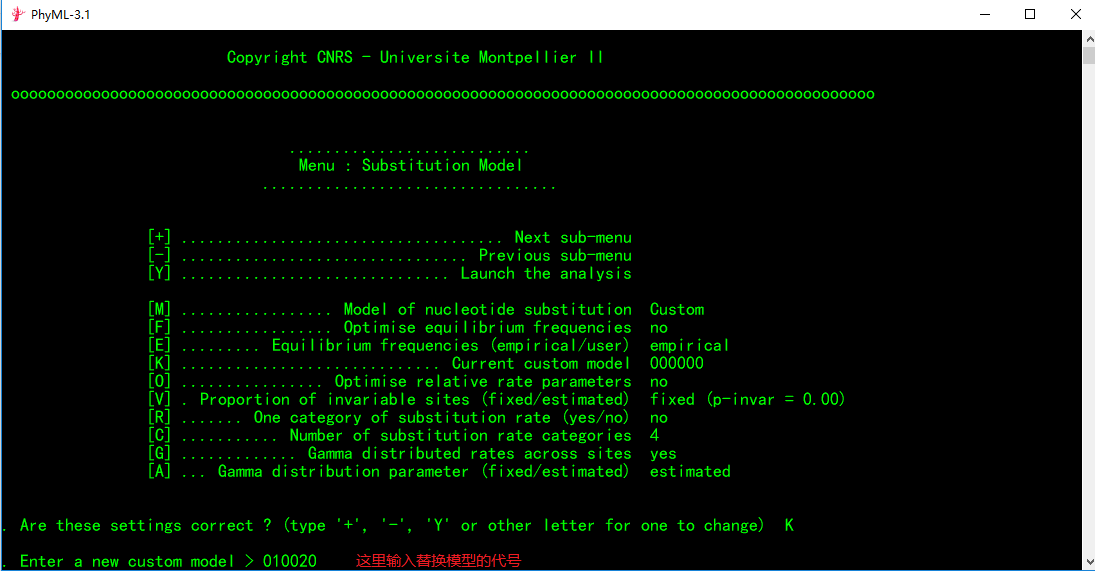
④模型代号输入完毕后,再按回车键,进入模型的参数设置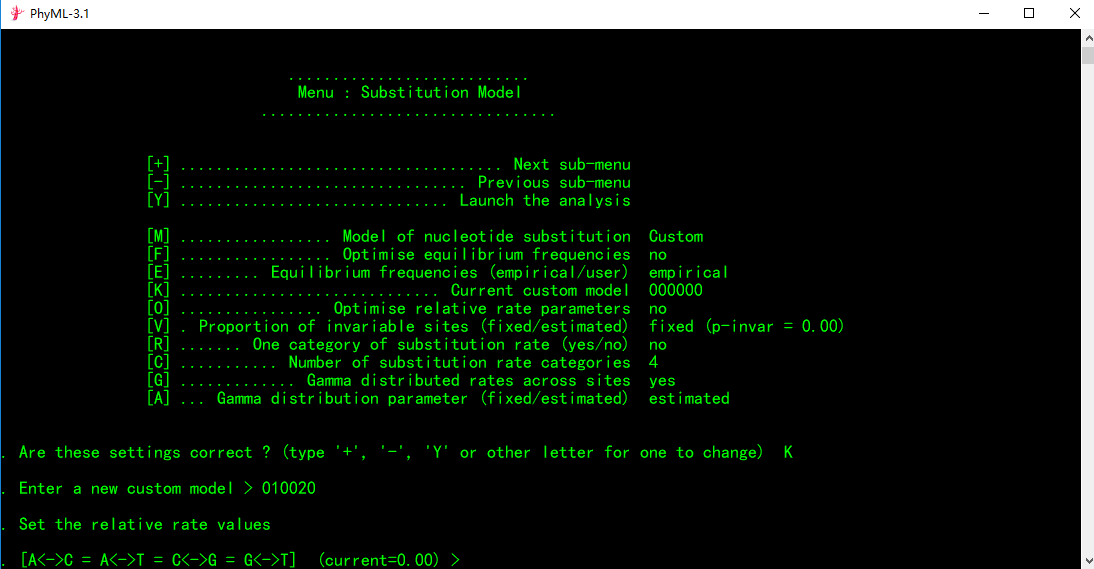
根据给出的提示,依次输入上述jmodeltest测得的三个Rate值(每输一个按一次回车键)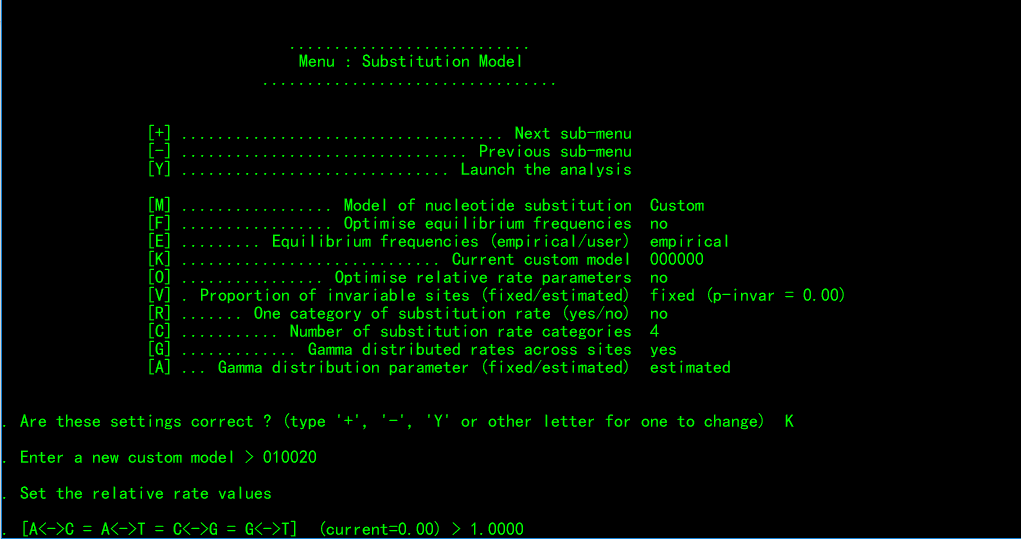
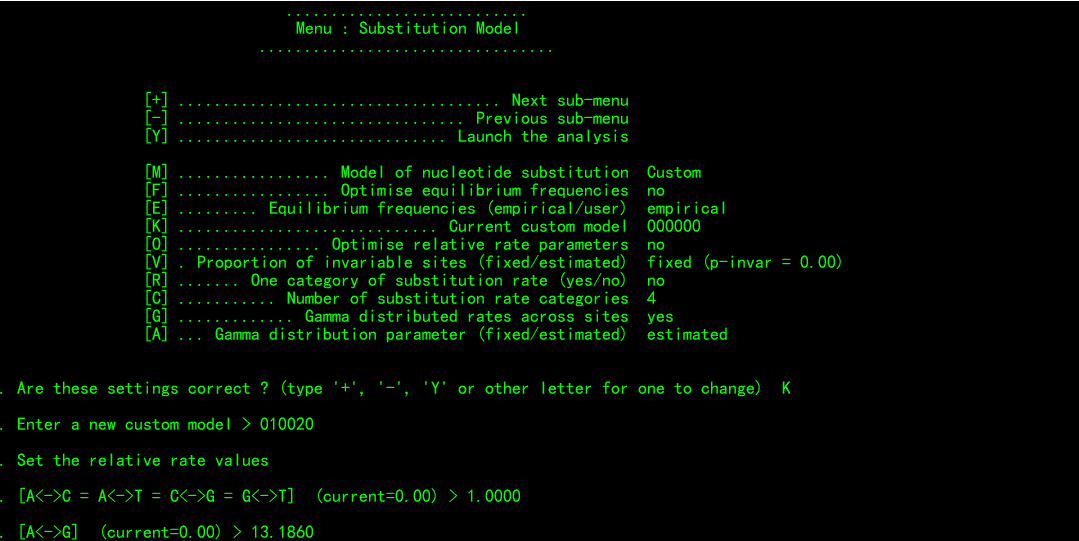
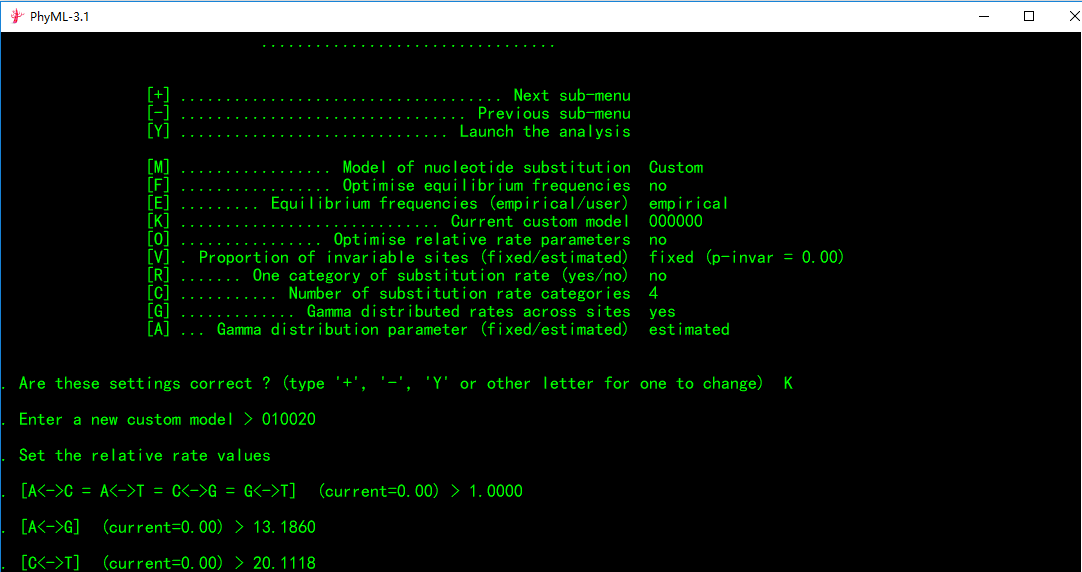
输入结束后按回车键,如下: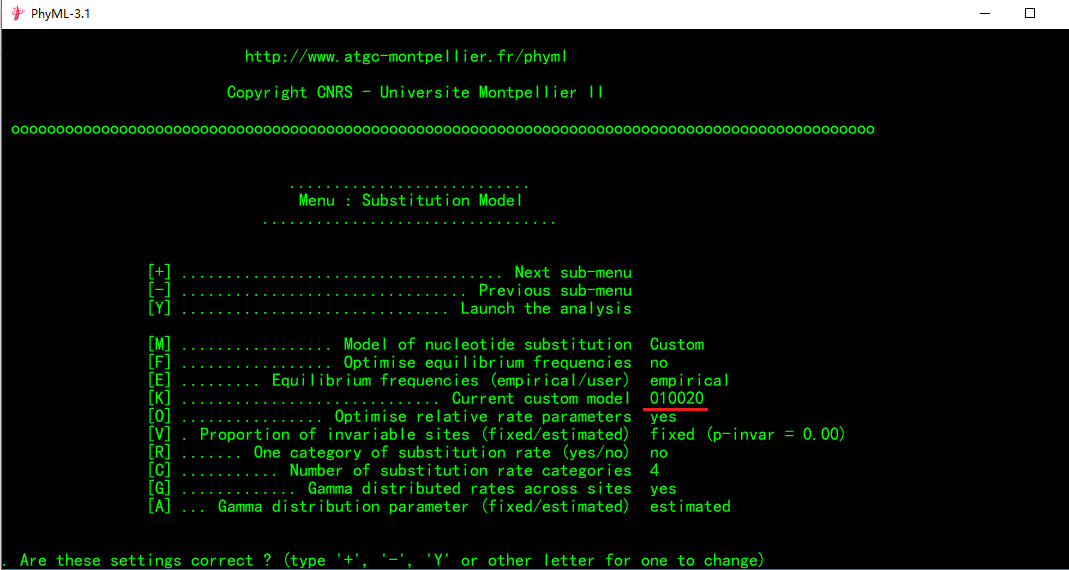
(2)设置Gamma
注意,本文中的示例数据因为通过jmodeltest测得变异速率为Gamma,所以才进行Gamma的设置,如果没有则将[G]后的参数设置为no。
(3)设置Gamma的shape(alpha)值
同样,只有当你的数据测得为Gamma变异速率时,才进行shape的设置(请注意这句话)。
①光标处输入“a”,按回车键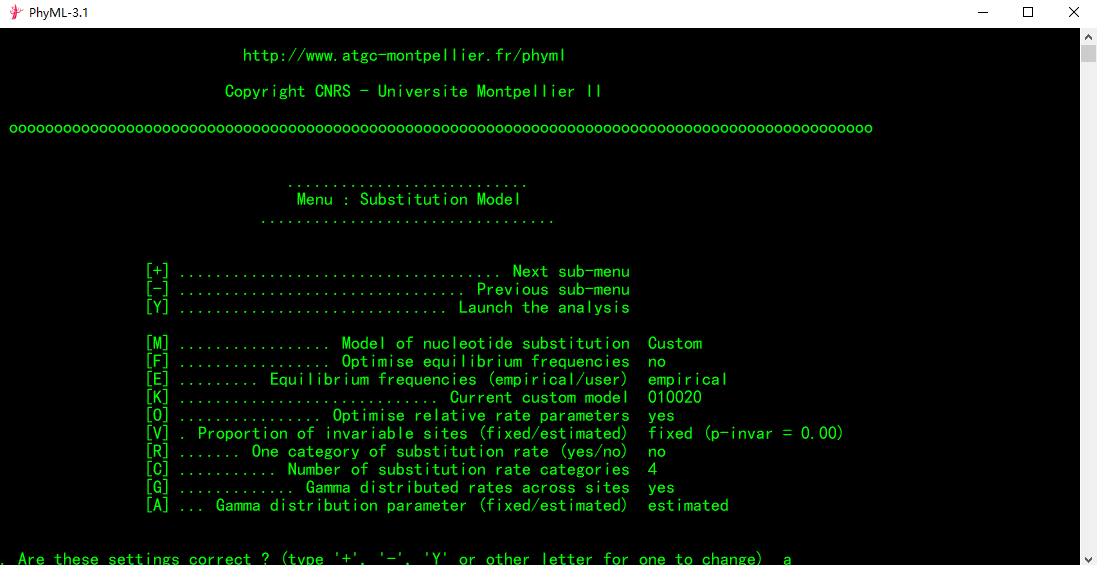
②选择“N”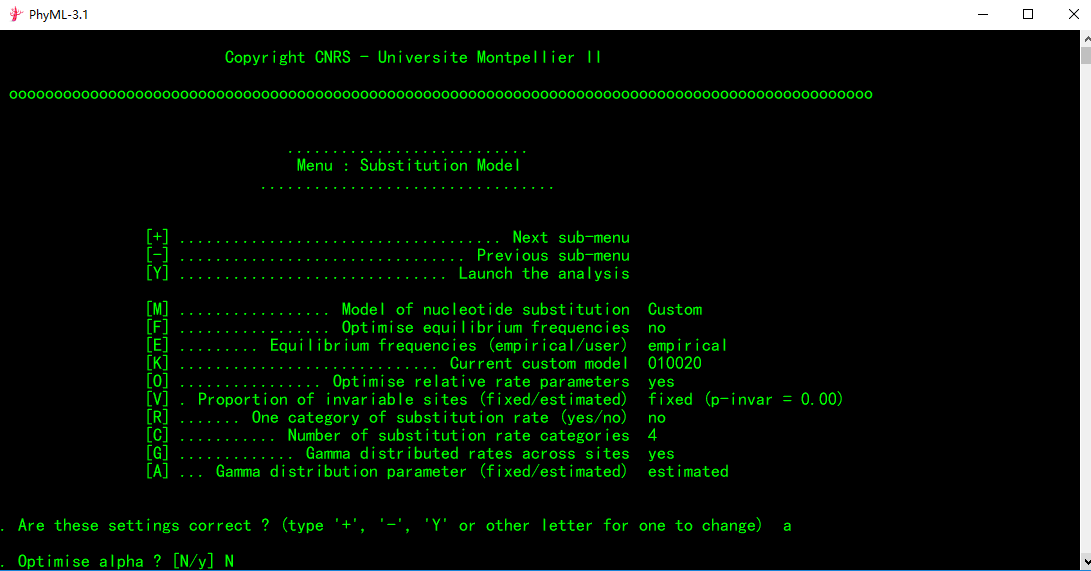
③输入alpha值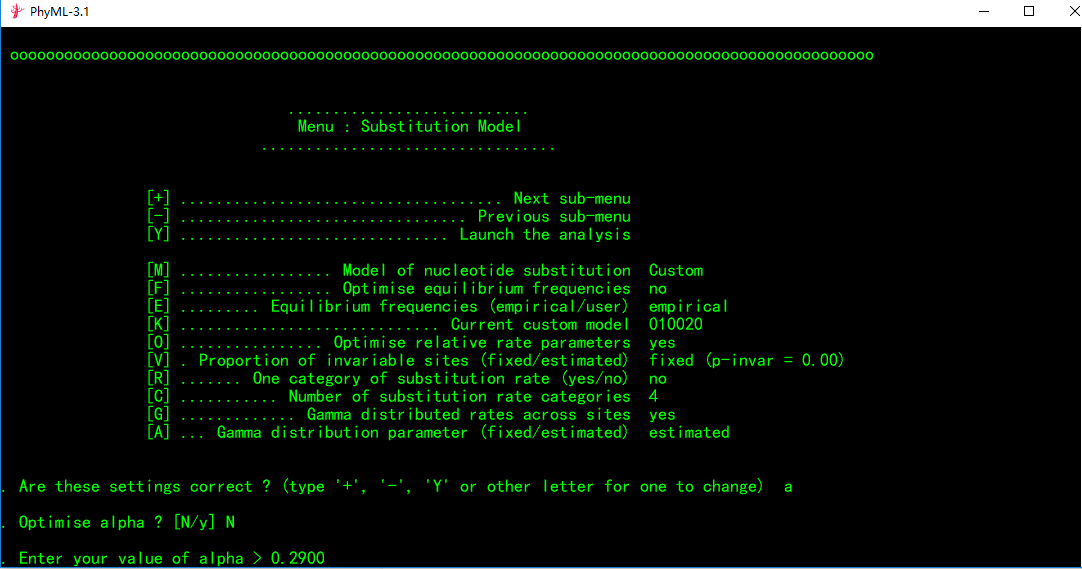
④关于[C]选项 “Number of substitution rate categories”(nCat值)的确定,请参考我的另外一篇文章“Mybayes使用要点”这里不再重复。
6.再次输入“+”进入下一页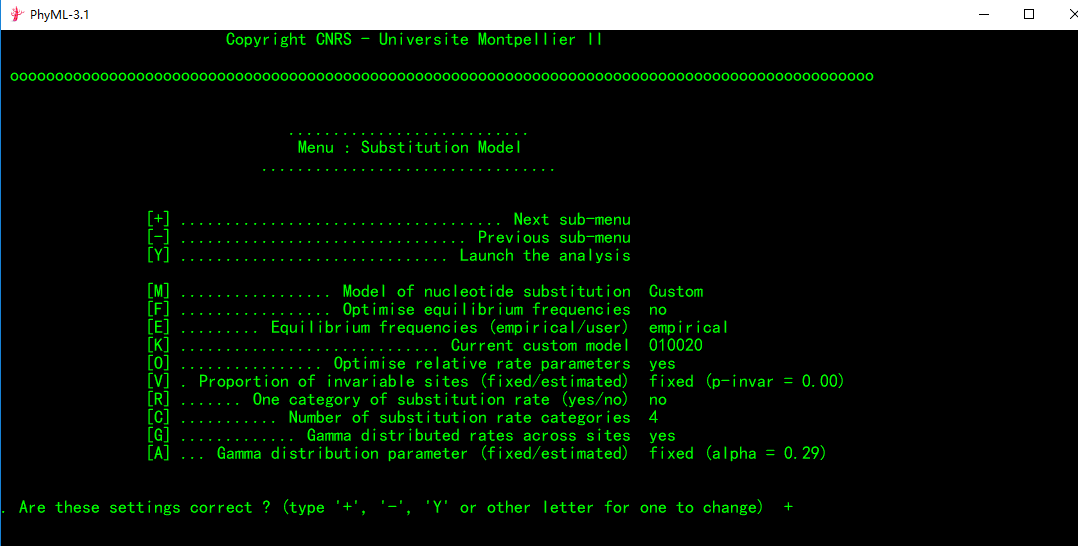
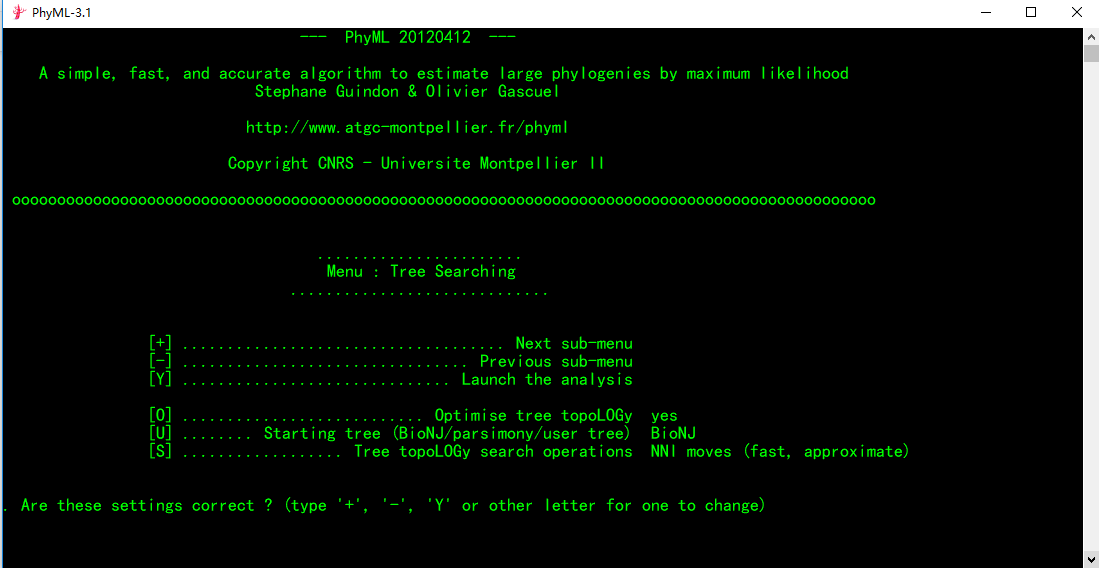
这一页的参数基本不需要调节,如果为了追求准确性,可以输入“s”将[S]后的参数设置为Best of NNI and SPR,需要注意的是,后期软件运行速度也会变慢。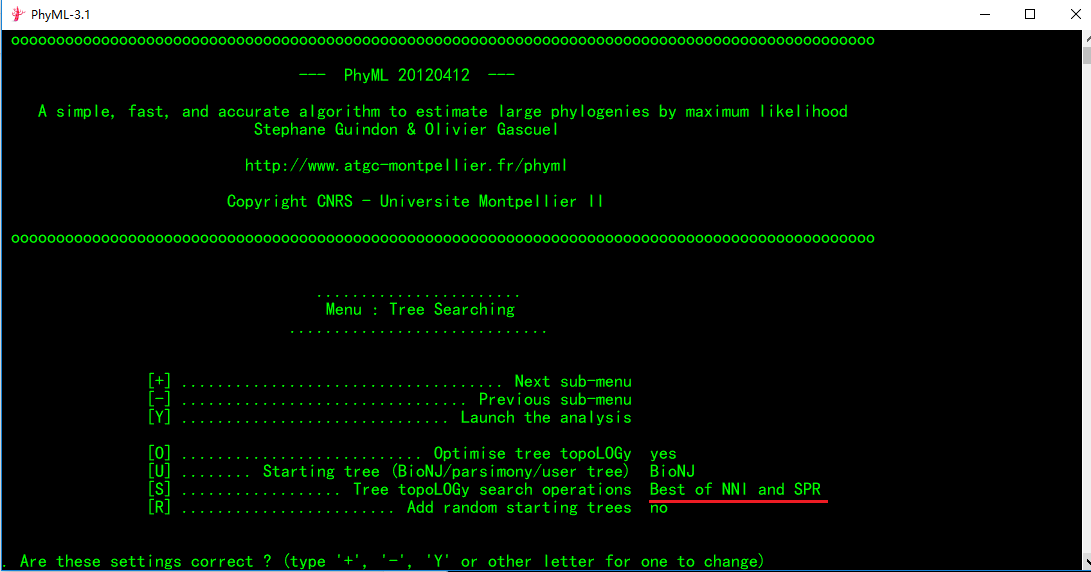
7.最后一页的参数设置
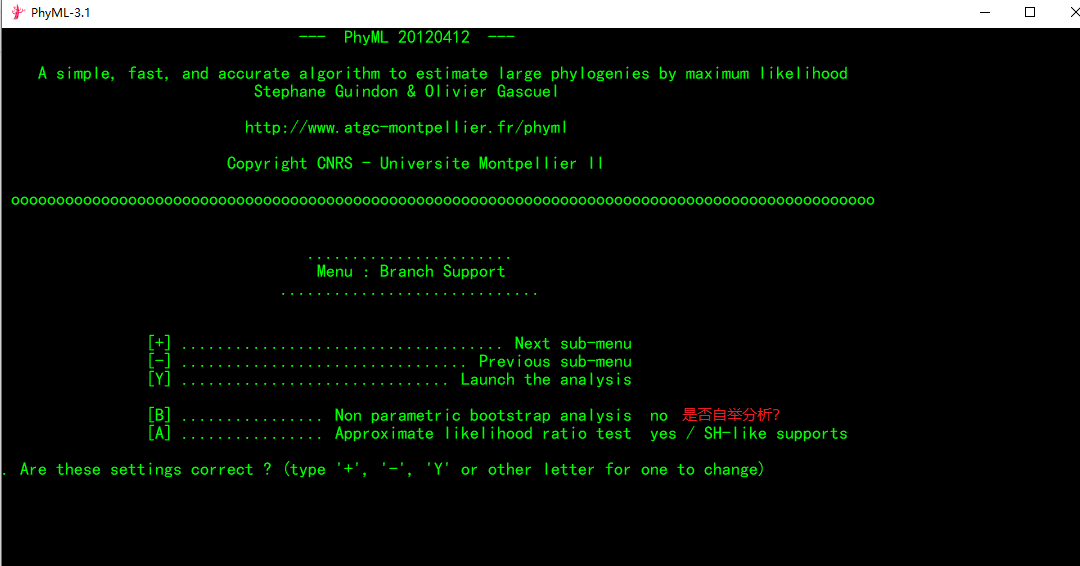
(1)光标输入处输入b,可设置分析的bootstrap值,一般设置为1000.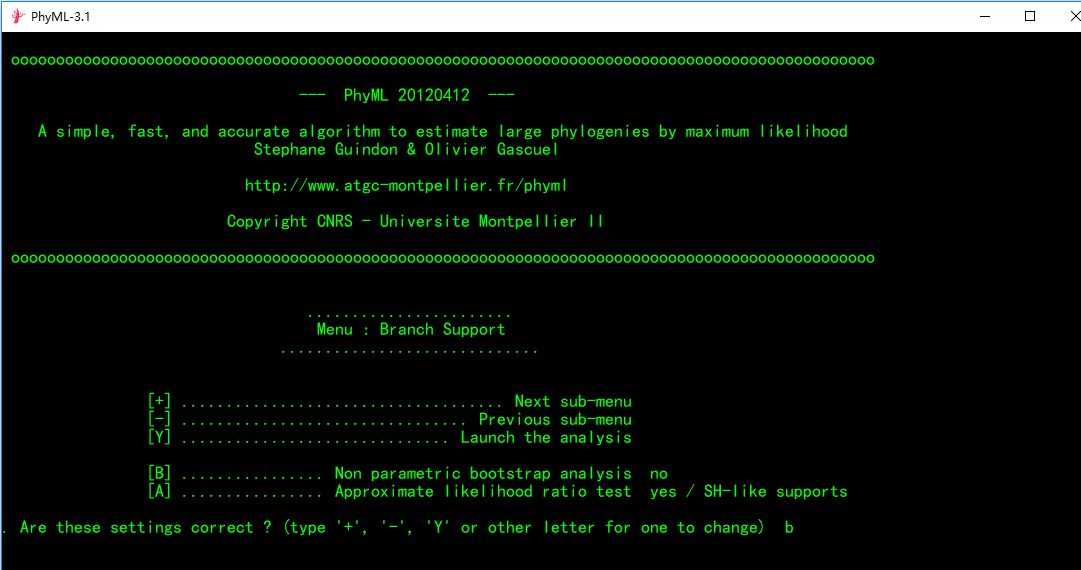
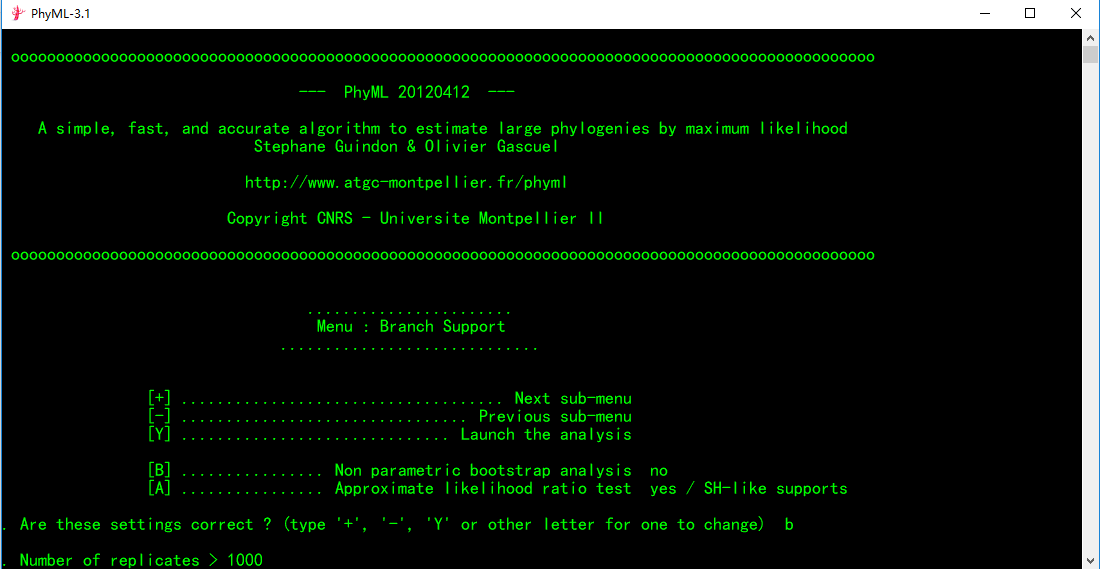
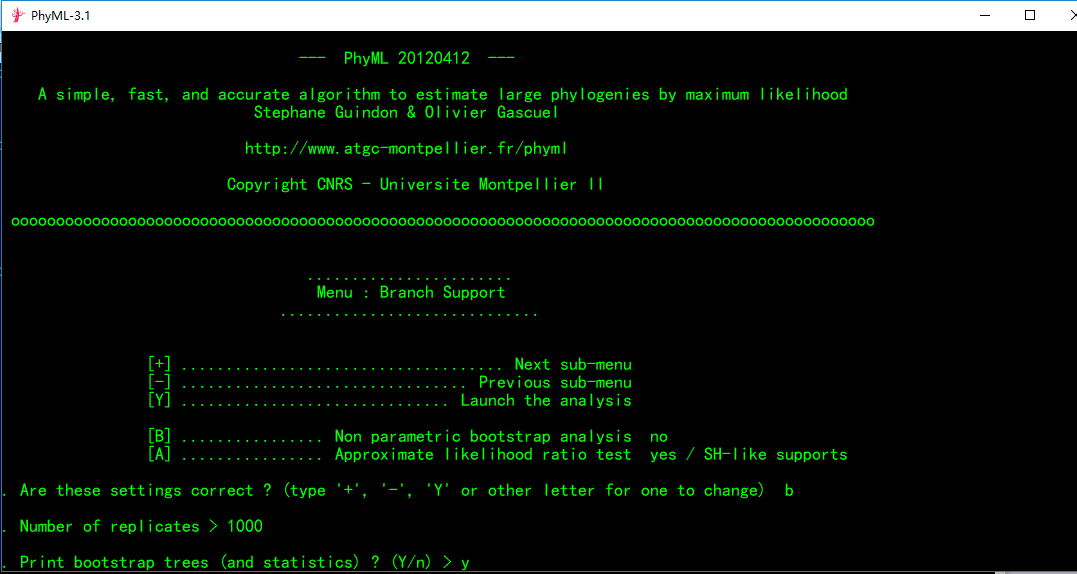
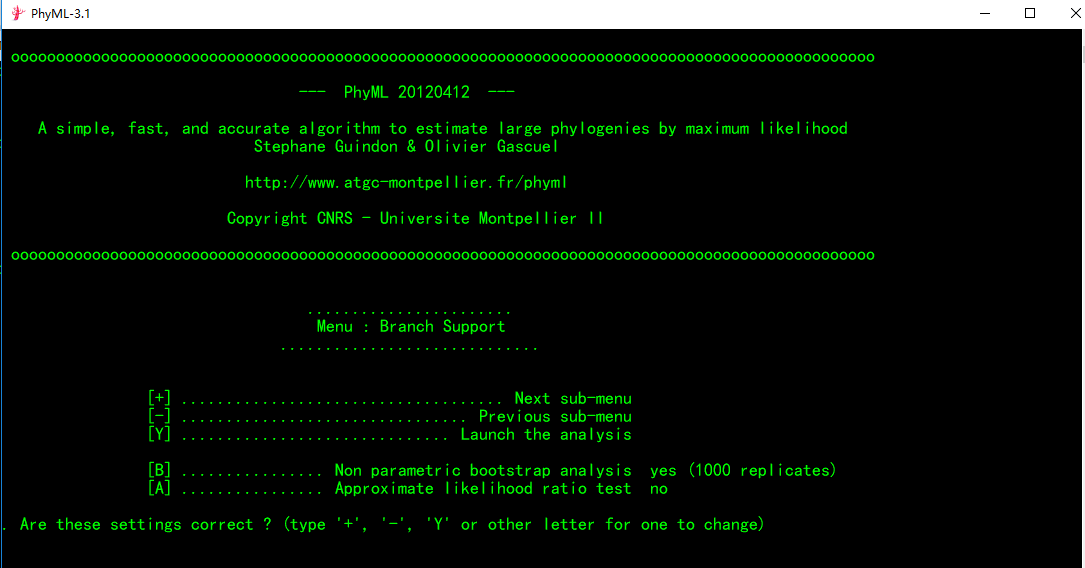
此时你会发现[A]选择自动设置为no,需要注意的是[B]和[A]两者只能选择一种。
如果你需要选择[A],可以通过在光标处输入a来进行调节。
8.开始运行
(1)所以参数设置完毕后,光标处输入y,开始构建ML树。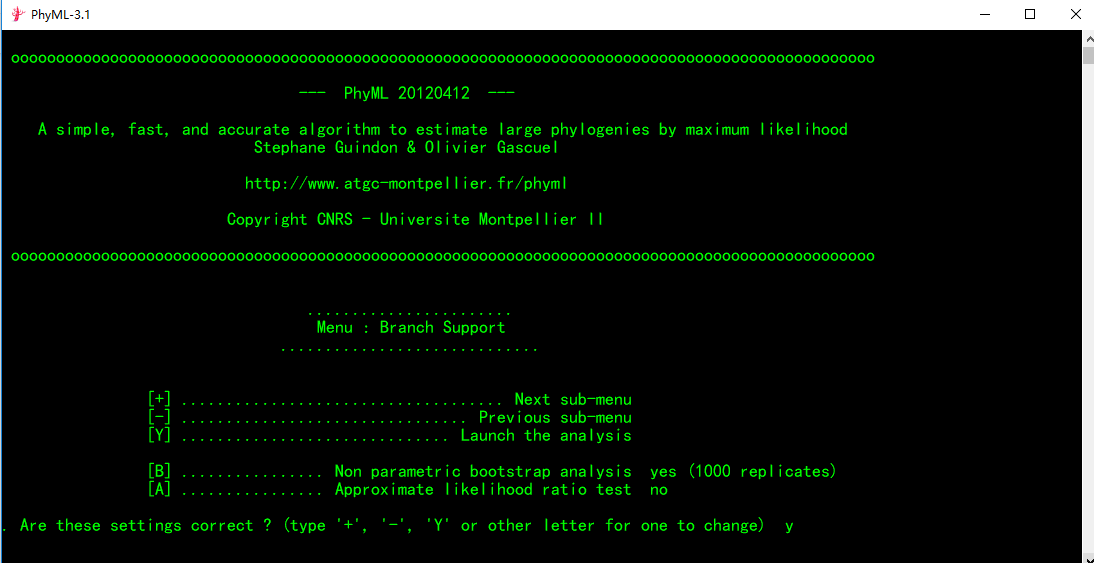
(2) 最终结果生成4个文件,其中cesi_zzj.phy_phyml_tree.txt是最终的最佳ML树,文件cesi_zzj.phy_phyml_stats.txt是运行的日志记录。
二.多线程运行PhyML3.1(适合服务器linux系统)
1.最简单的代码示例

需要注意的是,先要进入phyml执行程序所在的目录,再在命令终端敲代码。
上图这条代码意思是:以2个线程运行phyml,并设置自举值为100,其他所有参数选择默认值。
bootstrap replicates的数量必须是mpirun命令所需CPU数量的倍数。
2.各参数速查
注意:下面这些参数对于单线程版Phyml同样适用,意思是单线程版本的Phyml也可以不按照上面的一步步输入参数,可以像使用多线程版本一样用一条命令代码一次性搞定。
注:以下所有图片点击放大更清晰。






特别注意事项:PhyML版本3.0和版本3.1无法构建祖先序列, 即—ancestral这个参数选项无法用。 输出的树文件有自举值。
PhyML版本3.3可以构建祖先序列,但是输出的树文件没有自举值,这是该版本最大的bug。
以上笔者已经亲自调试过,希望大家不要再踩坑。
故推荐使用phyml3.1,软件压缩包见下:(windows版本+linux版本)
PhyML-3.1.zip
①服务器多线程运行phyml3.1调用命令示例:(前提是服务器有安装mpi程序)
$ mpirun -np 10 ./PhyML-3.1_linux64 -i /home/gong/桌面/zzj.phy -q -m GTR -b 1000 -a 0.4110 -s SPR —rand_start 5 —print_site_lnl —print_trace
②服务器单线程运行phyml3.1调用命令示例:
$ ./PhyML-3.1_linux64 -i /home/gong/桌面/zzj.phy -q -m GTR -b 1000 -a 0.4110 -s SPR —rand_start 5 —print_site_lnl —print_trace
这些参数的含义上面图片都有,不再复述,比如模型以及GAMMA的alpha值等取决于你的序列。
另外附上phyml版本3.3的linux版本包:
phyml3.3-master.zip
如果你想预测你的这些序列的祖先序列,这个版本还是用得到的。
转载请注明出处,如有错误欢迎您及时反馈,一起交流学习。