参考链接:降维算法(语雀-小西瓜)
数据降维的what&why
所谓降维,即用一组个数为的向量
来代表个数为
的向量
所包含的有用信息,其中
,通俗来讲,即将高维度下降至低维度;将高维数据下降为低维数据。
在机器学习领域中,我们对原始数据进行特征提取,有时会得到比较高位的特征向量,很多数据机的维度会高达成百乃至上千,例如经典的MNIST,其维度就是64。在这些向量所处的高维空间中,包含很多冗余和噪声。
但在实际应用中,我们所用到的有用信息却并不需要那么高的维度,而且每增加一维所需的样本个数呈指数级增长,这可能会直接带来极大的「维数灾难」;而数据降维就可以实现:
- 使得数据集更易使用
- 确保变量之间彼此独立
- 降低算法计算运算成本
- 去除噪音
我们希望通过降维的方式来寻找数据内部的特性,从而提升特征表达能力,降低训练复杂度。一旦我们能够正确处理这些信息,正确有效地进行降维,这将大大有助于减少计算量,进而提高机器运作效率。而数据降维,也常应用于文本处理、人脸识别、图片识别、自然语言处理等领域。
数据降维方法
往往高维空间的数据会出现分布稀疏的情况,所以在降维处理的过程中,我们通常会做一些数据删减,这些数据包括了冗余的数据、无效信息、重复表达内容等。因此,大部分经典降维技术也是基于这一内容而展开,其中降维方法又分为线性和非线性降维,非线性降维又分为基于核函数和基于特征值的方法。
线性降维方法
- PCA
- LDA、ICA LDA
- LFA
- LPP(LE 的线性表示)
非线性降维方法
- 基于核函数的非线性降维方法,核映射的基础概念可以参考支持向量机SVM一文,在此就不再累述哈
- KPCA 、KICA、KDA
- KPCA 、KICA、KDA
- 基于特征值的非线性降维方法(流型学习)
- ISOMAP、LLE、LE、LPP、LTSA、MVU
- ISOMAP、LLE、LE、LPP、LTSA、MVU
PCA主成分分析法
PCA算法虽然经典且较为常用,其不足之处也非常明显。它可以很好的解除线性相关,但是面对高阶相关性时,效果则较差;同时,PCA 实现的前提是假设数据各主特征是分布在正交方向上,因此对于在非正交方向上存在几个方差较大的方向,PCA 的效果也会大打折扣。
1 什么是主成分分析法
PCA( Principal Component Analysis),即主成分分析方法,是一种使用最广泛的数据降维算法(非监督的机器学习)方法
其最主要的用途在于“降维”,通过析取主成分显出的最大的个别差异,发现更便于人类理解的特征。也可以用来削减 回归分析和聚类分析中変量的数目。
2 为什么要做主成分分析
在很多场景中需要对多变量数据进行观测,在一定程度上增加了数据采集的工作量。更重要的是:多変量之间可能存 在相关性,从而增加了问题分析的复杂性。
如果对每个指标进行单独分析,其分析结果往往是孤立的,不能完全利用数据中的信息,因此盲目减少指标会损失很 多有用的信息,从而产生错误的结论。
因此需要找到一种合理的方法,在減少需要分析的指标同时,尽量减少原指标包含信息的损失,以达到对所收集数据 进行全面分析的目的。由于各变量之间存在一定的相关关系,因此可以考虑将关系紧密的变量变成尽可能少的新变 量,使这些新变量是两两不相关的,那么就可以用较少的综合指标分别代表存在于各个变量中的各类信息。主成分分 析与因子分析就属于这类降维算法
3 主成分分析法的思想
PCA的主要思想是将η维特征映射到k维上,这k维是全新的正交特征也被称为主成分,是在原有η维特征的基础上重 新构造出来的k维特征。
先假设用数据的两个特征画出散点图
如果我们只保留特征1或者只保留特征2。那么此时就有一个问题,留个哪个特征比较好呢?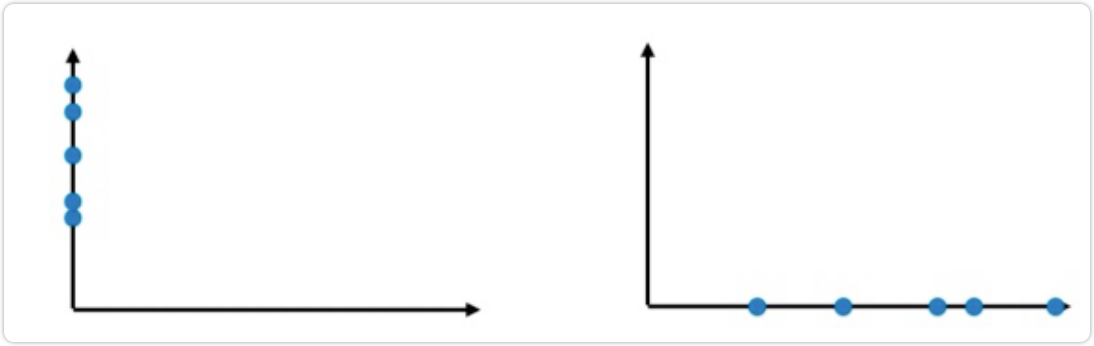
通过上面对两个特征的映射结果可以发现保留特征1(右面)比较好,因为保留特征1,当把所有的点映射到ⅹ轴上以 后,点和点之间的距离相对较大,也就是说,拥有更高的可区分度,同时还保留着部分映射之前的空间信息。
那么如果把点都映射到y轴上,发现点与点距离更近了,这不符合数据原来的空间分布。所以保留特征1相比保留特征 2更加合适,但是这是最好的方案吗?
将所有的点都映射到一根拟合的斜线上,从二维降到一维,整体和原样本的分布并没有多大的差距,点和点之间的距 离更大了,区分度也更加明显。
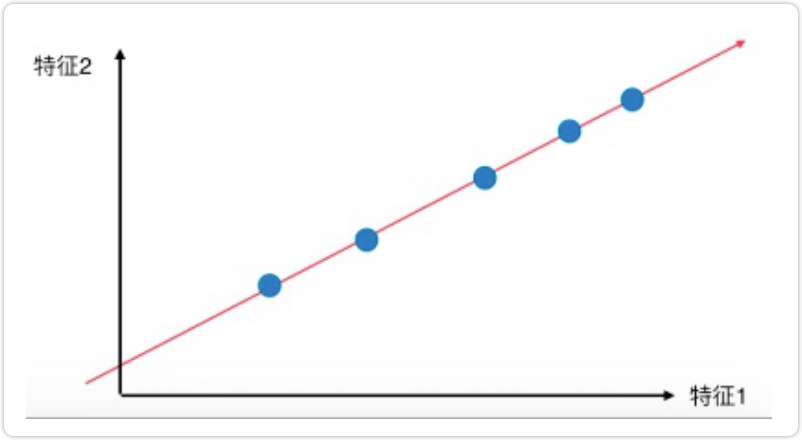
也就是说,我们要考虑的问题是
如何找到让样本同距最大的轴?
其中,一般我们会使用方差( Variance)来定义样本之间的间距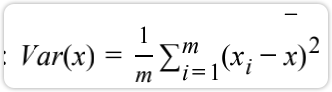
4 主成分分析法的步骤
对于如何找到一个轴,使得样本空间的所有点映射到这个轴的方差最大。
- 第一步:样本归0
将样本进行均值归0( demean),即所有样本将去样本的均值。样本的分布没有改变,只是将坐标轴进行了移动。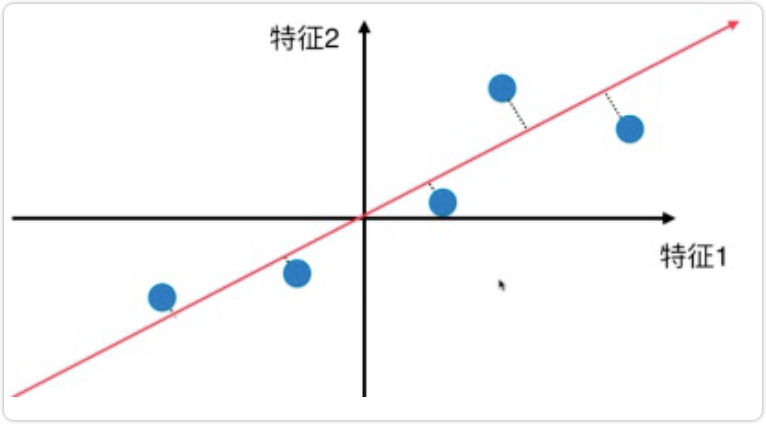
此时对于方差公式: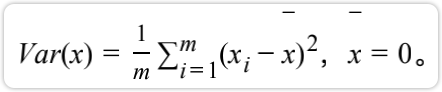 。其中此时计算过程就少ー一项,这就是为什么要进行样本均值归 0,可以化简的更加方便
。其中此时计算过程就少ー一项,这就是为什么要进行样本均值归 0,可以化简的更加方便
- 第二步:找到样本点映射后方差最大的单位向量w
求一个轴的方向W=(w1,w2)需要定义一个轴的方向W=(w1,w2),使得我们的样本,映射到w以后,使得X映射到w之后 的方差最大: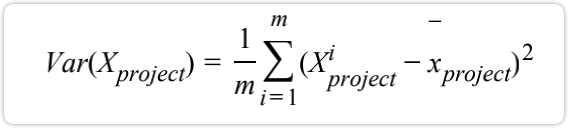
其实括号中的部分是一个向量,更加准确的描述应该是(向量的模):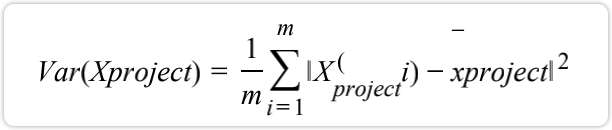
因为前面已经去均值,所以,这里只需下面的式子最大: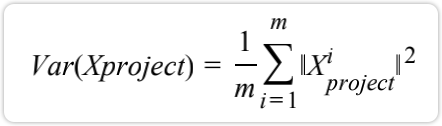
映射过程如下
红色的线是我们要找的方向w=(w1,w2);第行的样本点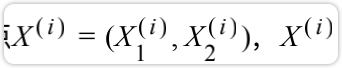 此时也是一个向量;映射到上做 垂线,交点的位置就是
此时也是一个向量;映射到上做 垂线,交点的位置就是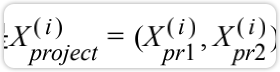 对应的点;真正要求的
对应的点;真正要求的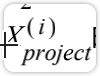 的模的平方,蓝色线段长度对应的平方
的模的平方,蓝色线段长度对应的平方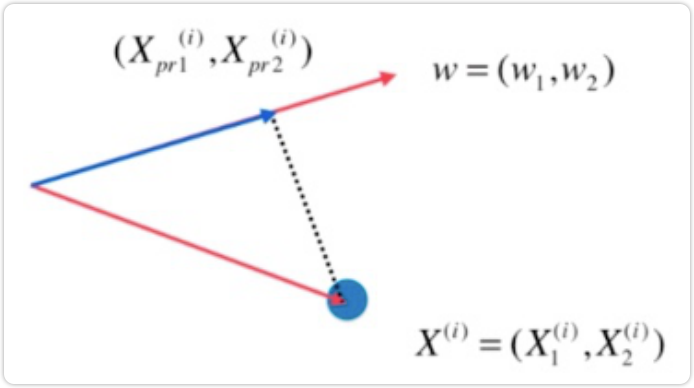
把一个向量映射到另ー个向量上,对应的映射长度是多少。实际上这种映射就是点乘: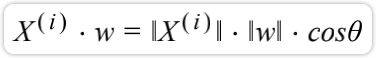
因为向量是我们要找的轴,是一个方向,因此使用方向向量就可以。因此长度为1: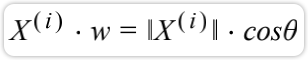
因此,在三角形中有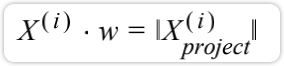
主成分分析法的目标是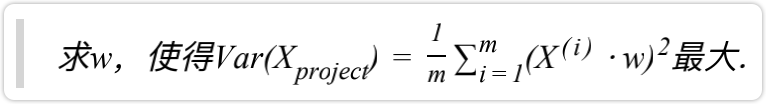
如果是n维数据,则有: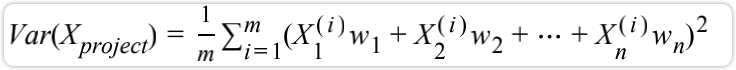
5 总结
主成分分析方法(PCA),是数据降维算法。将关系紧密的变量变成尽可能少的新变量,使这些新变量是两两不相关的,即用较少的综合指标分别代表存在于各个変量中的各类信息,达到数据降维的效果。
所用到的方法就是“映射”:将n维特征映射到k维上,这k维是全新的正交特征也被称为主成分,是在原有n维特征的 基础上重新构造出来的k维特征。我们要选择的就是让映射后样本间距最大的轴。
其过程分为两步:
- 样本归0
- 找到样本点映射后方差最大的单位向量
最后就能转为求目标函数的最优化问题: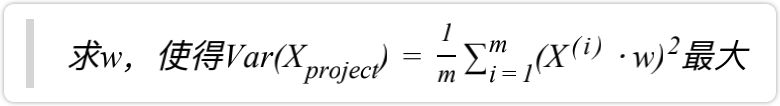
此时,我们就可以用搜索策略,使用梯度上升法来解決。
PCA降维算法实践
# PCA降维算法实践from __future__ import print_functionfrom sklearn import datasetsimport matplotlib.pyplot as pltimport numpy as npdef shuffle_data(X, y, seed=None):if seed:np.random.seed(seed)idx = np.arange(X.shape[0])np.random.shuffle(idx)return X[idx], y[idx]# 正规化数据集 Xdef normalize(X, axis=-1, p=2):lp_norm = np.atleast_1d(np.linalg.norm(X, p, axis))lp_norm[lp_norm == 0] = 1return X / np.expand_dims(lp_norm, axis)# 标准化数据集 Xdef standardize(X):X_std = np.zeros(X.shape)mean = X.mean(axis=0)std = X.std(axis=0)# 做除法运算时请永远记住分母不能等于 0 的情形# X_std = (X - X.mean(axis=0)) / X.std(axis=0)for col in range(np.shape(X)[1]):if std[col]:X_std[:, col] = (X_std[:, col] - mean[col]) / std[col]return X_std# 划分数据集为训练集和测试集def train_test_split(X, y, test_size=0.2, shuffle=True, seed=None):if shuffle:X, y = shuffle_data(X, y, seed)n_train_samples = int(X.shape[0] * (1 - test_size))x_train, x_test = X[:n_train_samples], X[n_train_samples:]y_train, y_test = y[:n_train_samples], y[n_train_samples:]return x_train, x_test, y_train, y_test# 计算矩阵 X 的协方差矩阵def calculate_covariance_matrix(X, Y=np.empty((0, 0))):if not Y.any():Y = Xn_samples = np.shape(X)[0]covariance_matrix = (1 / (n_samples - 1)) * (X - X.mean(axis=0)).T.dot(Y - Y.mean(axis=0))return np.array(covariance_matrix, dtype=float)# 计算数据集 X 每列的方差def calculate_variance(X):n_samples = np.shape(X)[0]variance = (1 / n_samples) * np.diag((X - X.mean(axis=0)).T.dot(X - X.mean(axis=0)))return variance# 计算数据集 X 每列的标准差def calculate_std_dev(X):std_dev = np.sqrt(calculate_variance(X))return std_dev# 计算相关系数矩阵def calculate_correlation_matrix(X, Y=np.empty([0])):# 先计算协方差矩阵covariance_matrix = calculate_covariance_matrix(X, Y)# 计算 X, Y 的标准差std_dev_X = np.expand_dims(calculate_std_dev(X), 1)std_dev_y = np.expand_dims(calculate_std_dev(Y), 1)correlation_matrix = np.divide(covariance_matrix, std_dev_X.dot(std_dev_y.T))return np.array(correlation_matrix, dtype=float)class PCA:"""主成份分析算法 PCA,非监督学习算法."""def __init__(self):self.eigen_values = Noneself.eigen_vectors = Noneself.k = 2def transform(self, X):"""将原始数据集 X 通过 PCA 进行降维"""covariance = calculate_covariance_matrix(X)# 求解特征值和特征向量self.eigen_values, self.eigen_vectors = np.linalg.eig(covariance)# 将特征值从大到小进行排序,注意特征向量是按列排的,即 self.eigen_vectors 第 k 列是 self.eigen_values 中第 k 个特征值对应的特征向量idx = self.eigen_values.argsort()[::-1]eigenvalues = self.eigen_values[idx][:self.k]eigenvectors = self.eigen_vectors[:, idx][:, :self.k]# 将原始数据集 X 映射到低维空间X_transformed = X.dot(eigenvectors)return X_transformeddef main():# Load the datasetdata = datasets.load_iris()X = data.datay = data.target# 将数据集 X 映射到低维空间X_trans = PCA().transform(X)x1 = X_trans[:, 0]x2 = X_trans[:, 1]cmap = plt.get_cmap('viridis')colors = [cmap(i) for i in np.linspace(0, 1, len(np.unique(y)))]class_distr = []# Plot the different class distributionsfor i, l in enumerate(np.unique(y)):_x1 = x1[y == l]_x2 = x2[y == l]_y = y[y == l]class_distr.append(plt.scatter(_x1, _x2, color=colors[i]))# Add a legendplt.legend(class_distr, y, loc=1)# Axis labelsplt.xlabel('Principal Component 1')plt.ylabel('Principal Component 2')plt.show()if __name__ == "__main__":main()

