说到本体论,生信的同学估计第一个想到的就是基因本体论 ( Gene Ontology, GO ) 了,而基因本体论其实也是开放生物医学系统注释(Open Biomedical Ontologies (OBO))下的一个分类项目。今天来说另外一个生物科学数据分析和数据管理本体论——EDAM - Bioscientific data analysis ontology。
背景知识
以下背景知识主要摘录于隔壁老湿在简书的《学习笔记:本体论与生物医学》一文。
本体论
本体论(Ontology)是个哲学的概念,作为一门学问起源于对万物本源的追问,最早这个词在17世纪诞生,从哲学意义上,本体论关注的是“存在”,即世界在本质上有什么样的东西存在,或者世界存在哪些类别的实体。
作者:隔壁老湿,链接:https://www.jianshu.com/p/b4d8922c9c42,来源:简书
本体论与信息科学
在人工智能领域,经历了 60 年代通用问题求解方法研究的困境,学者们开始研究通过专门领域的知识表达来支持自动推理,以 Dendral 为代表的知识库系统的成功确立了知识在解决人工智能问题方面的重要地位。为了减少构建知识库的代价,避免每次从头开始,有必要考虑知识的复用问题,通过复用,系统开发者可以在已有知识基础上更加专注于特定领域的知识构建,且新系统可以利用复用知识与现存其他系统进行交互。这样,描述性的知识、问题解决方法及推理服务都可以在系统中实现共享,从而可以方便的构建更大,更好的知识库。还为了解决数据库集成中专门化和不一致特征导致的实际问题。解决这些问题都需要对某个领域进行通用概念上的描述,就必须回答这样的问题:对于某个领域,本质上有些什么样的对象、过程、属性和关系?什么是一项事务、一个人及一个组织?它们之间的相互依赖关系如何?这就是本体论研究的内容。
AI 逻辑学家在 1980 年提出:以逻辑概念为基础的智能系统必须列出所有存在的事物,并构建一个本体描述我们的世界。
1993 年,斯坦福大学知识系统实验室的 Gruber 给出了第一个在信息科学领域广泛接受的 Ontology 定义: an ontology is an explicit specification of a conceptualization,概念化是从特定目的的出发对所表达的世界所进行的一种抽象的、简化的观察。本体论是对某一概念化所做的一种显式的解释说明。本体论是一个逻辑理论,用来说明一个正规(formal)词汇表的预定含义。因此本体是语言相关的,而概念化则是语言无关的,认清这一点对本体集成、融合和转换都非常重要。另外在上述定义中,概念化是比本体论(仅限于信息科学中)更为广泛的概念,前者更接近领域的事实和哲学上的本体论。
由于 Web 技术为全球信息共享提供了便捷手段,以共享为特征的本体论与 Web 技术结合是必然趋势。在此背景下,基于 Web 标准的本体描述语言成为本体论研究和运用的热点。在标准方面,由 W3C 主持制订的 RDF 和 RDF Schema 是建立在 XML 语法上,以语义网为理论基础,对信息资源进行语义描述的的语言规范,RDF 采用资源(Resource)、属性(Properties)、和声明(Statement)等三元组来描述事物。RDF schema 则做了进一步扩展。因此,RDF 成为一个能对本体进行初步描述的标准语言。
作者:隔壁老湿,链接:https://www.jianshu.com/p/b4d8922c9c42,来源:简书
本体论与语义 WEB
本体论在 WEP上的应用导致了语义 WEP 的诞生,其目的是解决 Web 上信息共享时的语义问题,语义 WEP 被看做是当前本体论从学术研究走向实际应用的最重要的构想和研究计划。
从技术上讲,语义 Web 要提供足够而又合适的语义描述机制,然而,从整个应用构想来看,语义 Web 要实现的是信息在知识级的共享和语义上的互操作性,这需要不同系统间有一个语义上的“共同理解”才行。于是,本体论自然地成为指导语义 Web 发展的理论基础。
作者:隔壁老湿,链接:https://www.jianshu.com/p/b4d8922c9c42,来源:简书
本体论与生物医学
复杂的生物学知识常常是以一种相互关联且有层级(hierarchical)的方式存在,需要有某种途径和分子数据(例如测序产生的数据)进行整合,需要的领域很多,比如怎样在解剖,信号传导途径,和表型之间的知识和数据建立联系。“生物本体论”(Bio-ontologies)的方法是解决这个问题的一种方式。用本体论的方法来规范特定生物领域的知识,用基于特定规则的概念来来界定和描述概念之间的关系,基于本体论“结构化”后的知识就能和分子数据建立联系。以“肱”这个概念为例,来解释本体论就是,需要一系列“声明”(assertions)来定义“肱”的内涵和外延,比如它是手臂的一部分,含成骨细胞,有肌肉的粘附点,可以是骨头等。在生物本体论的定义里,它不仅仅被定义成一系列关系的集合,它也有一个唯一的识别代码(比如 GO:0019505)。
以生物本体论领域应用最广的 GO(基因本体论,Gene Ontology)为例,目前它已经积累了 16500 个概念,GO连接了 12000 个基因来自 20 种模式生物,包含动物,植物,真菌,细菌和病毒,每个基因已经用唯一的 GOID 标记。就每一个基因而言,会对其功能(function)、参与的生物过程(biological process)和细胞定位(celluar localization)进行注释。
OBI(生物医学研究本体 Ontology for biomedical investigations)是本体论在生物医学研究的又一个例子。这是一个生物医学领域本体论学者自发形成的组织,其宗旨是构建和描述生命科学和临床研究相关的本体(An integrated ontology for the description of life-science and clinical investigations)。它涉及用本体论的方法对研究设计、方案、设备、研究数据、数据分析方法等进行注释。它源于功能基因组研究本体组织(FuGO),它们之间涉及的共同 Term 是共享且一致的。
作者:隔壁老湿,链接:https://www.jianshu.com/p/b4d8922c9c42,来源:简书
EDAM
EDAM 是生物医学领域研究除 GO、OBI 以外的又一个例子,个人最开始是从 Galaxy Project 了解得到,EDAM 在 Galaxy 项目中主要用于解决海量生物医学工具在 Galaxy 平台面板中分类不统一,用户难以查找的难题而被纳入使用。最早在 Galaxy Release 21.09 版本中上线(Galaxy Release (v 21.09),新的工具面板视图)。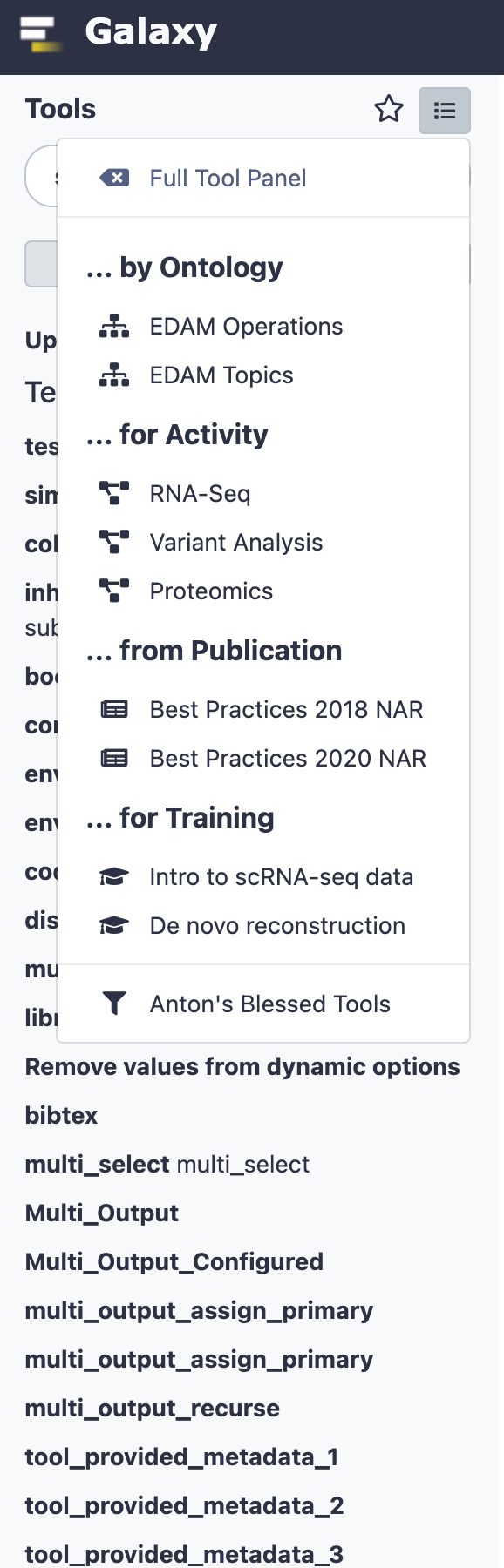
简介
EDAM - Ontology of bioscientific data analysis and data management,是一个全面的本体论,包含了在生物科学数据分析和数据管理(包括计算生物学、生物信息学和生物图像信息学)中广泛存在的既定的、熟悉的概念。EDAM 包括与生命科学中的数据分析和数据管理相关的主题(topics)、操作(operations)、数据类型(types of data)和数据标识符(data identifiers)以及数据格式(data formats)。EDAM 提供了一组包含首选术语和同义词、相关术语、定义和其他信息的概念,组织成一个简单直观的层次结构,以方便使用(见下图)。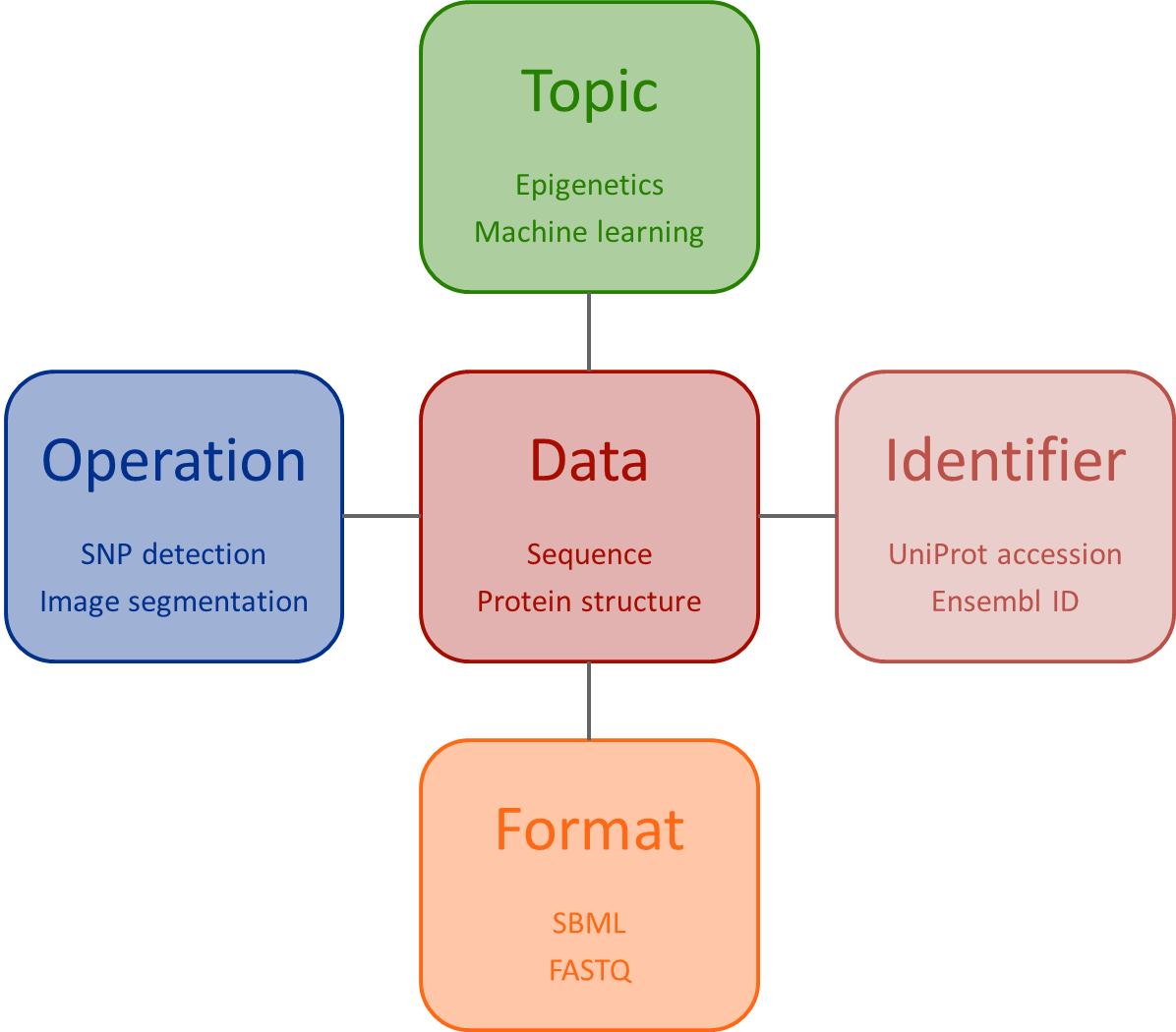
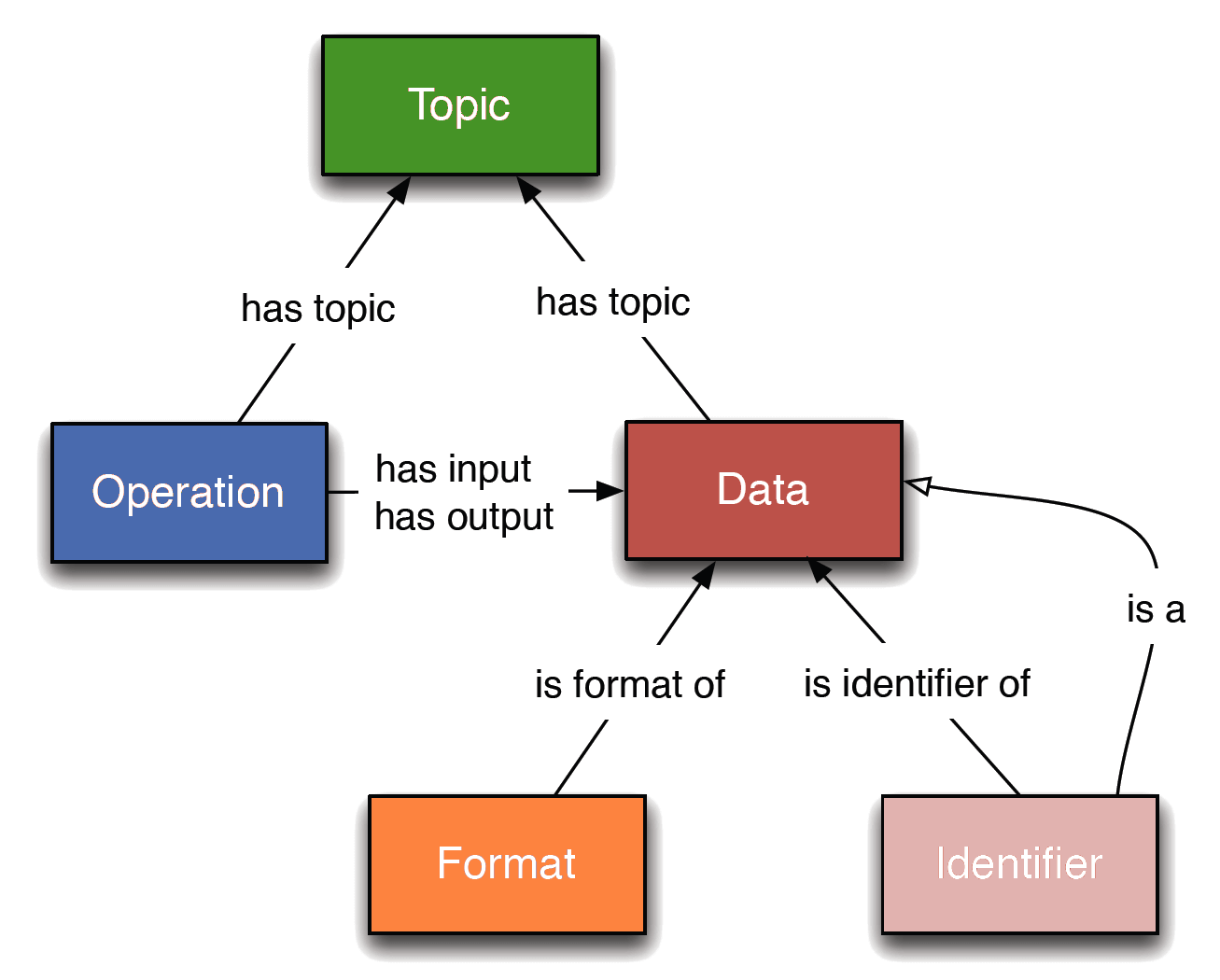
Bio.Tools
EDAM 目前包含了一下几个子项目,Galaxy Project 的工具面板正好是使用了 Bio.Tools 这一本体库。
- Bio.Tools
- BioSimulations
- BioSimulators
- BioXSD
- CEDAR FAIR Funder Form
- Common Workflow Language
- omputational Biology Institute of Montpellier
- EMBOSS
- eSysbio
- runBioSimulations
- SEQwiki
Bio.Tools 是一个致力于提供软件和数据库的全面登记册,以便来自生物和生物医学科学领域的研究人员寻找、理解、利用和引用他们日常工作所需的资源(software and databases)的服务。
Everything from simple command-line tools and online services, through to databases and complex, multi-functional analysis workflows is included. Resources are described in a rigorous semantics and syntax, providing end-users with the convenience of concise, consistent and therefore comparable information.
Each bio.tools entry is assigned a human-readable, unique identifier based on the resource name, e.g. biotools:signalp. These identifiers provide a persistent reference to our “Tool Cards” of essential information, as well as a means to trace resources and integrate bio.tools data with other resources.
每个 bio.tools 条目都会根据资源名称分配一个人类可读的唯一标识符,例如 biotools: signalp(或者 biotools: ssamtools)。这些标识符提供了对基本信息的“工具卡”的持久引用,以及跟踪资源并将 bio.tools 数据与其他资源集成的手段。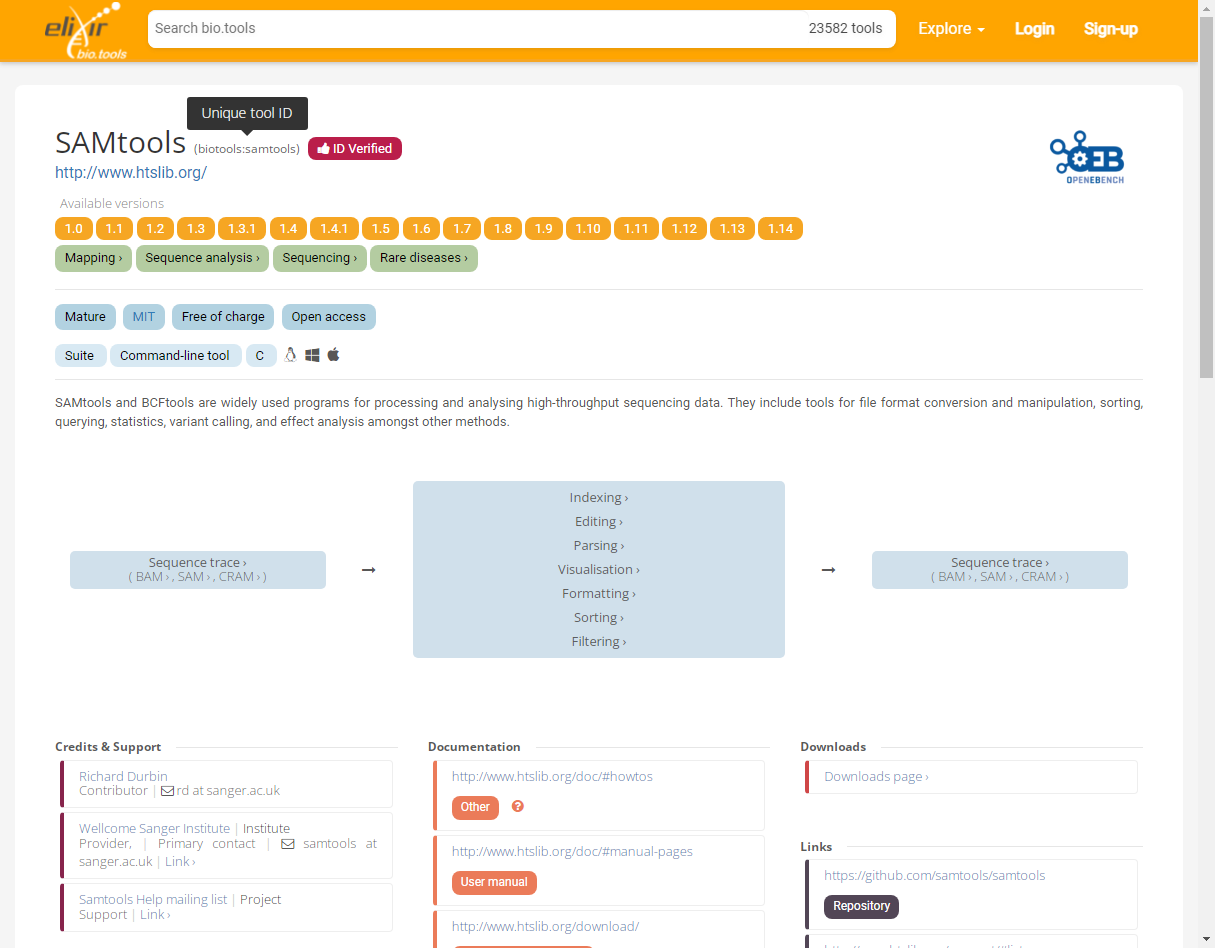
All the bio.tools data and technical components are available under open license and we warmly welcome you to get involved.
所有的生物工具数据和技术组件都是开放许可的,此外,Bio.Tools 是由 ELIXIR 通过 ELIXIR-exgelerate 补助金支持的,该补助金由欧盟的 European Union Horizon 2020 项目根据补助金协议 676559 资助。
总结
简单总结一下,目前对 DEAM 了解不多,Bio.Tools 也只还在探索中,但个人的感觉这其实就是一个生物医学软件与数据库的标准化数据库,把那些杂乱无章的软件统计分类管理,结构化也好层次化也好,最起码能一定程度降低对海量软件和数据库的搜索成本,可以有助于更容易地找到所需的工具。
Galaxy Project 是一个相当活跃的生物医学开源项目,它的一些标准一些思想是值得借鉴的,项目中对于最新技术的考量和接纳整合,让它从一个生物领域的项目不断扩展到其他领域,如农业、制药。随着开源技术的兴起,希望能遇到更多对 Galaxy 也感兴趣的人,一起交流分享。

