单分类问题

Sigmoid():将连续分布的值映射成“是”和“否”的回答
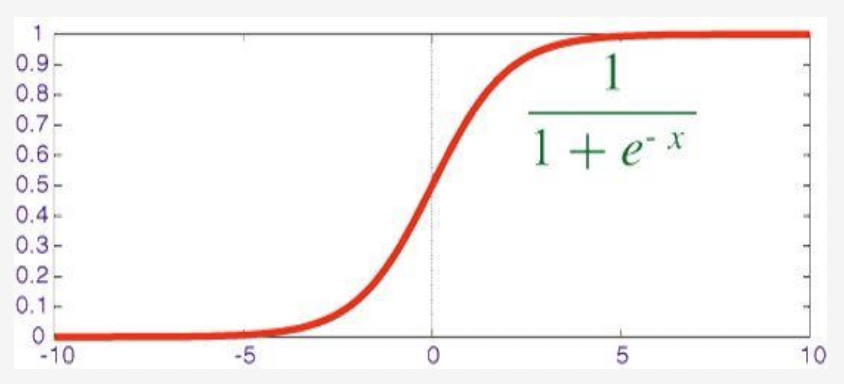
- 损失函数:使用
交叉熵损失函数。交叉熵刻画的是实际输出(概率)与期望输出(概率)的距离,也就是交叉熵的值越小,两个概率分布就越接近。

在keras里,我们使用 binary_crossentropy 来计算二元交叉熵。
数据预处理
- 独热编码
x.loc[:, 'Embarked_S'] = (x.Embarked == 'S').astype('int')x.loc[:, 'Embarked_C'] = (x.Embarked == 'C').astype('int')x.loc[:, 'Embarked_Q'] = (x.Embarked == 'Q').astype('int')del x['Embarked'] # 删除原有Embarked列x
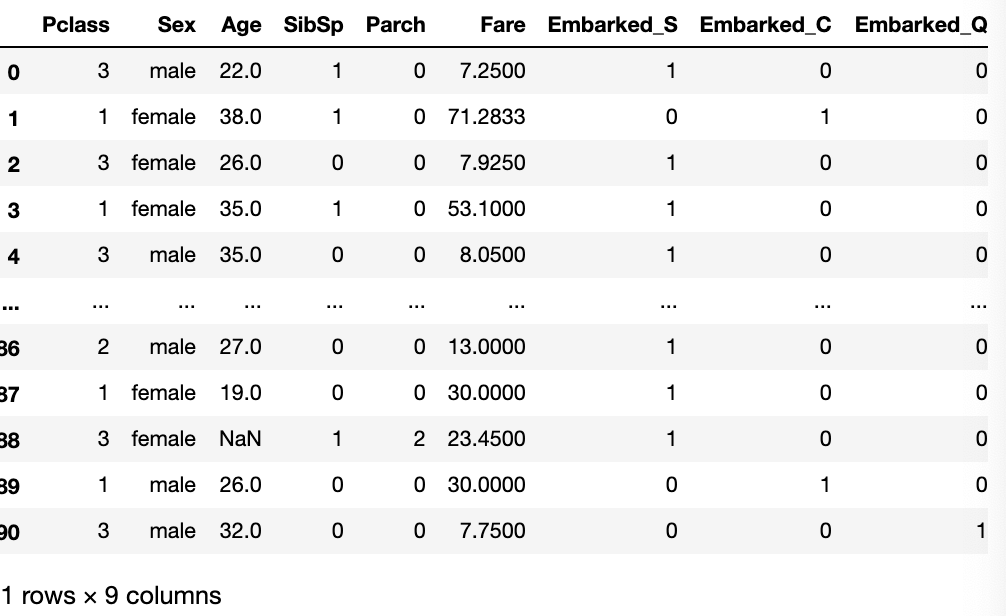
- 独热编码
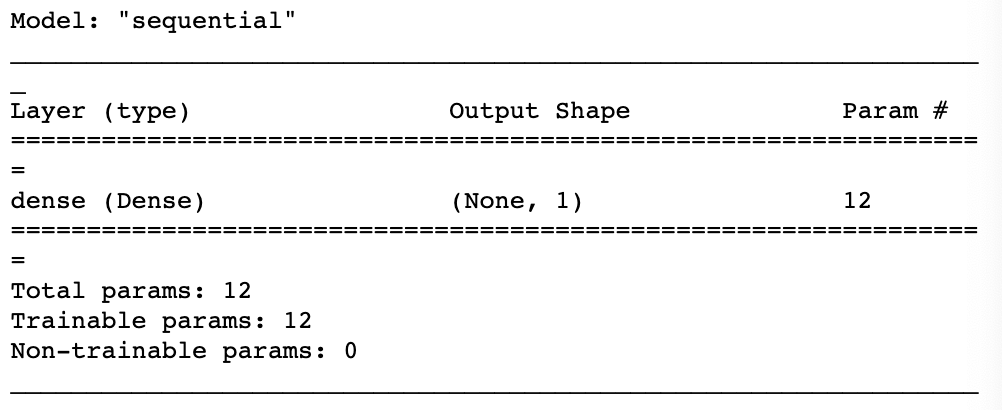
建模
model = keras.Sequential() # 初始化顺序模型from keras import layersmodel.add(layers.Dense(1, input_dim=11, activation='sigmoid'))#y_pre = (w1*x1 + w2*x2 + ... + w11*x11 + b)#sigmoid(y_pre) 变成概率值model.summary()
编译模型(给模型定义优化算法,以及优化的目标loss )
# 编译模型:# 优化函数:adam内置优化算法# 损失函数:二元交叉熵# 度量值:acc输出正确率model.compile(optimizer='adam',loss='binary_crossentropy',metrics=['acc'])
训练模型
# 训练模型history = model.fit(x, y, epochs=300)
测试模型
# 训练中保留的值history.history.keys()

import matplotlib.pyplot as plt%matplotlib inlineplt.plot(range(300), history.history.get('loss'))
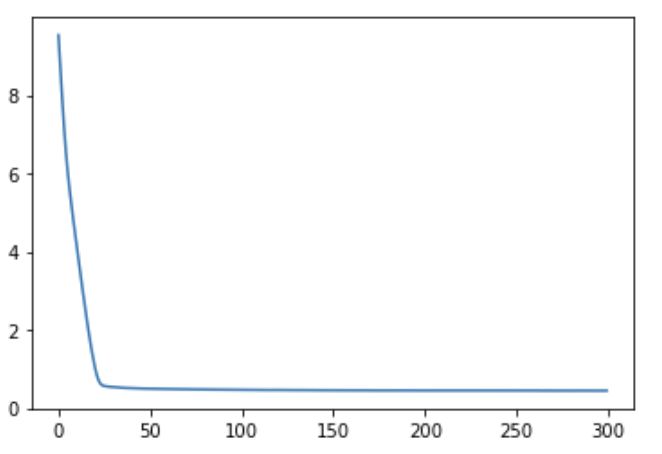
plt.plot(range(300), history.history.get('acc'))

多分类问题



- 损失函数:使用
softmax交叉熵。
在keras里,对于多分类问题我们使用categorical_crossentropy(独热编码) 和 sparse_categorical_crossentropy(顺序编码)
数据预处理
独热编码
data.Species.unique()
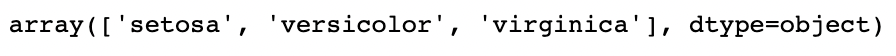
# pd.get_dummies()内置函数进行 独热编码data = data.join(pd.get_dummies(data.Species))del data['Species']data
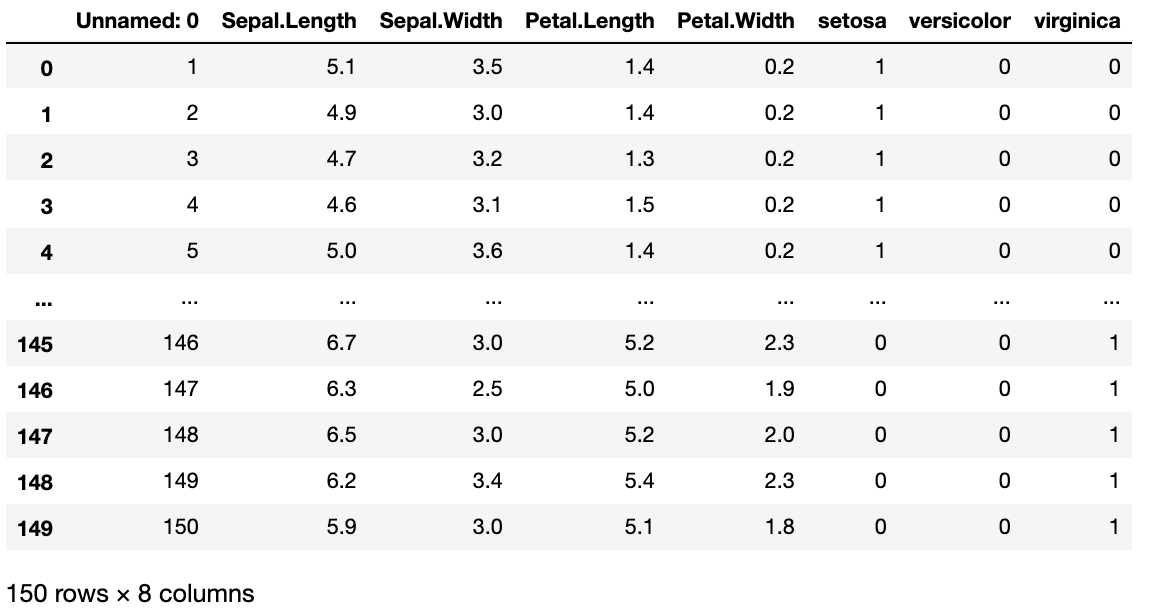
# 将data乱序,前面的同种花的数据都排在一起不利于训练index = np.random.permutation(len(data))data = data.iloc[index]
顺序编码
data.Species.unique()
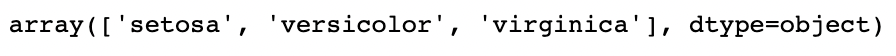
spc_dic = {'setosa': 0, 'versicolor': 1, 'virginica': 2}data['Species'] = data.Species.map(spc_dic)data
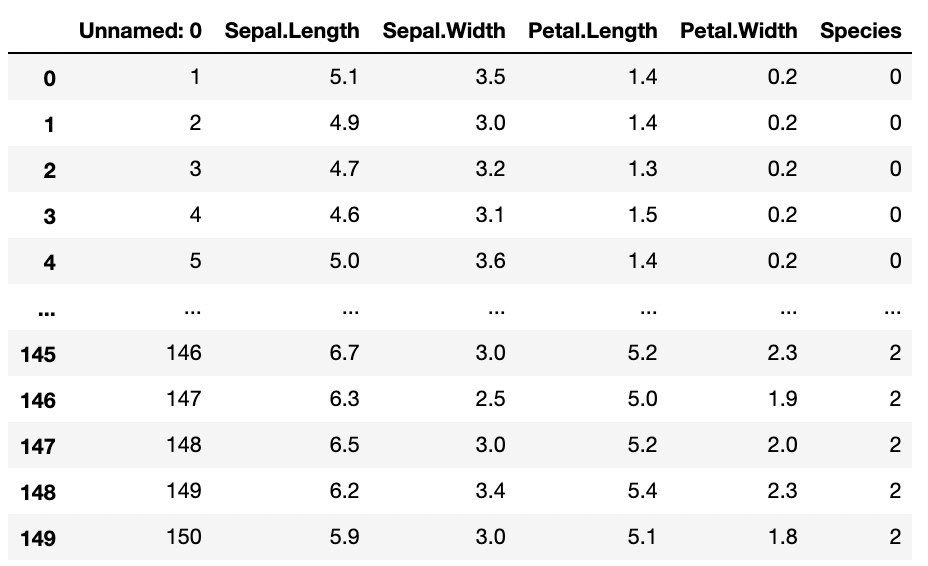
建模
# 建立模型model1 = keras.Sequential()# 添加Dense层:# 输出维度:3# 输入维度:4# 激活函数:sortmax多分类,将输出值变为概率值model1.add(layers.Dense(3, input_dim=4, activation='softmax'))# 注意这里的参数是15个,思考一下!model1.summary()

编译模型
独热编码
# 编译模型# 优化函数:adam内置优化算法# 损失函数:categorical_crossentropy(独热编码适用),来计算softmax交叉熵# 度量值:acc输出正确率model1.compile(optimizer='adam',loss='categorical_crossentropy',metrics=['acc'])
顺序编码
# 编译模型# 优化函数:adam内置优化算法# 损失函数:sparse_categorical_crossentropy(顺序编码适用),来计算softmax交叉熵# 度量值:acc输出正确率model.compile(optimizer='adam',loss='sparse_categorical_crossentropy',metrics=['acc'])
训练模型
# 训练模型history = model1.fit(x, y, epochs=100)

