调研进展
论文:《Deep learning for molecular design - a review of the state of the art》 2019年发表
分子表示
分子表示可以大体分为基于3D几何表示以及基于2D图表示,前者没有较好的表示方法,主要由于坐标对分子的平移、旋转和原子索引的排列不是不变的。
目前的方法都是基于2D图,最流行的就是把分子表示成SMILES,简单来说SMILES就是通过深搜把分子图转化成字符串,大多数模型处理的都是分子的SMILES表示形式。还有别的比较奇妙的办法,比如把graph表示成灰度图。tensor表示19年的综述里只提到了one-hot结合邻接矩阵这种表示(或许可以结合GNN提取的图表示进行辅助表示吗)。当然还有直接对图进行操作的。对小数据集可以用fingerprint。总之最流行的还是SMILES,以及直接对graph的操作也很promising。
模型
首先明确molecular design是一个generate的过程。作者总结了截止19年的模型采用的框架,总体来看包括RNN,VAE和GAN,其中还有RL的使用,在generator部分起到fine tune的作用或直接用pure RL来生成。
最常见的三种框架: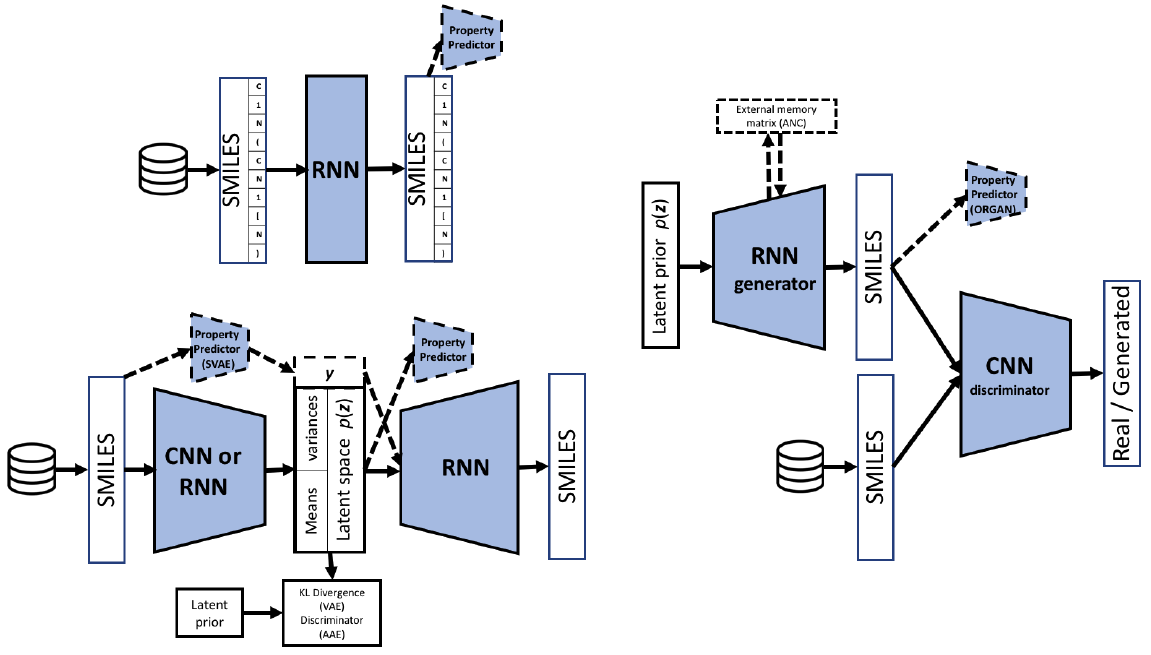
左上是直接利用RNN+RL,可以加入property信息的监督;左下是VAE的结构,CNN或RNN作为encoder,RNN作为decoder进行生成,在latent space部分同样可以加入property信息的监督;右边是GAN的结构,RNN作为generator,CNN作为discriminator。所有在用RNN做生成的部分都用RL进行fine tuning,奖励函数的设计是至关重要的。此外还有加入判别器的改进版VAE,AAE。
Metrics
metric方面可以参考已有的生成模型的metrics,比如inception score,目前也有针对分子进行评估的方法。

