Multidimensional and Multiresolution Ensemble Networks for Brain Tumor Segmentation 用于脑肿瘤分割的多维多分辨率集成网络 被引:3 脑肿瘤相关的概念
简概
WHY?
MRI分割脑肿瘤仍是一项艰巨的任务。
开发针对脑肿瘤的自动化,半自动化和交互式分割方法具有巨大的临床意义
WHAT?
我们建议对多分辨率和多维模型的输出进行集成以获得鲁棒的肿瘤分割。
- 利用现成的模型架构(DensNET-169,SERESNEXT-101和SENet-154)使用2D输入进行分割。
- 还实现了二维和三维残差起始Densenet(RID)网络,以基于补丁的输入(64x64和64x64x64)执行肿瘤分割。
HOW?
数据集:
Brats 2019数据集
网络架构:
训练了几种模型来产生鲁棒的肿瘤子成分分割。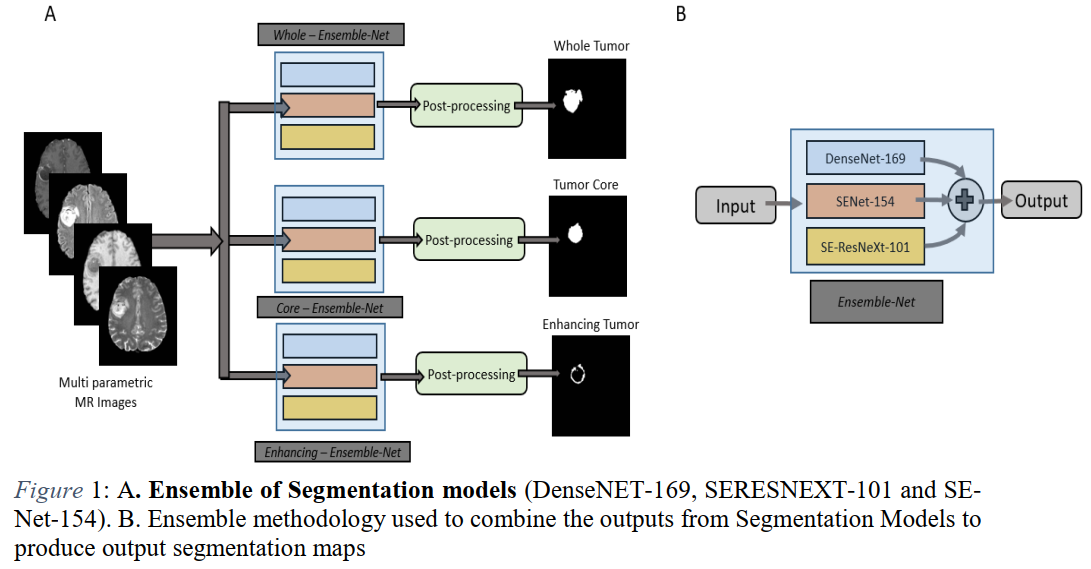

RESULT?
BRATS 2019验证数据集的初步结果证明了该方法可以实现出色的性能
FUTURE?
no
全文翻译
摘要
在这项工作中,我们开发了具有多分辨率输入的多个2D和3D分割模型,以分割脑肿瘤成分,然后对其进行组合以获得鲁棒的分割图。 这减少了过度拟合,并导致了更通用的模型。 来自BRATS 2019挑战赛的335个受试者的多参数MR图像用于训练模型。 此外,我们测试了一种经典的机器学习算法(xgboost),该算法具有从分割图提取的特征以对受试者的生存范围进行分类。BRATS 2019验证数据集的初步结果证明了该方法可以实现出色的性能,其DICE得分为0.898、0.784,全肿瘤为0.779,肿瘤核心为增强肿瘤,预测生存率为34.5%。
1简介
脑肿瘤占所有原发性中枢神经系统肿瘤的85%至90%。 原发性脑肿瘤的最常见类型是神经胶质瘤,根据其侵略性,它们可进一步分为高等级胶质瘤(HGG)和低等级胶质瘤(LGG)。 磁共振成像(MRI)是胶质瘤诊断和临床治疗中广泛使用的技术。 尽管是用于描述肿瘤和治疗计划的标准成像方式,但由于脑肿瘤形状,大小,位置的高度变化,特别是由于肿瘤区域相对于周围正常组织的细微强度变化,使用MRI分割脑肿瘤仍然是一项艰巨的任务。因此,手动进行肿瘤轮廓检查既费时,又会导致观察者之间和观察者内部的差异很大。 半自动或全自动脑肿瘤分割方法可以规避这种可变性,以实现更好的患者管理(Zhuge,Krauze等人2017)。 结果,开发针对脑肿瘤的自动化,半自动化和交互式分割方法具有巨大的临床意义,但是,这仍然具有很高的挑战性。 高效的深度学习算法可将脑肿瘤细分为其子组件,有助于对个别患者进行早期临床诊断,治疗和随访(Saouli,Akil等人2018)。
多模态脑肿瘤分割基准(BRATS)数据集通过将具有已知地面真相分割的独特脑肿瘤数据集外包出去,从而提供了一个通用平台,由专家手动完成(Menze,Jakab等,2014)。 在BRATS提供的这个独特平台上开发了几种高级深度学习算法,并针对通用数据集进行了基准测试,从而可以对它们进行一般比较。 基于CNN的方法在学习复杂功能的层次结构方面显示出优势,并且在最近的BRATS挑战中表现最佳。 基于U-net(Ronneberger,Fischer等人,2015)的网络架构已用于分割复杂的脑肿瘤结构。 Pereira等。 分别针对高品位和低品位胶质瘤开发了具有两种CNN架构的2D CNN方法,并在后处理步骤中组合了输出(Pereira,Pinto等人2016)。 Havaei等。 他开发了具有两个路径的多分辨率级联CNN架构,每个路径采用不同的2D斑块大小,并以四个MR序列作为通道(Havaei,Davy等人2017)。 BRATS 2018表现最好的人开发了3D解码器编码器样式的CNN架构,该架构具有层间跳过连接以分割肿瘤(Myronenko 2018)。 除解码器部分外,还包括变体自动编码器(VAE),以向模型添加重建损失。
在这项研究中,我们建议对多分辨率和多维模型的输出进行集成以获得鲁棒的肿瘤分割。 我们利用现成的模型架构(DensNET-169,SERESNEXT-101和SENet-154)使用2D输入进行分割。 我们还实现了二维和三维残差起始Densenet(RID)网络,以基于补丁的输入(64x64和64x64x64)执行肿瘤分割。 将经过不同分辨率和尺寸训练的模型的输出合并以消除误报,并使用聚类分析进行后处理以获得最终输出。
2材料和方法
2.1数据与预处理
Brats 2019数据集包括总共335个多机构受试者(Menze,Jakab等人2014,Bakas,Akbari等人2017,Bakas,Akbari等人2017,Bakas,Akbari等人2017,Bakas,Akbari 等(2017)。 它包括259个HGG主题和76个LGG主题。 BRATS组织者对所有MR图像进行的标准预处理步骤包括对解剖模板进行共配准(Rohlfing,Zahr等人2010),重新采样至各向同性分辨率(1 mm3)和颅骨剥离(Bakas,Reyes等人2018) )。 其他预处理步骤包括N4偏置场校正(Tustison,Cook等,2014),用于消除RF不均匀性并将多参数MR图像标准化为零均值和单位方差。
生存期预测任务的目的是基于多参数术前MR成像特征结合分割的肿瘤面罩来预测患者的总体生存期。 仅基于成像特征(具有年龄和切除状态)的生存预测是一项艰巨的任务(组织病理学成像,基因组信息,基于放射性示踪剂的成像和其他非成像特征等其他信息可用于改善所有生存预测。(mobaderany, Yousefi等人,2018)报告称,通过将基因组信息和组织病理图像结合在一起,形成基因组生存卷积神经网络体系结构(GSCNN模型),可以提高准确性。有几项研究报告使用11C-acetate和18FFDG在PET/CT扫描中预测脑胶质瘤的总生存率(Tsuchida, Takeuchi et al. 2008, Yamamoto, Nishiyama et al. 2008, Kim, Kim et al. 2018)。
2.2网络架构
我们训练了几种模型来分割肿瘤成分。 除分段残差密集网络(Residual Inception dense Network)外,所有用于分割任务的网络架构都是使用Python软件包Segmentation model(Yakubovskiy 2019)导入的。 选择用于脑肿瘤分割的模型具有不同的骨架(DenseNet-169(Huang,Liu等,2017),SERESNEXT-101(Chen,Fan等,2018)和SENet-154(Hu,Shen等。 2018))。 DenseNet架构在医学数据分类和分割任务中显示出了一些有希望的结果(Islam和Zhang,2017年,Chen,Wu等人,2018年,Dolz,Gopinath等人,2018年)。 DenseNet模型在从一个密集块到另一个密集块的特征传播方面具有优势,并克服了梯度消失的问题(Huang,Liu et al.2017)。 挤压和激励块旨在通过增强分类任务的特征之间的相互依赖性来改善特征传播。 这有助于将更多有用的功能传播到下一个块,并减少信息量较少的功能。 该网络体系结构是ILSVC 2017分类挑战中表现最好的。
SENet-154和SE-ResNeXt-101具有更多参数,计算量大,但在ImageNet分类任务中显示出了良好的效果(Hu,Shen et al.2018)。 集合了三个建议的模型以获得最终结果。 所有这些来自“细分模型”包的模型都使用240x240的2D轴向切片尺寸进行了训练(图1)。
残余初始密集网络(RID)最先由Khened等人提出并开发用于心脏分割。我们将自己的RID网络实现与Tensorflow后端合并在了Keras中(图2)。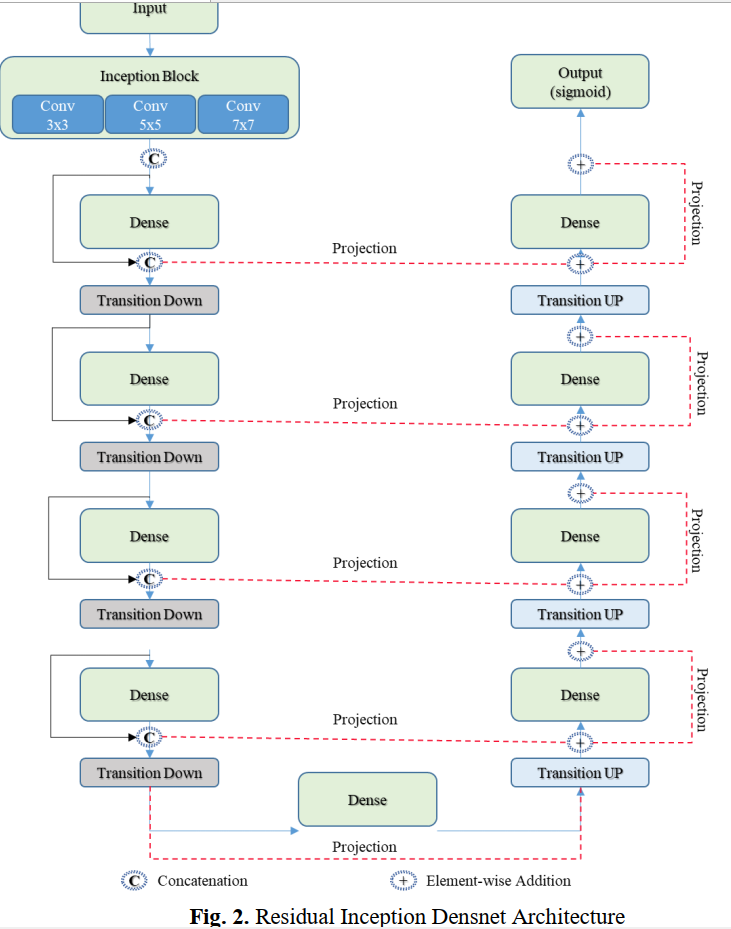
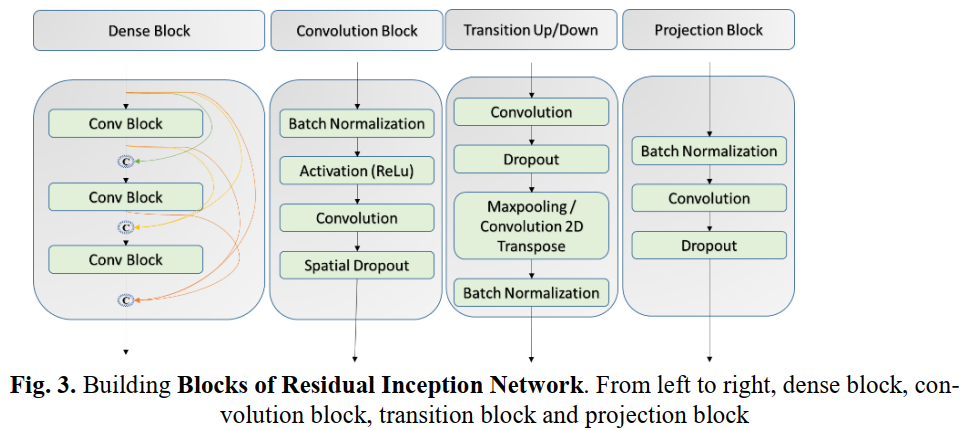
在Densenet架构下,GPU内存占用随着空间分辨率较大的特征图数量的增加而增加。从下采样路径到上采样路径的跳跃连接在该模型中使用了元素级联操作,而不是Densenet中的串联操作,以减轻上采样路径中的特征映射爆炸。对于skip连接,使用BN-1 × 1- convolu- dropout进行投影操作,以匹配元素相加的维度(图3)。这些增加到Densenet架构有助于减少参数和GPU内存占用,而不影响分割输出的质量。除了进行降维,投影操作有助于学习跨通道信息的交互(Lin, Chen et al. 2013)和更快的收敛。此外,RID网络的初始层包括平行的CNN分支,类似于具有多个不同接受域的核的inception模块,这有助于捕捉与视点相关的对象变化和在多个尺度上学习图像结构之间的关系。
2.2.1模型训练和集成方法论。
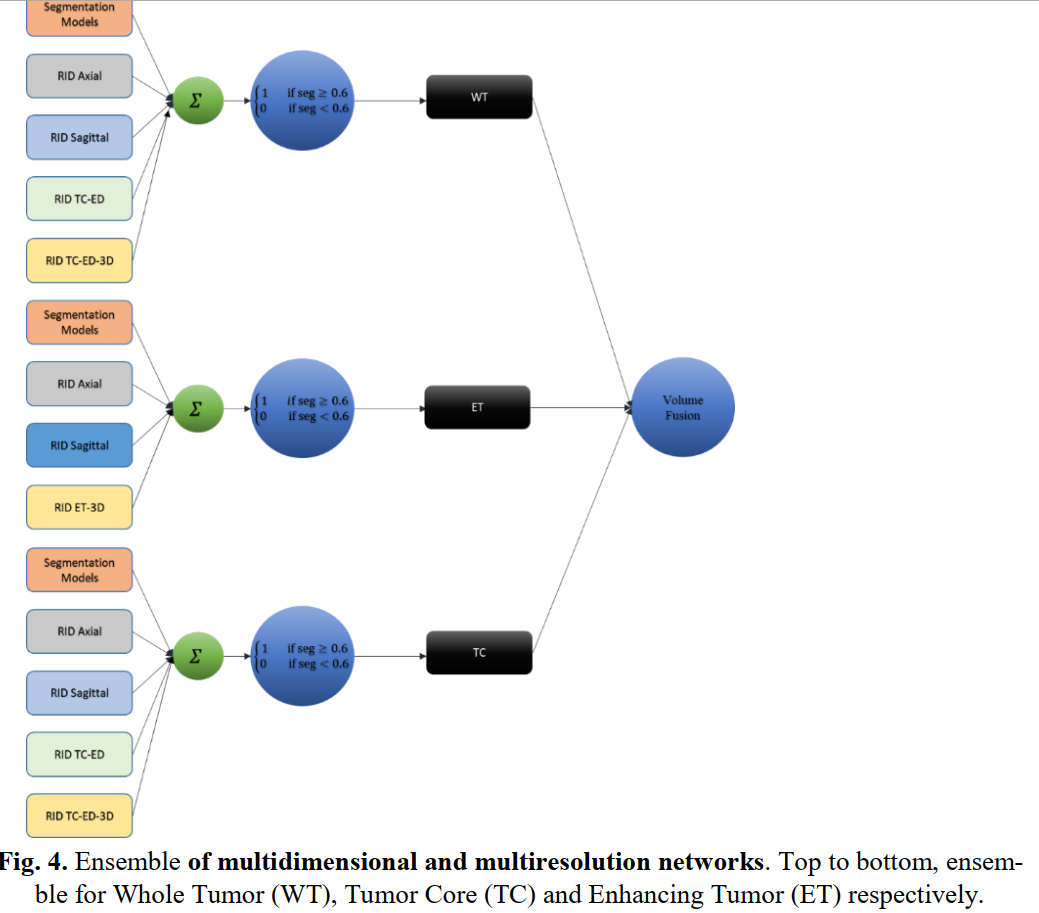
分割模型包中的所有模型均以240x240大小的全分辨率轴向切片作为输入进行训练,分别分割肿瘤子成分。模型中的每个组件的输出被合并,然后进行后期处理。后处理步骤包括去除较小规模的簇以减少误报。然后将每个肿瘤成分合并形成分割图(图1.B)。
在尺寸为64x64的2D输入块上训练RID模型。对于脑肿瘤的每个成分(例如,空穴肿瘤(WT),肿瘤核心(TC)和增强肿瘤(ET)),我们训练了一个单独的RID模型,以轴向和矢状切片作为输入。除了6个RID模型,我们还训练了一个轴向切片的RID,作为输入,贴片大小为64x64,同时分割TC和水肿(TC- ed)。我们还训练了一个三维RID网络模型来分割ET和一个多类TC-ED(TC-ED- 3d)。所有模型都是用骰子损耗训练的,使用NVIDA Tesla P40 GPU的Adam优化器学习率为0.001。
2.2.2整体方法论。
首先将DenseNET-169、SERESNEXT-101和SENet-154模型输出进行组合,形成如图1.B所示的分割图。我们称之为分割模型输出。然后,对于每个组件,我们将RID模型的输出和分割模型的输出组合在一起,如图3所示。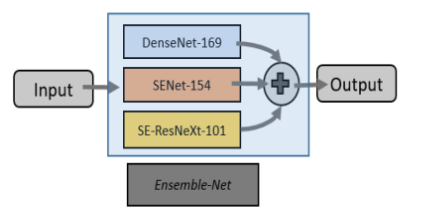

2.2.3生存预测。
从上述方法中提取的肿瘤分割图使用PyRadiomics (Van Griethuysen, Fedorov et al. 2017)和pywavelet (Lee, Gommers et al. 2019)包从每个肿瘤子成分中提取纹理和基于小波的特征。此外,我们还增加了每个肿瘤成分的体积和表面积特征(Feng, Tustison et al. 2018)和年龄。我们使用sklearn软件包(Pedregosa, Varoquaux et al. 2011, Buitinck, Louppe et al. 2013)基于SelectKBest特征进行特征选择,结果缩减了25个特征集。我们训练了XGBoost (XGB)模型(Chen and Guestrin 2016),将受试者分为低存活率(小于300天)、中存活率(300 - 450天)和长存活率(大于450天)。在预测受试者的类别(生存范围)后,将生存日分配给受试者,低生存期150天,中生存期375天,高生存期500天。来自验证数据集的29名受试者对训练后的模型进行验证。
3结果
3.1市场细分
在125个实验对象的验证数据集中,多分辨率2D网络集成的WT、TC和ET的准确率分别达到89.79%、78.43%和77.97%。
3.2生存预测
使用XGB的29名受试者的总体生存预测的准确性和均方误差分别为34.5天和122759.3天。
4讨论
我们集成了几个具有多分辨率输入的模型来分割脑肿瘤。
RID网络的参数和内存都是高效的,并且能够在(最少三个epochs)收敛。这让我们能够在很短的时间内训练几个模型。本文提出的多维模型结合方法提高了分割性能,取得了良好的分割结果,如表1所示。在生存预测方面,我们基于纹理、一阶统计量和小波提取了大量的特征。高效的基于模型的特征选择使我们能够将原本很大的特征集减少到每个主题25个特征。我们训练了几个经典的机器学习模型,然后将它们结合起来改进验证数据集的结果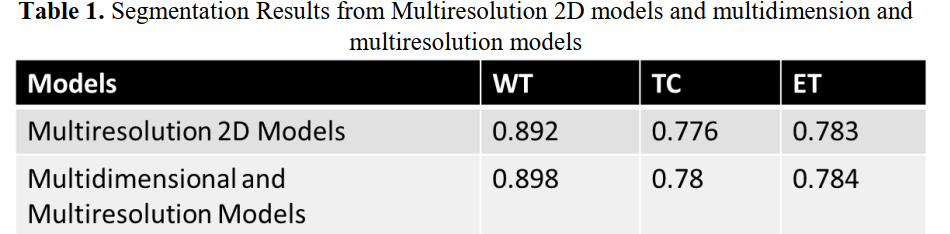
5结论
我们展示了用于脑肿瘤分割的2D多分辨率集成网络,以产生鲁棒的肿瘤子成分分割。我们使用xgboost模型预测了基于分割的mask的总体生存。

