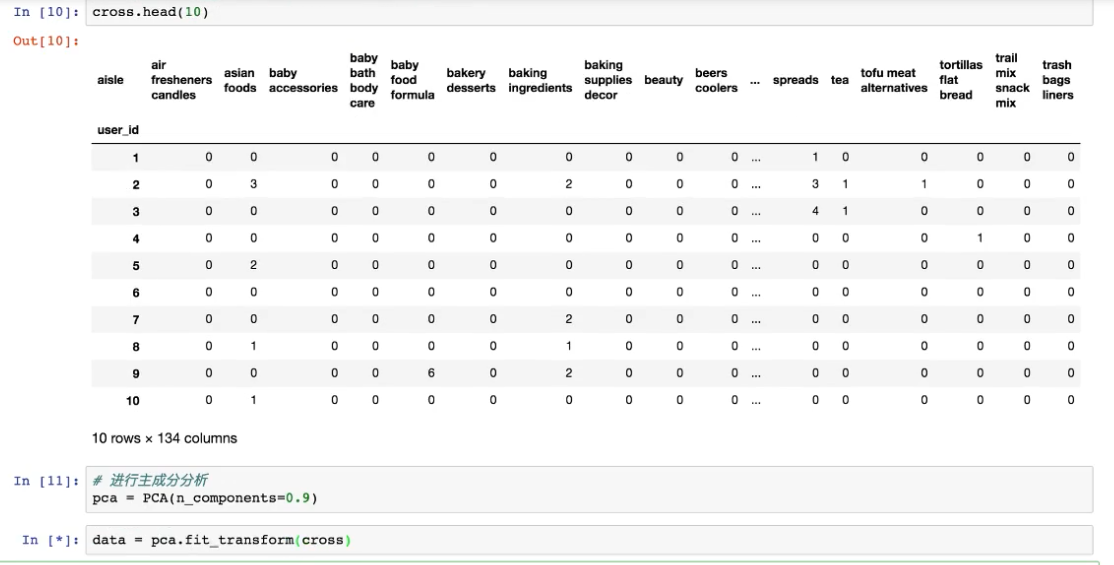
为何需要进行特征选择
对于某一对象的研究往往会有大量乃至成百上千的特征(维度),但是并非所有维度都是与我们的研究息息相关的,比如爱吃冰淇淋的程度和肤色,又或是有些特征几乎所有组分都拥有,或是某一特征 所有的组分都相差不大,计算它们只会大量消耗计算机的性能,为了提高我们的效率以及抓住问题的关键,这时我们就需要过滤一部分特征,即选择一部分有价值的特征进行研究。这一过程我们称为特征选择。
在大量的特征中进行筛选必然是个体力活,这时有一个工具便出现了
直接降维减少特征
data.drop([‘time’],axis=1) # 删除time的一列
工具
api:sklearn.feature_selection.VarianceThreshold
VarianceThreshold(threshold = 0.0)
删除所有低方差特征 ,方差小于等于threshold的特征被删除。
Variance.fit_transform(X,y) X是numpy array格式的数据[n_samples,n_features]
返回值:训练集差异低 threshold的特征将被删除。
默认值是保留所有非零方差特征,即删除所有样本
中具有相同值的特征。
栗子
>>> from sklearn.feature_selection import VarianceThreshold>>> var= VarianceThreshold(threshold = 0.0)>>> a = np.array([[1,2,3],[4,2,6],[3,2,1]])>>> aarray([[1, 2, 3],[4, 2, 6],[3, 2, 1]])>>> var.fit_transform(a)array([[1, 3],[4, 6],[3, 1]])
主成分分析综合特征
高维度数据容易出现某些特征之间通常是线性相关的,因此可以综合密一些特征,PCA的目的 是数据维数压缩,尽可能降低原数据的维数(复杂度),损失少量信息。
工具
导入
from sklearn. decomposition import PCA
使用
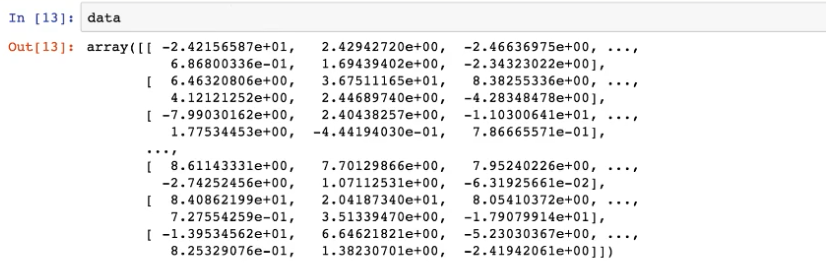
PCA(n_components=None)
将数据分解为较低维数空间 ,n_components一般是0.9~0.95
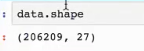
PCA.fit_transform(X),X:numpy array格式的数据[n_samples,n_features]
返回值:转换后指定维度的array